Genetic characteristics of influenza A3 virus circulated in Huzhou, 2009-2012
XU De-shun, JI Lei, CHEN Li-ping, WU Xiao-fang
Huzhou Center for Disease Control and Prevention, Huzhou 313000, Zhejiang, China
Abstract
Objective To understand the genetic variation of hemagglutinin(HA) and neuraminidase(NA) of influenza A3 virus circulated in Huzhou during 2009-2012. Methods The RNA of 11 influenza A3 virus strains, circulated in Huzhou during this period, were extracted. The HA1 and NA regions were amplified and sequenced. All the sequence data were analyzed using MegAlign. Results The HA1 and NA regions of all strains belonged to 987nt and 1362nt, encoding protein of 329 and 454 amino acids respectively. The strains shared amino acid homology of 94.7%-99.9% and 96.2%-100.0% in HA1 and NA regions respectively, the variation in HA1 region was greater than that in NA region. Multi amino acid loci variation had occurred compared with the influenza A3 virus strains isolated in previous years. In addition, there were some differences between the vaccine strains recommended by WHO and the circulated strains. Conclusion Obvious genetic variation had occurred among the strains of influenza A3 virus isolated in Huzhou from 2009 to 2012. Genetic reassortment and antigenic drift seemed to be the major evolutionary mechanism of influenza A3 virus.
2009-2012年浙江省湖州市甲3亚型流行株基因特性分析
徐德顺, 纪蕾, 陈莉萍, 吴晓芳
湖州市疾病预防控制中心,浙江 湖州 313000
摘要
目的 分析2009-2012年湖州市甲3亚型流感病毒主要抗原基因血凝素(HA)和神经氨酸酶(NA)的分子变异特征。 方法 选择浙江省湖州市2009-2012年流感流行期间分离的甲3亚型代表株11株,提取病毒RNA,扩增HA1和NA基因,进行序列测定,用MegAlign软件分析处理。 结果 湖州市近几年甲3亚型流感流行株在HA1区的核苷酸长度均为987 bp,编码329个氨基酸;在NA区的核苷酸长度为1362 bp,编码454个氨基酸。2009-2012年甲3亚型流感病毒分离株HA1区与NA区的氨基酸同源性分别在94.7%~99.9%与96.2%~100.0%之间,HA1区的变异较NA区更大。4个流感流行期分离的甲3亚型流感毒株相比HA和NA发生了多个氨基酸位点的替换。此外,近几年来世界卫生组织推荐的甲3亚型流感疫苗株与当年的流感流行株之间存在一定的差异。 结论 2009-2012年湖州市甲3亚型流感病毒流行株发生明显变异,基因重配和抗原漂移是病毒发生变异的主要机制。
内容大纲
-
1 材料与方法
- 1.1 标本采集和病毒分离
- 1.2 病毒 RNA 的提取及基因 PCR 扩增
- 1.3 序列测定与分析
- 2.1 HA1基因变异和进化树分析
- 2.2 HA1氨基酸变异分析
- 2.3 NA基因变异和进化树分析
- 2.4 NA氨基酸变异分析
1 材料与方法
1.1 标本采集和病毒分离 2009年1月至2012年6月湖州市2家流感监测哨点医院采集的体温>38 ℃伴上呼吸道感染疑似流感病例的咽拭子和/或含漱液标本,4 ℃运送至实验室于-80 ℃保存。病毒分离按《流行性感冒诊断标准 WS 285 - 2008》附录A病毒分离操作规程进行。MDCK细胞、流感标准抗原和抗血清由浙江省疾病预防控制中心(CDC)病毒所提供,本实验室保存。
1.2 病毒 RNA 的提取及基因 PCR 扩增
取病毒培养物100 μl, 使用德国 Qiagen 公司 Rneasy Mini Kit 中的Rneasy Mini Kit Handbook 所提供的方法提取病毒 RNA。HA1和NA引物序列参照文献[2],由宝生物(大连)工程有限公司合成纯化。反转录-聚合酶链反应(RT-PCR)反应体系参照TaKaRa公司One Step RT-PCR Kit试剂盒说明书进行设置,反应条件为42 ℃ 30 min,95 ℃ 10 min反转录后,94 ℃ 60 s,55 ℃ 60 s(NA为2 min),72 ℃ 45 s,共35个循环,然后在72 ℃ 9 min,最后转入 6 ℃ 保存。产物用1.5%的琼脂糖电泳检测。
1.3 序列测定与分析
RT-PCR产物送宝生物(大连)工程有限公司进行核苷酸序列测定。利用MegAlign等软件进行核苷酸和氨基酸序列分析。除本实验测序数据以外的其他基因序列以及历年流感疫苗株基因序列均从美国GenBank流感数据库中下载。
2 结果
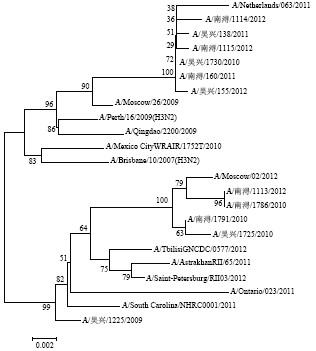
2.1 HA1基因变异和进化树分析 利用MegAlign软件对11株湖州市甲3亚型流感分离株以及WHO推荐的流感疫苗株A/Brisbane10/2007和A/Perth/16/2009 HA1基因编码序列构建系统进化树,见图1。湖州地区分离株大致可分为3组:2009年为独立的一组,第2组为2010年分离株,第3组为2011年和2012年分离株,各分离株间的HA1变异率最高达3.3%。与疫苗株 A/Brisbane10/2007和A/Perth/16/2009相比较:2009年和2010年湖州分离株与其存在较大的差异, 在系统树上距离较远,处于较大的2个分支上,但2011和2012年分离株与其相比,在系统树上距离较近,处在一个大的分支下的2个小分支里。另外图1也显示湖州市2012年度内有2个不同分支的甲3亚型流感毒株在当地流行,其他年份毒株该特征不明显。而且2011和2012年湖州分离株与A/Netherlands/063/2011接近,处于同一独立的分支上。而2009 - 2010年分离株与其他地区甲3亚型流行株HA1区呈现较大的差异,分处于几个距离较远的进化分支上。
图1 2009 - 2012年湖州市甲3亚型的流感病毒分离株HA1进化树
Figure 1 Phylogentic tree of HA1 of influenza A3 virus strains isolated in Huzhou,2009 - 2012 2.2 HA1氨基酸变异分析
对2009 - 2012年间的11株甲3亚型的流感病毒HA1基因区进行了序列测定,发现11株分离株HA1区核苷酸均为987 bp,未发现核苷酸的丢失或插入。由此推导HA1区的氨基酸为329个。利用MegAlign等软件分析发现11株甲3亚型HA1区的氨基酸同源性在94.7%~99.9%之间,2009年与2010年分离株同源性为97.2%~98.1%,2010年与2011年分离株同源性为95.3%~100%,2011年与2012年分离株同源性为94.7%~100%,分离年代越接近的毒株同源性越好。与疫苗株A/Perth/16/2009相比,湖州市甲3亚型流感分离株HA1区的氨基酸存在一定的差异,其中有7个差异位点分别位于抗原决定簇A区的133~137和140~146位氨基酸,B区的156~160位和187~198位氨基酸,C区的278位氨基酸以及E区的63位氨基酸,见表1。
此外,潜在的糖基化位点(Asn-X-Ser/Thr)的分析比较发现所测序的11株毒株在HA1区有9~11个潜在的糖基化位点,除A/南浔/1113/2012(少144位,133位)只有9个潜在的糖基化位点,A/吴兴/1730/2010比以前增加了122位糖基化位点外,其余的分离株都有11个潜在的糖基化位点(8、22、38、63、122、126、133、144、165、246、285),基本保持不变。
表1 2009 - 2012年湖州市甲3亚型流感病毒株HA1氨基酸主要差异位点
Table 1 Loci of amino acid variation of HA1 of influenza A3 virus strains isolated in Huzhou,2009 - 2012
| 毒株 | 33 | 45 | 48 | 49 | 63 | 124 | 133 | 144 | 158 | 162 | 173 | 187 | 198 | 212 | 214 | 223 | 260 | 261 | 268 | 273 | 278 | 306 | 312 |
| A/Perth/16/2009 | Q | S | T | E | K | S | R | K | N | P | Q | L | A | T | S | V | I | R | M | P | N | R | N |
| A/Brisbane/10/2007 | Q | S | T | E | E | S | R | N | K | P | K | H | A | T | I | V | I | R | M | P | N | R | N |
| A/吴兴/1225/2009 | Q | S | T | E | E | S | R | N | N | P | Q | H | A | A | I | V | I | R | M | P | N | R | N |
| A/吴兴/1725/2010 | Q | N | I | E | K | S | R | N | N | P | Q | H | S | A | I | I | I | R | M | P | K | R | S |
| A/吴兴/1730/2010 | Q | S | T | K | E | S | K | K | N | S | Q | H | A | T | S | V | I | M | Q | P | N | R | N |
| A/南浔/1791/2010 | Q | N | I | E | K | S | R | N | N | P | Q | H | S | A | I | I | I | R | M | P | K | R | S |
| A/南浔/1786/2010 | R | N | I | E | K | S | R | N | N | P | Q | H | S | A | I | I | I | R | I | P | K | R | S |
| A/吴兴/138/2011 | Q | S | T | K | E | N | R | K | N | S | Q | H | A | T | I | V | M | Q | M | A | N | R | N |
| A/南浔/160/2011 | Q | S | T | K | E | S | R | K | N | S | Q | H | A | T | I | V | M | Q | M | P | N | R | N |
| A/吴兴/155/2012 | Q | S | T | K | E | S | R | K | N | S | Q | H | A | T | I | V | M | Q | M | P | N | R | N |
| A/南浔/1113/2012 | R | N | I | E | K | N | R | N | N | P | Q | H | S | A | I | I | I | R | I | P | N | R | N |
| A/南浔/1114/2012 | Q | S | T | K | E | S | R | K | N | S | Q | H | A | T | I | V | M | Q | M | P | N | K | N |
| A/南浔/1115/2012 | Q | S | T | K | E | S | R | K | N | S | Q | H | A | T | I | V | M | Q | M | P | N | R | N |
2.3 NA基因变异和进化树分析
用MegAlign软件对11株流感流行株以及近几年WHO推荐的流感疫苗株NA基因构建系统进化树,见图2。NA基因的系统进化树提示:与HA1基因分析一样,湖州 地区分离株NA基因大致也可分为3组:2009年为独立的一组,第2组为2010和2011年分离株,第3组为2012年分离株,各分离株间的NA变异率最高达2.3%,说明湖州市分离到的甲3 亚型流感病毒的NA基因特性也发生了一些变化。从NA基因层面上看,2009 - 2012 年湖州流感分离株之间存在较
图2 2009 - 2012年湖州市甲3亚型的流感病毒分离株NA进化树
Figure 2 Phylogentic tree of NA of influenza A3 virus strains isolated in Huzhou,2009 - 2012 大的差异,而在同一年份流感流行株之间变化不大,与HA1序列分析结果相一致。2009 - 2010年 的甲3亚型疫苗株A/Brisbane/10/2007与2010年湖州市流感流行株存在的差异大,且与2009年甲3亚型流感毒株A/huzhou/1225/2009也有一定的差异,分处于2个较大的分支上。2010年和2011年湖州分离株在系统分支上都远离了当年的流感疫苗推荐株A/Perth/16/2009。
2.4 NA氨基酸变异分析
所测11株甲3亚型流感毒株NA基因区均为1362个碱基,由此推导的氨基酸为454个,也未发现编码氨基酸的缺失与插入。利用软件分析发现11株甲3亚型NA区的氨基酸同源性在96.2%~100%之间,2009年与2010年分离株同源性为97.1%~98.8%,2010年与2011年分离株同源性为97.8%~100%,2011年与2012年分离株同源性为97.8%~99.9%。各年份分离株氨基酸相比,2009年与2010年在NA区域氨基酸差异位点为7个,2010年与2011年差异位点为0个,2011年与2012年差异位点为19~21个,其中5个氨基酸差异位点分别涉及NA的3个抗原决定簇,即A区1个(383~387位氨基酸),B区的3个(197~200位和221位氨基酸)以及C区的339位氨基酸。发生差异的主要氨基酸位点见表2。
甲3亚型NA区的糖基化位点分别位于61、70、86、146、200、234、329和402位点上(2个在茎区,6个在球部)[3],从测序比对结果分析,湖州市分离株这8个潜在的糖基化位点基本保持不变,但2011年的A/吴兴/138/2011在93位上新增加了一个潜在的糖基化位点,2012年的分离株A/南浔/1114/2012该位点又消失了,但大体上糖基化位点是保守的。
表2 2009 - 2012年湖州市甲3亚型流感病毒株NA氨基酸主要差异位点
Table 2 Loci of amino acid variation of NA of influenza A3 virus strains isolated in Huzhou,2009 - 2012
| 毒株 | 78 | 85 | 89 | 125 | 132 | 147 | 197 | 199 | 211 | 221 | 246 | 262 | 265 | 286 | 288 | 296 | 330 | 332 | 337 | 339 | 357 | 386 | 416 | 419 | 425 | 439 |
| A/Perth/16/2009 | A | V | F | G | T | P | C | H | P | E | N | A | G | I | A | K | Q | G | G | D | E | S | S | E | S | V |
| A/Brisbane/10/2007 | A | I | F | G | A | P | C | H | P | E | N | A | G | L | A | K | Q | R | S | D | E | S | S | E | S | V |
| A/吴兴/1225/2009 | A | I | F | R | A | P | C | R | P | E | R | T | G | L | A | E | R | R | S | D | E | S | S | V | Y | G |
| A/吴兴/1725/2010 | T | I | L | G | A | S | G | H | S | K | N | A | R | L | S | K | Q | R | S | N | D | N | L | E | S | V |
| A/吴兴/1730/2010 | A | I | F | R | A | P | C | R | P | E | R | T | G | L | A | E | R | R | S | D | E | S | S | V | Y | G |
| A/南浔/1791/2010 | T | I | L | G | A | S | G | H | S | K | N | A | R | S | A | K | Q | R | S | N | D | N | L | E | S | V |
| A/南浔/1786/2010 | T | I | L | G | A | S | G | H | S | K | N | A | R | L | S | K | Q | R | S | N | D | N | L | E | S | V |
| A/吴兴/138/2011 | T | I | L | G | A | S | G | H | S | K | N | A | R | L | S | K | Q | R | S | N | D | N | L | E | S | V |
| A/南浔/160/2011 | A | I | F | G | A | S | C | H | P | E | N | A | G | L | A | K | Q | R | S | D | E | S | S | V | Y | G |
| A/吴兴/155/2012 | A | I | F | R | A | P | C | R | P | E | R | T | G | L | A | E | R | R | S | D | E | S | S | V | Y | G |
| A/南浔/1113/2012 | A | I | F | R | A | P | C | R | P | E | R | T | G | L | A | E | R | R | S | D | E | S | S | V | Y | G |
| A/南浔/1114/2012 | A | I | F | R | A | P | C | R | P | E | R | T | G | L | A | E | R | R | S | D | E | S | S | V | Y | G |
| A/南浔/1115/2012 | A | I | F | R | A | P | C | R | P | E | R | T | G | L | A | E | R | R | S | D | E | S | S | V | Y | G |
3 讨论
流感病毒的抗原变异与流感的发生和流行密切相关, 流感病毒正是通过不断改变其抗原性来逃避宿主特异性免疫的识别和清除, 从而不断引起流行。目前, 已知甲 3型流感病毒的 HA1上至少有 5个抗原决定簇, 为A~E, 且 A和 B区更为重要[4],NA蛋白分子上也已知有A、B、C三个抗原决定簇[5], 一般认为 HA1区氨基酸替换发生 4个以上并分布在 2个以上的抗原决定簇则该毒株的变异具有流行病学意义[6]。本文资料显示:湖州市分离株与2009 - 2010年甲 3流感疫苗株 A/Brisbane/10/2007疫苗株比较,2009年毒株氨基酸差异数为6, 且所在抗原决定簇为2个;2010年分离株氨基酸差异数不仅>6,且所在抗原决定簇>3,而与2010 - 2012年疫苗株A/Perth/16/2009比较发现, 3年湖州分离株氨基酸差异数均>5。在NA区,A/吴兴/1225/2009与当年疫苗株A/Brisbane/10/2007之间仅4个氨基酸差异,表明NA 区的差异要小于HA1区。2010年和2011年湖州市的甲3亚型流感病毒株与当年的疫苗株A/Perth/16/2009之间均有14个氨基酸差异,涉及3个抗原决定簇,且氨基酸同源性为88.9%~99.9%。从氨基酸进化树分析:2009 - 2010年的甲3亚型疫苗株A/Brisbane/10/2007与2010年湖州市流感分离株在进化树上相距较远,且与2009年甲3亚型流感分离株A/吴兴/1225/2009也分处于2个较大的分支上,2010 - 2012年疫苗推荐株A/Perth/16/2009与2010 - 2012年分离株之间,在系统分支上都远离了当年的流感流行毒株,2009 - 2012年WHO推出的疫苗株与湖州市的甲3亚型流行株之间有一定的滞后现象,提示甲3亚型疫苗对湖州市人群的保护效果会有一定的影响。
有研究表明,糖基化在流感病毒等微生物感染宿主过程中发挥重要作用,具有防止流感病毒蛋白被水解,稳定蛋白的结构,阻碍抗体对病毒颗粒的识别和清除等功能[7]。糖基化位点的增加或减少对病毒的抗原性及其他生物特性均有一定的影响。糖基化位点的改变如果发生在抗原决定簇, 就会妨碍特异抗体对病毒粒的识别和清除,而造成抗原性漂移。将湖州市11株分离株与A/Perth/16/2009和A/Brisbane/10/2007疫苗株的受体结合位点、二硫键位点进行比较, 发现均未发生位点替换,对影响抗原特性的糖基化位点替换情况进行分析发现, 湖州市绝大多数分离株其 HA1编码区均含有 11个糖基化位点, 此特性与2010 - 2012年甲 3 流感疫苗株 A/Perth/16/2009疫苗株相同。但A/南浔/1113/2012分离株在 144位和133位发生氨基酸替换, 导致丢失2个潜在糖基化位点,只有9个糖基化位点,A/吴兴/1730/2010在122位发生氨基酸替换,增加了1个糖基化位点;在NA区,除了个别分离株增加或消失了93位上的糖基化位点外,均为8个糖基化位点,变异不大,相对比较保守,大部分结构功能区的位点都保持不变。
湖州市季节性流感甲型流行株除了2009年为新甲型H1N1亚型外,均以甲3亚型流感病毒为流行的优势亚型。而甲型流感病毒最显著的特点是其基因易发生变异,变异可使其逃避宿主免疫系统的识别与清除,从而能多次反复有效的在人群流行[8]。因此,密切监测HA1与NA基因,特别是HA1基因的变异情况,对流感预测、预防与疫苗筛选都具有十分重要的意义。
参考文献
[1] Liu J,Jiang T,Xu LJ,et al. Development and evaluation of a novel realtime reversetranscriptase PCR assay for detection of R292K mutation of the H3N2 influenza A virus[J]. Military Medical Sciences, 2011,35(6):432-436.(in Chinese) 刘娟,姜涛,徐莉娟,等. 实时RTPCR检测甲型H3N2流感病毒R292K耐药位点[J]. 军事医学,2011,35(6):432-436.
[2] Lu YY, Yan JY, Mao HY, et al. Analysis on genetic characteristics of HA1 and NA regions of influenza A3 virus strains in Zhejiang,1998-2005 [J]. Chinese Journal of Epidemiology, 2007,29(11):1092-1095.(in Chinese) 卢亦愚,严菊英,茅海燕,等. 浙江省1998-2005年甲3亚型流感病毒株HA1区和NA区基因特性分析[J]. 中华流行病学杂志,2007,28(11):1092-1095.
[3 Guo YJ, Qi JL, Dong J, et al. The origin of a novel kind of reassortant(H1N2) of influenza A virus [J]. Science in China(Series C: Life Sciences),2000,30(5):499-504.(in Chinese) 郭元吉,祁俊林,董婕,等. 一种新的甲型流感病毒重配株(H1N2)来源的研究[J]. 中国科学C 辑,2000,30(5): 499-504.
[4] Bush RM, Bender CA, Subbarao K, et al. Pred icting the evolution of human in fluenza A [J]. Science, 1999, 286: 1921-1925.
[5] Enrique TM, Michael WD. Epitope analysis for influenza vaccine design[J]. Vaccine, 2005,23:1144-1148.
[6] Guntler L. A H1 hemagglutin in ofahum an influenza Avirus with a carbohydrate modulated recep to rbinding site and all unusual cleavages site[J]. Virus Res, 1993,2:147-160.
[7] Zhu RN, Qian Y, Wang F, et al. Gene sequencing of influenza A3 virus isolates in infants and young children in Beijing, 1998-2004 [J]. Chinese Journal of Epidemiology, 2006,27(3): 241-244. (in Chinese) 朱汝南,钱渊,王芳,等. 北京市1998-2004年婴幼儿A3型流感病毒分离株基因序列分析[J]. 中华流行病学杂志,2006,27(3):241-244.
[8] Kang N, Tan Y, Bi FY, et al. Research on genetic variation characteristics in HA1 of influenza A(H3N2) viruses in Guangxi,2007-2009 [J]. Disease Surveillance, 2011,26(4):278-283.(in Chinese) 康宁,谭毅,闭福银,等. 2007-2009年广西壮族自治区甲3亚型流感病毒HA1基因变异的特征研究[J]. 疾病监测,2011,26(4):278-283.
[2] Lu YY, Yan JY, Mao HY, et al. Analysis on genetic characteristics of HA1 and NA regions of influenza A3 virus strains in Zhejiang,1998-2005 [J]. Chinese Journal of Epidemiology, 2007,29(11):1092-1095.(in Chinese) 卢亦愚,严菊英,茅海燕,等. 浙江省1998-2005年甲3亚型流感病毒株HA1区和NA区基因特性分析[J]. 中华流行病学杂志,2007,28(11):1092-1095.
[3 Guo YJ, Qi JL, Dong J, et al. The origin of a novel kind of reassortant(H1N2) of influenza A virus [J]. Science in China(Series C: Life Sciences),2000,30(5):499-504.(in Chinese) 郭元吉,祁俊林,董婕,等. 一种新的甲型流感病毒重配株(H1N2)来源的研究[J]. 中国科学C 辑,2000,30(5): 499-504.
[4] Bush RM, Bender CA, Subbarao K, et al. Pred icting the evolution of human in fluenza A [J]. Science, 1999, 286: 1921-1925.
[5] Enrique TM, Michael WD. Epitope analysis for influenza vaccine design[J]. Vaccine, 2005,23:1144-1148.
[6] Guntler L. A H1 hemagglutin in ofahum an influenza Avirus with a carbohydrate modulated recep to rbinding site and all unusual cleavages site[J]. Virus Res, 1993,2:147-160.
[7] Zhu RN, Qian Y, Wang F, et al. Gene sequencing of influenza A3 virus isolates in infants and young children in Beijing, 1998-2004 [J]. Chinese Journal of Epidemiology, 2006,27(3): 241-244. (in Chinese) 朱汝南,钱渊,王芳,等. 北京市1998-2004年婴幼儿A3型流感病毒分离株基因序列分析[J]. 中华流行病学杂志,2006,27(3):241-244.
[8] Kang N, Tan Y, Bi FY, et al. Research on genetic variation characteristics in HA1 of influenza A(H3N2) viruses in Guangxi,2007-2009 [J]. Disease Surveillance, 2011,26(4):278-283.(in Chinese) 康宁,谭毅,闭福银,等. 2007-2009年广西壮族自治区甲3亚型流感病毒HA1基因变异的特征研究[J]. 疾病监测,2011,26(4):278-283.
|
扩展功能
|
|
| 本文信息 | |
| PDF全文 | |
| HTML全文 | |
| 参考文献 | |
| 服务与反馈 | |
| 加入引用管理器 | |
| 引用本文 | |
| Email Alert | |
| 本文作者相关文章 | |
| 徐德顺 | |
| 纪蕾 | |
| 陈莉萍 | |
| 吴晓芳 | |
| PubMed | |
| Article by XU De-shun | |
| Article by JI Lei | |
| Article by CHEN Li-ping | |
| Article by WU Xiao-fang | |