Study on evolution of pO157 in E. coli O157:H7 strains isolated in China
XU Xue-fang, WU Jian-hong, LIU Xue-tong, XIONG Yan-wen, XU Jian-guo
State Key Laboratory for Communicable Diseases Prevention and Control, Institute for Communicable Disease Control and Prevention, Chinese Center for Disease Control and Prevention, Beijing 102206, China
Abstract
Objective To investigate the evolution of pO157 of O157:H7 strains isolated in China. Methods The classification of the strains was conducted by using restriction enzyme digestion, the amplification by using overlapping primer PCR amplification and the confirmation by using Southern blot. Finally the sequencing was done. Results Sixty strains of E. coli O157:H7 were classified as 5 groups(A-E) by enzyme digestion. However A group and C group were actually belonged to one group because the enzyme digestion difference was caused by a small plasmid in C group using HindⅢ digestion. PCR products from primer pairs 5 and 25 differed between A, C groups and pO157. Only PCR fragments from primer pair 25 showed difference between B group and pO157. The reason for the PCR fragment difference from pair 25 between A-C groups and pO157 was the introduction of IS1310. Conclusion pO157 in the isolated O157:H7 strains in China is relatively conserved. The evolution of pO157 in the O157:H7 strains isolated in China was mainly caused by some inserted sequences such as IS1310.
我国大肠埃希菌O157:H7分离株大质粒pO157变异性研究
徐雪芳, 伍建宏, 刘学通, 熊衍文, 徐建国
中国疾病预防控制中心传染病预防控制所, 传染病预防控制国家重点实验室, 北京 102206
摘要
目的 分析我国分离的大肠埃希菌O157:H7菌株中大质粒pO157的变异情况。 方法 采用限制性内切酶酶切法分析质粒酶切图谱,而后采用嵌套引物进行聚合酶链反应(polymerase chain reaction,PCR)扩增,并用Southern blot进行验证,最后测序。 结果 根据酶切图谱将60株大肠埃希菌O157:H7分为A~E 5个群,而进一步研究表明造成A群与C群HindⅢ 酶切图谱的不同是由C群中的一个小质粒造成的。PCR结果显示A群与C群中的引物对5和25,B群中的引物对25得到的PCR条带与pO157出发菌株不同,其他均相同。测序结果显示造成引物对25在A~C群中与致病性大质粒pO157不同的原因是IS1310序列的插入。 结论 我国大肠埃希菌O157:H7分离株致病性大质粒pO157相对保守,部分菌株中存在插入序列如IS1310,这也是造成我国大肠埃希菌O157:H7 pO157质粒变异的主要原因。
内容大纲
-
1 材料与方法
- 1.1 试验用菌株与质粒
- 1.2 聚合酶链反应(polymerase chain reaction,PCR)扩增
- 1.3 DNA杂交
- 2.1 大肠埃希菌O157 ∶ H7致病性大质粒酶切分析
- 2.2 大肠埃希菌O157 ∶ H7致病性大质粒变异区的查找
- 2.3 1128、998和1005菌株致病性大质粒酶切位点分布
- 2.4 变异区DNA的克隆及测序分析
Schmidt等[1]报道,在所有的被检测的大肠埃希菌O157 ∶ H7菌株中,均含有一个分子质量大小为 6×107的大质粒。该质粒通常称为致病性大质粒pO157。在肠出血性大肠埃希菌O26 ∶ H11菌株和大多数从腹泻患者中分离到的产志贺毒素大肠埃希菌菌株中也含有该质粒。流行病学资料表明该质粒与HUS的形成有很明显的相关性[2]。相继报道的该质粒上的一些可能与致病有关的基因,包括双功能基因-过氧化氢/过氧化物酶基因KatP[3]、胞外丝氨酸蛋白酶基因espP[4]、溶血素基因hlyA[5],以及与生物膜形成相关基因等[6]。本研究对60株大肠埃希菌O157 ∶ H7分离株大质粒进行酶切分析时发现该质粒的HindⅢ酶切图谱十分相似,它们之间只有少量条带的差异。对造成这些变异的位点进行研究,可能为认识该质粒,甚至O157 ∶ H7大肠埃希菌的起源和进化提供线索。
1 材料与方法
1.1 试验用菌株与质粒 用于质粒限制性酶切图谱分析的60株大肠埃希菌O157 ∶ H7均为本室保存菌株,部分菌株的特征和质粒见表1。
表1 实验所用部分菌株和质粒
Table 1 Partial strains and plasmid used in this study
| 菌株/质粒 | 特征 | 来源 |
| 菌株 | ||
| JM109 | 本室保存 | |
| pO157c | 含有经过人工基因工程改造的E. coli O157 ∶ H7 EDL933致病性大质粒(pO157)的C600,在pO157的5413 bp处插入了Tn801[14],插入位置位于引物对45的扩增区间。 | 本室保存 |
| E. coli 998 | O157 ∶ H7,stx | 本室保存 |
| E. coli 1128 | O157 ∶ H7,stx | 本室保存 |
| E. coli 1003 | O157 ∶ H7,stx | 本室保存 |
| E. coli 1005 | O157 ∶ H7,stx | 本室保存 |
| E. coli 1006 | O157 ∶ H7,溶血素基因缺失,stx | 本室保存 |
| 质粒 | ||
| pUC19 | Apr | 本室保存 |
1.2 聚合酶链反应(polymerase chain reaction,PCR)扩增
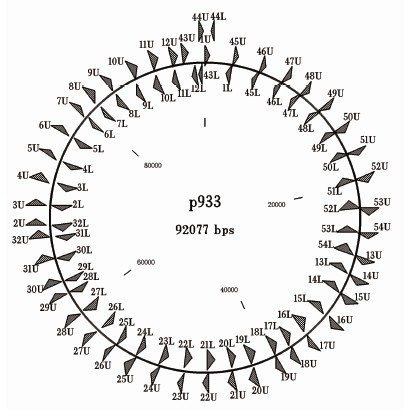
根据已发表的大肠埃希菌O157 ∶ H7 EDL933菌株的致病性大质粒pO157的全序列(GenBank Number:AF074613),采用Primer-Walking的方法,应用Oligo软件设计44对引物(图1),用这些引物(表2)[7]分别对大肠埃希菌O157 ∶ H7 1128、998、1005和1006菌株的致病性大质粒进行PCR扩增, 所用聚合酶为Pfu(Fermentas)。扩增条件如下:94 ℃预变性 4 min→94 ℃变性30 s,相应的退火温度下退火45 s,72 ℃延伸 2.5 min,25个循环→72 ℃延伸10 min。
1.3 DNA杂交
探针标记DNA标记采用DIG DNA
图1 嵌套引物在质粒上的分布
Figure 1 Location of overlapping primers on large plasmid
Labeling and Detection Kit(Boehringer Mannhein,Germany) 对DNA进行标记,操作按说明书进行。
1.3.1 转膜及膜处理
将电泳完的胶切去多余部分,切去左上角;在变性液中变性45 min,不断地摇动;中和液中和15 min,2次;将硝酸纤维素膜在去离子水中浸透,再用20×SSC浸泡至少5 min。同时用2×SSC浸泡两张与膜同样大小的滤纸;将中和好胶翻转(加样孔朝下)放于用滤纸包好的支撑物上,用塑料膜围绕胶一周,以防短路;将膜放于胶上,除去气泡,将滤纸放在膜上方,再将略小于滤纸的吸水纸压于其上;转膜8~24 h,取出膜,用6×SSC浸泡5 min,以除去粘在膜上的琼脂糖碎片;将膜置于干净的滤纸上晾干30 min以上;80 ℃固定2 h。
1.3.2 预杂交和显色检测
将固定后的硝酸纤维素膜装于塑料袋中,加入预杂交液(20 ml/100 cm2),封口,68 ℃水浴2 h;将探针加入少量的预杂交液中,沸水中煮10 min后立即置于冰上,加入一定量的预杂交液,使探针终浓度为5~25 ng/ml,将杂交液加入袋中(5 ml/100 cm2),68 ℃水浴16 h;2×SSC 0.1%SDS室温下洗5 min,2次;0.1×SSC 0.1%SDS 68 ℃洗15 min,2次;Buffer Ⅰ室温下洗1 min;Buffer Ⅱ室温下洗30 min;用Buffer Ⅱ按1 ∶ 10 000的比例稀释抗体(Boehringer Mannheim,Germany),膜在抗体溶液中室温下温育30 min;Buffer Ⅰ室温下洗15 min,2次;Buffer Ⅲ室温下平衡2~5 min;37 ℃显色10 min。
表2 基于pO157序列设计的引物
Table 2 Primers based on plasmid pO157 complete sequence
| 引物 | 退火温度 (℃) | 扩增片段 长度(bp) | 引物 | 退火温度 (℃) | 扩增片段 长度(bp) | |
| 1U:5′-GAA AAC GGA AGG AGA GAC GC-3′ | ||||||
| 1L:5′-CAC TCA AGA ATG TAA GCC C-3′ | 60 | 2722 | ||||
| 2U:5′-CAT CAC GGA CTA CAA AGC AC-3′ | ||||||
| 2L:5′-ATT ACG GAC AAC CAG GCG AA-3′ | 60 | 2376 | ||||
| 3U:5′-GGA GAG ATG AGG ATG AGC GA-3′ | ||||||
| 3L:5′-TGG CAT CCG TCT GTG GGC GT-3′ | 60 | 2235 | ||||
| 4U:5′-GAG AAG ATG AAG AAA GGC GA-3′ | ||||||
| 4L:5′-TAA TCC AGC CGT TGC CAG AG-3′ | 58 | 2385 | ||||
| 5U:5′-CAT TTT GCC GAA CGC TTA CC-3′ | ||||||
| 5L:5′-AAC CTT ACC TTT CTG CTT AC-3′ | 58 | 2514 | ||||
| 6U:5′-GCA TAA GTA AGC AGA AAG GT-3′ | ||||||
| 6L:5′-TCA TTC ATT TCT TCC GCT AA-3′ | 56 | 2453 | ||||
| 7U:5′-CCT ACC TGC TCT GGA TTG AT-3′ | ||||||
| 7L:5′-TAT GCT TTT ATT ATC CCA CC-3′ | 56 | 1565 | ||||
| 8U:5′-AAA ACT ACC TAC CTC CCT AT-3′ | ||||||
| 8L:5′-CAG AAG AGA TAA ACC AAG GC-3′ | 56 | 1945 | ||||
| 9U:5′-GTG CCT TGG TTT ATC TCT TC-3′ | ||||||
| 9L:5′-TAA TCA CCA ATA CCA CCA AC-3′ | 56 | 2397 | ||||
| 10U:5′-ATG GTT GGT GGT ATT GGT GA-3′ | ||||||
| 10L:5′-AAA AAT AAC CAC AAC CCC AC-3′ | 56 | 2142 | ||||
| 11U:5′-TTA TTT TTC ACA ACC CGA GT-3′ | ||||||
| 11L:5′-GTT GCT CTG CCC GCT CCT GG-3′ | 58 | 2394 | ||||
| 12U:5′-TGA TTC GCC CCG CCC AGG AG-3′ | ||||||
| 12L:5′-GCC CTC GTT CTC CAT CAC TC-3′ | 61 | 2168 | ||||
| 13U:5′-TTG CGT GAT ACC AGA CAG GC-3′ | ||||||
| 13L:5′-GGC ACT TTA CTC TCA CTC CC-3′ | 60 | 1793 | ||||
| 14U:5′-GGG GGA GTG AGA GTA AAG TG-3′ | ||||||
| 14L:5′-GTG AGT GCT TGT GAT GGA CC-3′ | 58 | 1983 | ||||
| 15U:5′-ACA GGT CCA TCA CAA GCA CT-3′ | ||||||
| 15L:5′-AAA AAC AGC CAG TGC CAA CA-3′ | 58 | 2527 | ||||
| 16U:5′-CTA CTG GAA ATG CTG TTG GC-3′ | ||||||
| 16L:5′-CGC CTG AAA AGA AGC CGC CT-3′ | 60 | 2635 | ||||
| 17U:5′-ACC TCA ACG ATA CGC TTA CA-3′ | ||||||
| 17L:5′-TTA TCC ACG ACC CCT TGC CA-3′ | 60 | 2090 | ||||
| 18U:5′-AGT CAC CCT CCA TCC ATA AG-3′ | ||||||
| 18L:5′-AGT TAC CGA AAT GAC GAG TT-3′ | 58 | 1913 | ||||
| 19U:5′-GAG GTT TGC TTT TCT GTG TA-3′ | ||||||
| 19L:5′-TGT TGA AAG GTC TAT GGT CC-3′ | 56 | 2465 | ||||
| 20U:5′-TCA AAA AGG GAC CAT AGA CC-3′ | ||||||
| 20L:5′-ACA GAG GGG ATA AAC CAA GA-3′ | 55 | 1948 | ||||
| 21U:5′-AGC AAC AGT AGT CGG AGA GT-3′ | ||||||
| 21L:5′-ATT GAG TTT TCT TCC TTC CT-3′ | 56 | 2458 | ||||
| 22U:5′-CAT CAG GTA TCA CAG GAA GG-3′ | ||||||
| 22L:5′-TTT CCA TCT GCC TGA CAT AG-3′ | 58 | 2241 | ||||
| 23U:5′-AAG ATG AAG TCC GCC TGG TT-3′ | ||||||
| 23L:5′-TAA AAA CCC GCC AGA TTG AT-3′ | 56 | 2491 | ||||
| 24U:5′-GGC TTC CCT TGC TTC TTC TA-3′ | ||||||
| 24L:5′-GTC CGT TGT TTC AGG GTC GC-3′ | 60 | 2536 | ||||
| 25U:5′-GCT GCC TGT TGC CAT TGA TT-3′ | ||||||
| 25L:5′-ATG ATG ACG AAC TAA CCC CC-3′ | 58 | 2561 | ||||
| 26U:5′-TCA ACA GCA GAA CTC CAA TG-3′ | ||||||
| 26L:5′-AGA GCA ACA ACT GAT TTA GG-3′ | 56 | 2489 | ||||
| 27U:5′-ATC TCA TCC CTT CTT TTC TA-3′ | ||||||
| 27L:5′-ATC CCC CTC AAG CAC CCA TT-3′ | 58 | 2568 | ||||
| 28U:5′-TGA ATG GGT GCT TGA GGG GG-3′ | ||||||
| 28L:5′-CGC AGT AAA AAG AGG AGC CC-3′ | 61 | 2239 | ||||
| 29U:5′-CGA CAT CTG GAG TAA CGA GG-3′ | ||||||
| 29L:5′-TCC ATT CCG TCA GCG AGG GC-3′ | 61 | 1936 | ||||
| 30U:5′-GGA ACT GGA TGC GTG CTT TA-3′ | ||||||
| 30L:5′-CGC TTT TCT TCC ATC AAC TC-3′ | 58 | 2423 | ||||
| 31U:5′-AGT GAA TGC GTT TTT GCC GT-3′ | ||||||
| 31L:5′-CGC CCC CGT TCT TCCC AGT A-3′ | 58 | 2337 | ||||
| 32U:5′-GAA TCC GCAC AGG CAC TTG A-3′ | ||||||
| 32L:5′-CGG GGC AGG AGT CAC AAA AA-3′ | 61 | 1195 | ||||
| 43U:5′-GAG AAC AAA CCT GAC CTG CC-3′ | ||||||
| 43L:5′-CGG TGT TTT TCT TCT TCT CT-3′ | 56 | 1572 | ||||
| 44U:5′-TGA ATA AAA TGG CAG AGA AG-3′ | ||||||
| 44L:5′-TTC GTT ACC CCC TTC CTC GG-3′ | 56 | 1410 | ||||
| 45U:5′-TGG GCT TAC ATT CTT GAG T-3′ | ||||||
| 45L:5′-AAT CTA CCT GTT TTC TCG T-3′ | 54 | 2632 | ||||
| 46U:5′-CAC ACG AGA AAA CAG GTA G-3′ | ||||||
| 46L:5′-TGC CAA GCC ATA CGA ATA A-3′ | 54 | 2330 | ||||
| 47U:5′-TTA TTC GTA TGG CTT GGC A-3′ | ||||||
| 47L:5′-CTC ATC TAC AGC ATC ATC C-3′ | 54 | 2157 | ||||
| 48U:5′-GAA TGG ATG ATG CTG TAG A-3′ | ||||||
| 48L:5′-CAG ACA GTT CCA GCG ACA A-3′ | 56 | 2297 | ||||
| 49U:5′-AGA AAA AGT GGG TTG TCG C-3′ | ||||||
| 49L:5′-CAA ATC CAG TCC GTC CAG C-3′ | 56 | 2414 | ||||
| 50U:5′-GCA AAC AAG GAA GCA ACC C-3′ | ||||||
| 50L:5′-AAG TTT CAG CCA GCG ACG A-3′ | 56 | 2553 | ||||
| 51U:5′-TGT CAA CTC AGC CCC TCG G-3′ | ||||||
| 51L:5′-CCC AGA GAA TGA AAC CGA C-3′ | 58 | 2850 | ||||
| 52U:5′-GTC GGT TTC ATT CTC TGG G-3′ | ||||||
| 52L:5′-TCA GGA TGT GGA CGA ACG A-3′ | 56 | 2511 | ||||
| 53U:5′-GGT ATT GCC TCC TTT GTC C-3′ | ||||||
| 53L:5′-TCA CCA TCC CAT CCC CAA G-3′ | 56 | 2699 | ||||
| 54U:5′-TGT AGA TGG TTT CAA GGG T-3′ | ||||||
| 54L:5′-GCC TGT CTG GTA TCA CGC AA-3′ | 56 | 2186 |
2 结果
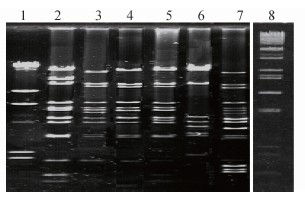
2.1 大肠埃希菌O157 ∶ H7致病性大质粒酶切分析 对60株不同来源的大肠埃希菌O157 ∶ H7的致病性大质粒用HindⅢ进行酶切分析,结果表明这些菌株的质粒酶切图谱十分相似,只存在部分条带的差异,见图2。根据这些条带的差异,将60株O157 ∶ H7大肠埃希菌分为A、B、C、D和E 5个群,其中A群42株,B群11株,C群4株,D群2株,E群1株。1006为溶血素基因缺失株,其HindⅢ酶切图谱相对以上5群的质粒变化较大(图2)。
2.2 大肠埃希菌O157 ∶ H7致病性大质粒变异区的查找
为了找出致病性大质粒的变异区域以及造 成这些变异的因素,我们根据已经发表的 O157 ∶ H7 大肠
图2 致病性大质粒HindⅢ酶切图谱
Figure 2 Restriction patterns of large plasmids digested by HindⅢ 注:1.λDNA/HindⅢ
marker; 2. pO157c; 3. 1128(A群); 4. 1005(B群); 5. 1003(D群); 6. 1006; 7. 998(C群); 8. 1191(E群)。
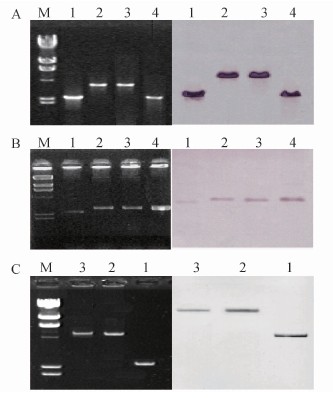
埃 希菌EDL933菌株致病性大质粒的全序列设计44对叠套引物(overlapping primer)(表2),分别以pO157c、 1128(A群)、1005(B群)、998(C群)和1006的致病性大质粒作为模板,用这些引物对其进行PCR扩增,结果表明:1128(A群)和998(C群)的PCR扩增结果一致,引物对5、25扩出的片段比pO157的明显变大(图3A,3B),而引物对27和28的PCR扩增结果均为阴性,其余的引物的扩增片段大小与pO157的一致。1005(B群)只有引物对25扩出的片段比pO157的大(图3B),其余43对扩出的片段大小均与pO157的一致。
图3 致病性大质粒变异区的PCR扩增结果及其杂交结果
Figure 3 PCR products indicating variation regions and Southern blot 注:A. 引物对5扩增片段及其杂交结果。探针为用该引物对pO157c扩增得到的片段; B. 引物对25扩增片段及其杂交结果。探针为用该引物对pO157c扩增得到的片段; C. 引物对 27U/28L扩增片段及其杂交结果。探针为用该引物对pO157c扩增得到的片段;M.λDNA/HindⅢ Marker; 1. pO157c; 2. 1128; 3. 998; 4. 1005。
对于1128、998和1005 的大质粒PCR扩增出来的、与pO157的大小一致的片段,其核苷酸序列是否具同源性?作者对pO157c 43对引物(其中引物对45扩增的区域之间插入了Tn801,因此未用该引物对对pO157c进行PCR扩增和片段的标记)的扩增片段进行地高辛标记,获得探针,分别与相应的引物对对1128(A群)、998(C群)和1005(B群)大质粒进行PCR扩增得到的片段进行杂交,结果表明:各引物对的1128(A群)、998(C群)和1005(B群)的PCR产物都与相应的pO157c探针杂交。
2.3 1128、998和1005菌株致病性大质粒酶切位点分布
以上PCR结果表明1128(A群)和998(C群)致病性大质粒变异区域完全相同,这与它们的HindⅢ和BglⅡ的酶切图谱差异明显相矛盾。通过对1128和998的质粒进行电泳分析发现998菌中含有另外一个稍小的质粒(图4)。由此推测1128和998的致病性大质粒酶切图谱相同,是小质粒导致998致病性大质粒的HindⅢ酶切图谱比933的多出一条带。
图4 O157 ∶ H7大肠埃希菌1128和998的质粒图谱
Figure 4 Plasmid patterns of E. coli O157 ∶ H7 strains 1128 and 998 注:M:DNA/HindⅢ Marker; 1.998;2.1128。
2.4 变异区DNA的克隆及测序分析
采用引物对5、25和27-U/28-L对1128和998的致病性大质粒进行PCR扩增,将经过纯化的PCR产物与pGEM-T克隆载体连接,转入JM109。将1128用以上3对引物的PCR扩增产物的克隆子分别命名为pV5-1128、pV25-1128和pV278-1128。同样,998对应的克隆子分别为pV5-998、pV25-998和pV278-998。引物对25对1005致病性大质粒的PCR扩增产物的克隆子命名为pV25-1005。对这些克隆子进行序列测定。克隆子pV5-1128、pV25-1128、pV278-1128的序列测定结果分别与pV5-998、pV25-998、pV278-998的完全相同。
将pV5-1128和pV5-998的测序结果分别与EDL933致病性大质粒对应的序列进行比较,发现1128和998的致病性大质粒在引物对5的扩增区域中含有一个长为1310 bp的片段。该片段可能为一个完整的插入序列,将其命名为IS1310。正是由于该插入序列导致了引物对5对1128和998致病性大质粒的扩增产物变大,该片段的插入引入了一个HindⅢ酶切位点。
pV278-1128和pV278-998的序列测定结果表明1128和998致病性大质粒缺失了933质粒上58 241~60 646之间的2406 bp的片段,从而使1128和998致病性大质粒在该片段上的BglⅡ酶切位点丧失,导致1128和998致病型大质粒BglⅡ酶切图谱发生改变,这也说明了1128和998致病性大质粒BglⅡ酶切产物与探针P27和P28杂交结果相同、与pO157不同的原因。引物27-L和28-U的结合位置恰好位于缺失的片段内,这与引物对27和28对1128和998的大质粒的PCR扩增均为阴性的结果是一致的。
pV25-1128和pV25-998的序列测定表明:1128和998的致病性大质粒相当于在EDL933致病性大质粒的53 268和53 269之间、54 578和54 579之间分别多出635 bp和116 bp,635 bp片段内有一个BglⅡ酶切位点。同源性检索结果提示635 bp和116 bp的片段可能为F质粒的成分。
1005的致病性大质粒引物对25的PCR扩增产物比pO157的多出639 bp,多出的核苷酸引入了一个BglⅡ酶切位点。将引物对25的扩增区段进行分析,发现在该区段内,1128/998和1005大质粒中含有另外一个与IS1310完全相同的序列。
3 讨论
根据O157 ∶ H7大肠埃希菌的致病大质粒HindⅢ酶切图谱将60株大肠埃希菌O157 ∶ H7分为5个群:A群(42株)、B群(11株)、C群(4株)、D群(2株)和E群(1株)。A群的1128和C群的998的致病大质粒的完全相同,造成它们之间HindⅢ酶切图谱不同的原因是998菌中含有另外一个比大质粒稍小的质粒。因此,按照致病性大质粒HindⅢ酶切图进行分群时,A群和C群应该归为同一个群。
菌株1006中引物3、29和30能够扩出预期大小的片段,可能是该质粒含有与pO157同源的片段。由于1006检测不到溶血素基因,而且绝大多数引物的PCR扩增结果均为阴性,因此怀疑1006的大质粒与O157 ∶ H7大肠埃希菌致病性大质粒的同源性较低,可能为其他类型的大质粒。 在1128(A群)和998(C群)的大质粒中存在两个IS1310拷贝,在1005(B群)的质粒中含有一个IS1310拷贝,而在933大质粒中含有一个与IS629[8]同源的序列。IS1310与IS3411[9]的同源性达95.6%,与IS629的同源性为96.0%,这些插入序列两侧具有十分相似的转座酶和反向重复,它们可能采用相同的转座机制进行转座。通过比较它们在不同位置的插入位点,发现它们的插入位点是随机的,而且造成2个或者3个或者4个碱基的正向重复。
大肠埃希菌O157 ∶ H7的致病大质粒被认为是由F质粒进化而来的,但对其进化趋势人们还知之甚少。在引物对25扩增区域内的IS1310或IS629样序列的侧翼序列都为F质粒的同源序列,在这些侧翼序列对应的F质粒上相应的序列中不含有IS1310或IS629,因此,认为IS1310或IS629样序列是后来插入到O157 ∶ H7大肠埃希菌致病性大质粒中的。这些结构是造成O157 ∶ H7大肠埃希菌的致病大质粒变异的主要因素之一。
参考文献
[1] Schmidt H, Karch H, Beutin L. The large-sized plasmids of enterohemorrhagic Escherichia coli O157 strains encode hemolysins which are presumably members of the E. coli α-hemolysin family[J]. FEMS Microbiol Lett,1994,117:189-196.
[2] Lim JY, Yoon J, Hovde CJ. A brief overview of Escherichia coli O157:H7 and its plasmid O157[J]. J Microbiol Biotechnol,2010,20(1):5-14.
[3] Brunder W, Schmidt H, Karch H. KatP, a novel catalase-peroxidase encoded by the large plasmid of enterohaemorrhagic Escherichia coli O157:H7[J]. Microbiology,1996,14:3305-3315.
[4] Brunder W, Schmidt H, Karch H. EspP, a novel extracellular serine protease of enterohaemorrhagic Escherichia coli O157:H7 cleaves human coagulation factor V[J]. Microbiology,1997,24:767-778.
[5] Schmidt H, Beutin L, Karch H. Molecular analysis of the plasmid-encoded hemolysin of Escherichia coli O157:H7 strain EDL933[J]. Infect Immun,1995,63:1055-1061.
[6] Puttamreddy S, Cornick NA, Minion FC. Genome-wide transposon mutagenesis reveals a role for pO157 genes in biofilm development in Escherichia coli O157:H7 EDL933[J]. Infect Immun,2010,78(6):2377-2384.
[7] Burland V, Shao Y, Perna NT, et al. The complete DNA sequence and analysis of the large virulence plasmid of Escherichia coli O157:H7[J]. Nucleic Acids Res,1998,26(18):4196-4204.
[8] Rump LV, Fischer M, Gonzalez-Escalona N. Prevalence, distribution and evolutionary significance of the IS629 insertion element in the stepwise emergence of Escherichia coli O157:H7[J]. BMC Microbiol,2011,11:133.
[9] Ishiguro N, Sato G. Nucleotide sequence of insertion sequence IS3411, which flanks the citrate utilization determinant of transposon Tn3411[J]. J Bacteriol,1988,170(4):1902-1906.
[2] Lim JY, Yoon J, Hovde CJ. A brief overview of Escherichia coli O157:H7 and its plasmid O157[J]. J Microbiol Biotechnol,2010,20(1):5-14.
[3] Brunder W, Schmidt H, Karch H. KatP, a novel catalase-peroxidase encoded by the large plasmid of enterohaemorrhagic Escherichia coli O157:H7[J]. Microbiology,1996,14:3305-3315.
[4] Brunder W, Schmidt H, Karch H. EspP, a novel extracellular serine protease of enterohaemorrhagic Escherichia coli O157:H7 cleaves human coagulation factor V[J]. Microbiology,1997,24:767-778.
[5] Schmidt H, Beutin L, Karch H. Molecular analysis of the plasmid-encoded hemolysin of Escherichia coli O157:H7 strain EDL933[J]. Infect Immun,1995,63:1055-1061.
[6] Puttamreddy S, Cornick NA, Minion FC. Genome-wide transposon mutagenesis reveals a role for pO157 genes in biofilm development in Escherichia coli O157:H7 EDL933[J]. Infect Immun,2010,78(6):2377-2384.
[7] Burland V, Shao Y, Perna NT, et al. The complete DNA sequence and analysis of the large virulence plasmid of Escherichia coli O157:H7[J]. Nucleic Acids Res,1998,26(18):4196-4204.
[8] Rump LV, Fischer M, Gonzalez-Escalona N. Prevalence, distribution and evolutionary significance of the IS629 insertion element in the stepwise emergence of Escherichia coli O157:H7[J]. BMC Microbiol,2011,11:133.
[9] Ishiguro N, Sato G. Nucleotide sequence of insertion sequence IS3411, which flanks the citrate utilization determinant of transposon Tn3411[J]. J Bacteriol,1988,170(4):1902-1906.
|
扩展功能
|
|
| 本文信息 | |
| PDF全文 | |
| HTML全文 | |
| 参考文献 | |
| 服务与反馈 | |
| 加入引用管理器 | |
| 引用本文 | |
| Email Alert | |
| 本文作者相关文章 | |
| 徐雪芳 | |
| 伍建宏 | |
| 刘学通 | |
| 熊衍文 | |
| 徐建国 | |
| PubMed | |
| Article by XU Xue-fang | |
| Article by WU Jian-hong | |
| Article by LIU Xue-tong | |
| Article by XIONG Yan-wen | |
| Article by XU Jian-guo | |