1. Guyiyang Medical University, Guiyang 550004, Guizhou, China;
2. State Key Laboratory for Communicable Disease Prevention and Control, Institute for Communicable Disease Prevention and Control, Chinese Center for Disease Control and Prevention, Beijing 102206, China
Distribution of virulence-related genes of selected Listeria monocytogences strains isolated from food samples in China
HE Chun-yue1,2, WANG Yan2, WANG Tian-shu1,2, YE Zheng-xing2, DAI Hang2, XU Ke3, YU Hua3, WANG Yong-lu4, ZHANG Lan-rong5, WANG Lian-xiu6, ZENG Ying-chun7, XU Xue-bing8, WANG Hong9, CHEN Wei-wei10, RAN Lu11, YAO Ying-bo12, WANG He1, YE Chang-yun2
Abstract
Objective To understand the distribution of virulence related genes of Listeria monocytogences strains isolated from food samples in China. Methods A total of 383 L. monocytogences strains were isolated from food samples, including raw meat, cooked food, vegetable, cold drink and aquatic product, in 14 provinces or municipalities. Polymerase chain reaction and DNA dot hybridization were conducted to detect 55 virulence related genes of L. monocytogences. Results The detection rates of the 50 virulence related genes were 100%, the detection rates of inlF, fbpA and mprF genes were 99.73%, the detection rate of aut gene was 99.22% and the detection rate of vip gene was 98.17%. Conclusion Most virulence related genes were detected in the 383 L. monocytogences strains from food samples in China, the deletion patterns of some virulence genes varied.
Keywords:
Listeria monocytogences
virulence related gene
polymerase chain reaction
DNA dot hybridization
中国部分食品来源单增李斯特菌中致病相关基因的分布研究
贺春月1,2, 王艳2, 王天姝1,2, 叶正兴2, 代航2, 许柯3, 俞骅3, 汪永禄4, 张兰荣5, 王连秀6, 曾莹春7, 许学斌8, 王红9, 陈伟伟10, 冉陆11, 姚颖波12, 王和1, 叶长芸2
1. 贵阳医学院微生物学教研室, 贵州 贵阳 550004;
2. 中国疾病预防控制中心传染病预防控制所, 传染病预防控制国家重点实验室, 北京 昌平 102206;
3. 浙江省杭州市疾病预防控制中心;
4. 安徽省马鞍山市疾病预防控制中心;
5. 北京市通州区疾病预防控制中心;
6. 北京市昌平区疾病预防控制中心;
7. 湖北省武汉市疾病预防控制中心;
8. 上海市疾病预防控制中心;
9. 四川省自贡市疾病预防控制中心;
10. 福建省疾病预防控制中心;
11. 中国疾病预防控制中心;
12. 云南省玉溪市疾病预防控制中心
2. 中国疾病预防控制中心传染病预防控制所, 传染病预防控制国家重点实验室, 北京 昌平 102206;
3. 浙江省杭州市疾病预防控制中心;
4. 安徽省马鞍山市疾病预防控制中心;
5. 北京市通州区疾病预防控制中心;
6. 北京市昌平区疾病预防控制中心;
7. 湖北省武汉市疾病预防控制中心;
8. 上海市疾病预防控制中心;
9. 四川省自贡市疾病预防控制中心;
10. 福建省疾病预防控制中心;
11. 中国疾病预防控制中心;
12. 云南省玉溪市疾病预防控制中心
摘要
目的 了解中国食品来源部分单增李斯特菌(Listeria monocytogences, LM)中致病相关基因的分布情况。 方法 383株LM分离自2002-2011年中国14省(市)的生肉、熟食、蔬菜、冷饮、水产等食品。采用PCR(polymerase chain reaction)和DNA打点杂交方法在单增李斯特菌中进行了55个致病相关基因的检测。 结果 50个致病相关基因检出率均为100%,inlF、fbpA和mprF基因检出率为99.73%,aut基因检出率为99.22%(380/383),vip基因检出率为98.17%(376/383)。 结论 383株食品来源单增李斯特菌中,大部分致病相关基因均存在,部分毒力基因的缺失存在多样性。
内容大纲
-
1 材料与方法
- 1.1 实验材料
- 1.2 方法
- 1.2.1 DNA提取
- 1.2.2 聚合酶链反应(PCR)引物设计与合成
- 1.2.3 PCR反应体系与反应程序
- 1.2.4 阳性对照菌株基因测序验证
- 1.2.5 基因探针的测序验证
- 1.3 DNA杂交
- 1.4 统计学分析
- 1.5 毒力相关基因缺失菌株MLST分析
- 2.1 检测结果
- 2.2 毒力基因缺失与菌株来源的相关性
- 2.3 毒力基因缺失与菌株血清型相关性
- 2.4 毒力基因分布规律
细菌感染宿主后,会诱导宿主的免疫反应识别 并被宿主清除,但LM能逃避并调控宿主的免疫反应,为自身创造合适的胞内生存环境[6]。通过对LM与无害李斯特菌基因组比较后发现,LM具有许多特有的毒力相关蛋白[7]。LM的致病力与毒力蛋白密切相关,感染过程中的每个阶段均由相关毒力因子调控,包括负责毒力基因的表达和调控的因子,应激耐受与生存相关的因子,以及侵袭、粘附相关的因子等。关于耐受环境压力、毒力基因调控、细胞内增殖、逃避宿主免疫反应等致病相关基因在我国食品来源LM中的分布情况尚不是很清楚,本研究拟对分离自我国14省(市)(北京、浙江、湖北、福建、安徽、河南、四川、重庆、江苏、上海、广东、广西、吉林、云南)的383株食品来源的LM进行了毒力相关基因的检测,以了解这部分菌株中致病相关基因的分布情况,从而为其致病性的评估和研究提供参考。
1 材料与方法
1.1 实验材料 1.1.1 菌株
LM参考株54003购自中国药品生物鉴定所、EGD-e购自美国标准生物品收藏中心(ATCC),其余383株LM来自14个省(市)(北京、浙江、湖北、福建、安徽、河南、四川、重庆、广西 、吉林、江苏、上海、深圳、云南),分别于2002-2011年从生肉、熟食、蔬菜、冷饮、水产等食品中分离获得,经生化鉴定证实为LM,血清型分布分别为:1/2a型115株、1/2b型65株、1/2c型121株、3a型42株、3b型7株、4b型11株、4c型11株、4d型8株、4a型1株、4ab型1株、未知血清型1株。
1.1.2 试剂
脑心浸液(北京陆桥技术有限公司);脱氧核糖核酸(DNA)提取试剂盒(北京康为世纪生物科技有限公司);r-taq酶(大连宝生物有限公司);琼脂糖(上海易通生命科学仪器设备有限责任公司);非放射性ECL直接核酸标记及检测杂交试剂盒(美国GE公司);杂交显影液和定影液(北京天海开拓科技有限责任公司);杂交膜(美国GE公司);2×Taq MasterMix(北京康为世纪生物科技有限公司)。
1.2 方法
1.2.1 DNA提取
将实验菌株接种于脑心液体培养基中,37 ℃过夜培养。培养物用细菌DNA提取试剂盒按操作说明进行染色体DNA的提取。
1.2.2 聚合酶链反应(PCR)引物设计与合成
针对LM的多个致病相关基因,从GenBank上下载基因序列,用Primer 5.0软件设计引物。引物由上海生物工程技术有限公司合成。
表1 致病相关基因的功能和引物
Table 1 Function of 55 virulence-related genes and PCR primers used in this study
| 基因 | 功能 |
| prfA | 毒力基因的表达和调控 |
| sigB、gadB、bsh、clpB、clpC、clpE、hfq | 耐受环境的压力应答 |
| virR(1) | 正向调控毒力基因的转录 |
| degU、gmaR、mogR | 耐受不同的环境温度 |
| hly、plcA、plcB、mpl、prsA(2)、svpA、sipZ、lsp、actA、srtB、dltA | 逃避巨噬细胞吞噬 |
| hpt、pycA、fri、relA、oppA | 细胞内增殖相关 |
| sod、pgdA | 逃避宿主的免疫反应 |
| lipA、agrA、agrB、agrC、lmaB | 与细胞内的毒力有关 |
| ami、iap、srtA、lap、fbpA、ctaP、lapB、recA、vip、aut、lgt、gtcA、mprF、flaA、inlA、inlB、inlC(2)、inlD1(3)、inlD2(4)、inlF、inlH(5)、inlJ(6) | 与细菌的侵袭、粘附相关 |
1.2.3 PCR反应体系与反应程序
PCR反应体系:DNA模板1.0 μl(20~50 ng)、10 μmol/L上、下游引物各1.0 μl、2×Taq MasterMix 10 μl、RNase-Free water 7 μl、总体积20 μl。PCR反应程序:94 ℃预变性5 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 2 min,30个循环;72 ℃延伸10 min,4 ℃保存。反应结束后,经1.0%琼脂糖凝胶电泳,6 V/cm,30 min,用凝胶成像系统读取扩增结果。
1.2.4 阳性对照菌株基因测序验证
阳性参考菌株EGD-e、54003的55种毒力基因的PCR产物由上海生工进行测序,序列比对证实为本研究所需扩增产物,可作为PCR检测的阳性对照。
1.2.5 基因探针的测序验证
毒力相关基因PCR扩增阴性菌株需用DNA打点杂交方法进行验证,分别以参考菌株54003、EDG-e为模板进行杂交探针基因的PCR扩增,将PCR产物经纯化试剂盒纯化回收后,由上海生工进行测序及序列比对,证实为本研究所需的目的片段,作为DNA打点杂交验证的探针使用。
1.3 DNA杂交
1.3.1 目的基因片段的纯化
按照QIAGEN DNA纯化试剂盒说明进行目的基因片段PCR产物的纯化。
1.3.2 探针标记
DNA标记采用非放射性酵素标记,具体步骤为:取20 ng/μl纯化后的探针10 μl,99 ℃ 5 min,冰浴2 min,加入20 μl DNA Labelling reagent混匀,再加入20 μl Glutar aldehyde(戊二醛),混匀,离心1 min,37 ℃孵育10 min。
1.3.3 杂交
(1)取6 μl细菌DNA模板点于已划好格的杂交膜上,晾干后,用两张滤纸夹好,80 ℃烘箱中固定2 h。(2)杂交前,将膜置于0.4 mol/L NaOH液体中浸湿2 min,再用5×SSC洗膜10 min。(3)将膜转入杂交筒内,倒入预杂交液,42 ℃预杂交10 min。把预杂交液倒掉,加入标记的探针42 ℃杂交过夜。(4)杂交后加入Primary washing Buffer 50 ml,42 ℃洗膜2次,每次20 min;再加入2×SSC 250 ml,室温洗膜2次,每次5 min。(5)检测:将杂交膜和显色试剂混匀,放于胶片上暗室内曝光,显影后定影,读取杂交结果。
1.4 统计学分析
采用SPSS 15.0软件对不同食品中分离的菌株毒力基因缺失差异情况进行Pearson χ2检验分析,以P<0.05为差异有统计学意义。
1.5 毒力相关基因缺失菌株MLST分析
根据管家基因绘制系统进化树,了解缺失毒力基因分布规律。
2 结果
2.1 检测结果 383株LM 55个致病相关基因的分布情况:50个致病相关基因(包括prfA、virR、sigB、hfq、inlA、inlB、hly、plcA、plcB、actA、hpt等)在所有实验菌株都存在,见表2。此外,6个基因(inlD、plcB、gtcA、iap、inlB、prfA)通过杂交验证后,也存在于所有菌株中。5个基因在383株菌中出现不同的检出率,其中vip检出率为98.17%(376/383株),aut检出率为99.22%(380/383株),inlF、fbpA、mprF检出率为99.74%(382/383株)。
2.2 毒力基因缺失与菌株来源的相关性
不同食品中分离的菌株毒力基因缺失差异无统计学意义(χ2 = 0.482,P>0.05),从生食品和其他食品中分离获得的LM的毒力基因缺失种类的差异也无统计学意义。
2.3 毒力基因缺失与菌株血清型相关性
通过对LM血清型与其毒力基因缺失规律之间的比较发现,发生毒力相关基因缺失的13株LM,9株为1/2a血清型,1/2c、4b、4c、4d血清型各有1株。
表2 比较使用PCR和PCR+DNA打点杂交对383株LM的检测结果
Table 2 Comparison of detection of virulence-related genes of 383 L. monocytogenes strains by PCR and DNA dot hybridization
| 致病相关基因 | PCR检测结果 | 交检测结果 | |||
| 株阳性菌株数(株)数 | 检出率(%) | 株阳性菌株数(株)数 | 检出率(%) | ||
| vip | 376 | 98.17 | 376 | 98.17 | |
| aut | 380 | 99.22 | 380 | 99.22 | |
| inlF | 374 | 97.65 | 382 | 99.74 | |
| fbpA | 372 | 97.13 | 382 | 99.74 | |
| mprF | 381 | 99.48 | 382 | 99.74 | |
| inlD | 373 | 97.39 | 383 | 100.00 | |
| plcB | 382 | 99.74 | 383 | 100.00 | |
| gtcA | 381 | 99.48 | 383 | 100.00 | |
| iap、inlB、prfA | 382 | 99.74 | 383 | 100.00 | |
| inlA、inlC、inlJ、inlH、hly、actA、srtA、plcA、sigB、srtB、agrA、agrB、agrC、mpl、hfq、hpt、flaA、bsh、degU、gadB、ctaP、mogR、clpB、clpC、clpE、lapB、lipA、prsA2、virR、lap、dltA、recA、lgt、sipZ、lsp、pycA | 383 | 100.00 | N(1) | N | |
2.4 毒力基因分布规律
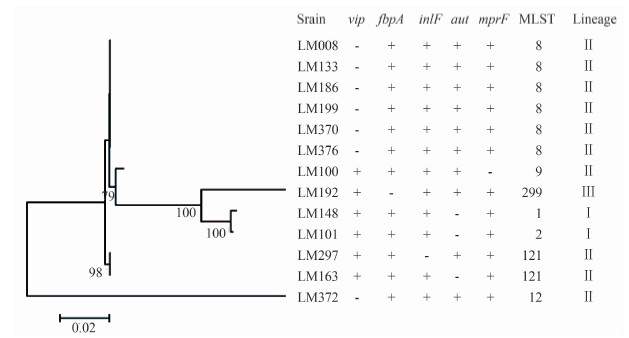
根据毒力相关基因的携带情况可将383株食源性LM分为6种毒力基因型,见表3。96.08%(368/383)的分离株携带55个毒力相关基因,属于毒力基因6型; 1.83%(7/383)的分离株缺失了vip,属于毒力基因1型;0.78%(3/383)的分离株缺失了aut, 属于毒力基因4型;其余3个型各有1株菌出现相应毒力基因缺失,见表3。毒力相关基因缺失型别与菌株序列型别间没有相关性,见图1。
表3 383株食源性LM的毒力相关基因携带型别
Table 3 Type of virulence-related genes of 383 L. monocytogenes strains
| 毒力基因携带型别 | 缺失的毒力基因 | 菌株数(株) | 构成比(%) |
| 1型 | vip | 7 | 1.83 |
| 2型 | inlF | 1 | 0.26 |
| 3型 | fbpA | 1 | 0.26 |
| 4型 | aut | 3 | 0.78 |
| 5型 | mprF | 1 | 0.26 |
| 6型 | -(1) | 370 | 96.61 |
图1 实验菌株根据7个管家基因的系统发生树
Figure 1 Phylogenetic tree of 7 housekeeping genes of isolates 注:MLST表示多位点序列分型。MLST管家基因包括abcZ、blgA、cat、dapE、dat、ldh、lhkA。 图中“+”表示致病基因检测结果为阳性,“-”表示致病基因检测的结果为阴性。
3 讨论
本研究使用PCR和DNA打点杂交相结合的方法,对从14个省/市不同食品来源383株LM进行了55种致病相关基因的检测。结果显示,由于分离菌株在不同的生长环境和进化过程中发生了部分基因的缺失或突变,当使用PCR方法检测时,会出现漏检的情况。而PCR结合DNA打点杂交方法,能够减少基因变异所导致的假阴性结果。对比杨洋等[13]的检测结果,我们发现,同样是使用PCR检测方法,本实验致病相关基因的检出率高,这可能与本实验设计的引物在该致病基因的保守区域有关。Liu等[12]发现,inlJ在LM的部分4a血清型菌株中不存在。我们的检测结果发现,inlJ在LM的4a血清型菌株中都存在。Doumith等[14]发现,inlC、inlF、inlH在LM的1/2a血清型都存在,在部分4a血清型菌株中也不存在,对比我们的检测结果,inlF在1株血清型为1/2a的LM中不存在,在部分4a血清型菌株中也不存在, inlC、inlH在LM的所有血清型菌株中都存在。我们的检测结果证实,所有实验菌株都能检测到的致病相关基因(100%)有50个,包括编码毒力基因的表达和调控(prfA)、耐受环境的压力应答(sigB、gadB、bsh、clpB、clpC、clpE)、正向调控毒力基因的转录(virR)、耐受不同的环境温度(degU、gmaR、mogR)、与细菌的侵袭、粘附相关(ami、iap、srtA、lap、fbpA、ctaP、lapB、recA、vip、aut、lgt、gtcA、mprF、flaA)、内化素蛋白家族(inlA、inlB、inlC、inlD、inlF、inlH、inlJ)、溶解细胞吞噬体膜使LM逃离到胞浆(hly、plcA、plcB、mpl、svpA、sipZ、lsp、actA、srtB、dltA)、细胞内增殖(pycA、hpt、relA、prsA2、oppA)、逃避宿主的免疫反应(sod、pgdA)等的基因均为阳性,提示这些致病相关基因在LM耐受环境、侵袭和粘附宿主细胞、在胞内生存复制、逃逸吞噬胞液及细胞间的扩散等方面发挥重要作用,对LM的生存是必需的。6个毒力因子编码基因prfA、plcA、hly、mpl、actA、plcB所在的LIPI-1毒力岛与LM的致病力密切相关,这对细菌在细胞内的生存是必不可少的[15],本研究的检测结果与此发现一致。部分菌株在5个致病相关基因(vip、aut、fbpA、inlF、mprF)中存在不同程度的缺失,这些基因与细菌的侵袭、粘附以及在细胞中的毒力有关,推测这些毒力基因在LM中具有不稳定性,在不同的环境中以及与不同的宿主相互作用时发生了缺失。但是Doumith等[14]发现毒力基因vip存在于LM的家系Ⅰ、Ⅱ的菌株中(其中包括与人类疾病密切相关的血清型1/2a、1/2b和4b),我们的检测结果发现毒力基因vip在7株1/2a血清型的LM株中不存在,提示我国食源性LM中一些菌株的毒力基因携带情况比较独特。
不同菌株间毒力基因分布规律显示,383株食源性LM可分为6个毒力基因型,其中6型是毒力基因型的优势型别,368株此型菌株均携带55个致病相关基因。毒力基因型中发生缺失基因菌株数较多的是1型,均为1/2a型血清型菌株;其次是4型,其血清型分别为1/2a、4b、4d。 致病相关基因缺失型与菌株序列型相关分析结果显示两者不具有相关性。致病相关基因的缺失可能与菌株不同生存环境所带来的压力不同所致,其机制有待于进一步研究证实。
参考文献
[1] Vazquez Boland JA, Kuhn M, Berche P,et al. Listeria pathogenesis and molecular virulence determinants[J]. Clin Microbiol Rev, 2001,14(3):584-640.
[2] Dussurget O. New Insights into Determinants of Listeria Monocytogenes Virulence[J]. Int Rev Cell Mol Biol, 2008, 270:1-38.
[3] Dalton CB,Austin CC,Sobel J,et al.An outbreak of gastroenteritis and fever due to Listeria monocytogenes in milk[J]. N Engl Med, 1997,336(2):100-105
[4] Aureli P,Fiorucci GC,Caroli D,et al.An outbreak of febrile gastroenteritis associated with corn contaminated by Listeria monocytogenes[J]. N Engl Med,2000,342(17):1236-1241.
[5] Outbreak of Listeria monocytogense infections associated with pasteurized milk from a local dairy-Massachusetts,2007[J]. MMWR, 2008,57(40):1097-1100.
[6] Li AH, Ye CY.Research advance on pathopoiesis mechanism of Listeria monocytogenes[J]. Disease Surveillance, 2011, 26(11):914-919.(in Chinese) 李爱华,叶长芸.单核细胞增生性李斯特菌致病相关机制的研究进展[J].疾病监测,2011,26(11):914-919.
[7] Cabanes D, Dehoux P, Dussurget O, et al. Surface proteins and the pathogenic potential of Listeria monocytogenes[J]. Trends Microbiol, 2002,10(5):238-245.
[8] Mandin P, Fsihi H, Dussurget O, et al.VirR, a response regulator critical for Listeria monocytogenes virulence[J]. Mol Microbiol, 2005, 57(5):1367-1380.
[9] Jia Y, Nightingale KK, Boor KJ, et al.Distribution of internalin gene profiles of Listeria monocytogenes isolates from different sources associated with phylogenetic lineages[J]. Foodborne Pathog Dis, 2007,4(2):222-232.
[10] Markkula A, Lindstrőm M, Korkeala H. Listeria monocytogenes Serotypes 1/2c and 3c Possess inlH[J]. Foodborne Pathog Dis, 2011,8(10):1125-1129.
[11] Chen J,Luo X,Jiang L,et al.Molecular characteristics and virulence potential of Listeria monocytogenes isolates from Chinese food systems[J]. Food Microbiol, 2009,103-111.
[12] Liu D, Ainsworth AJ, Austin FW, et al.Characterization of virulent and avirulent Listeria monocytogenes strains by PCR amplification of putative transcriptional regulator and internalin genes[J]. J Med Microbiol, 2003,52(Pt 12):1065-1070.
[13] Yang Y, Fu P, Guo YC, et al.Virulent gene prevalence of foodborne Listeria monocytogenes in China in 2005[J]. China Preventive Medicine, 2010,44(12):1097-1101.(in Chinese) 杨洋,付萍,郭云昌, 等. 2005年中国食源性单核细胞增生李斯特菌毒力基因分布[J]. 中华预防医学杂志,2010,44(12):1097-1101.
[14] Doumith M, Cazalet C, Simoes N, et al.New Aspects Regarding Evolution and Virulence of Listeria monocytogenes Revealed by Comparative Genomics and DNA Arrays[J].Infect Immun, 2004,72(2):1072-1083.
[15] Vazquez Boland JA, Dominguez Bernal G, Gonzalez Zorn B, et al.Pathogenicity islands and virulence evolution in Listeria[J]. Microbes Infect, 2001,3(7):571-584.
[2] Dussurget O. New Insights into Determinants of Listeria Monocytogenes Virulence[J]. Int Rev Cell Mol Biol, 2008, 270:1-38.
[3] Dalton CB,Austin CC,Sobel J,et al.An outbreak of gastroenteritis and fever due to Listeria monocytogenes in milk[J]. N Engl Med, 1997,336(2):100-105
[4] Aureli P,Fiorucci GC,Caroli D,et al.An outbreak of febrile gastroenteritis associated with corn contaminated by Listeria monocytogenes[J]. N Engl Med,2000,342(17):1236-1241.
[5] Outbreak of Listeria monocytogense infections associated with pasteurized milk from a local dairy-Massachusetts,2007[J]. MMWR, 2008,57(40):1097-1100.
[6] Li AH, Ye CY.Research advance on pathopoiesis mechanism of Listeria monocytogenes[J]. Disease Surveillance, 2011, 26(11):914-919.(in Chinese) 李爱华,叶长芸.单核细胞增生性李斯特菌致病相关机制的研究进展[J].疾病监测,2011,26(11):914-919.
[7] Cabanes D, Dehoux P, Dussurget O, et al. Surface proteins and the pathogenic potential of Listeria monocytogenes[J]. Trends Microbiol, 2002,10(5):238-245.
[8] Mandin P, Fsihi H, Dussurget O, et al.VirR, a response regulator critical for Listeria monocytogenes virulence[J]. Mol Microbiol, 2005, 57(5):1367-1380.
[9] Jia Y, Nightingale KK, Boor KJ, et al.Distribution of internalin gene profiles of Listeria monocytogenes isolates from different sources associated with phylogenetic lineages[J]. Foodborne Pathog Dis, 2007,4(2):222-232.
[10] Markkula A, Lindstrőm M, Korkeala H. Listeria monocytogenes Serotypes 1/2c and 3c Possess inlH[J]. Foodborne Pathog Dis, 2011,8(10):1125-1129.
[11] Chen J,Luo X,Jiang L,et al.Molecular characteristics and virulence potential of Listeria monocytogenes isolates from Chinese food systems[J]. Food Microbiol, 2009,103-111.
[12] Liu D, Ainsworth AJ, Austin FW, et al.Characterization of virulent and avirulent Listeria monocytogenes strains by PCR amplification of putative transcriptional regulator and internalin genes[J]. J Med Microbiol, 2003,52(Pt 12):1065-1070.
[13] Yang Y, Fu P, Guo YC, et al.Virulent gene prevalence of foodborne Listeria monocytogenes in China in 2005[J]. China Preventive Medicine, 2010,44(12):1097-1101.(in Chinese) 杨洋,付萍,郭云昌, 等. 2005年中国食源性单核细胞增生李斯特菌毒力基因分布[J]. 中华预防医学杂志,2010,44(12):1097-1101.
[14] Doumith M, Cazalet C, Simoes N, et al.New Aspects Regarding Evolution and Virulence of Listeria monocytogenes Revealed by Comparative Genomics and DNA Arrays[J].Infect Immun, 2004,72(2):1072-1083.
[15] Vazquez Boland JA, Dominguez Bernal G, Gonzalez Zorn B, et al.Pathogenicity islands and virulence evolution in Listeria[J]. Microbes Infect, 2001,3(7):571-584.