Characterization of Salmonella enteritidis with pulsed-field gel electrophoresis in China
LOU Jing, DIAO Bao-wei, LI Jie, KAN Biao, YAN Mei-ying
State Laboratory for Communicable Disease Prevention and Control, Chinese Center for Disease Control and Prevention, Beijing 102206, China
Abstract
Objective To understand the pulsed-field gel electrophoresis (PFGE) patterns of the clinical isolates of Salmonella enteritidis and their epidemiological characteristics in China and provide evidence for the laboratory based detection of food borne diseases outbreak and response effort. Methods Molecular typing of 981 Salmonelle enteritidis strains isolated in 13 provinces from 2004 to 2012 were conducted according to PFGE protocol published on PulseNet and epidemiological analysis was conducted. Results The chromosomal DNA of 981 Salmonella enteritidis strains were digested with XbaⅠrestriction endonuclease and 126 PFGE patterns were observed. The discriminatory index (D) was 0.8336 for PFGE typing method. The predominate patterns of PFGE included JEGX01.CN0003, JEGX01.CN0001, JEGX01.CN0002, JEGX01.CN0005 and JEGX01.CN0016. Of 126 patterns, JEGX01.CN0003、JEGX01.CN0001and JEGX01.CN0150 were related with outbreaks caused by Salmonella enteritidis. Common bands (±0.8%) of 655 kbp, 325 kbp, 310 kbp, 245 kbp, 178 kbp and 110 kbp were found in the predominant patterns. Conclusion There were predominant PFGE patterns among the Salmonella enteritidis strains isolated in China, however, double or multiple endonucleotidases analysis of PFGE is necessary for outbreak detection due to the limited discriminatory power of single XbaⅠdigestion.
中国肠炎沙门菌脉冲场凝胶电泳分子分型数据库的分析
娄静, 刁保卫, 李杰, 阚飙, 闫梅英
中国疾病预防控制中心传染病预防控制所, 北京 102206
摘要
目的 了解近几年我国肠炎沙门菌临床分离株的流行病学及脉冲场凝胶电泳(PFGE)分子分型特点,为以实验室为基础的食源性疾病暴发的发现及疫情控制提供依据。 方法 根据PulseNet公布的沙门菌PFGE分型方案,对2004-2012年分离自我国13个省(直辖市)的981株肠炎沙门菌进行流行病学及PFGE分子分型分析。 结果 981株肠炎沙门菌经XbaⅠ酶切,PFGE获得126种带型,其分辨能力(D值)为0.8336。优势带型为JEGX01.CN0003、JEGX01.CN0001、JEGX01.CN0002、JEGX01.CN0005和JEGX01.CN0016。暴发相关带型是JEGX01.CN0003、JEGX01.CN0001和JEGX01.CN0150。肠炎沙门菌优势带型PFGE的共有条带分别为(±0.8%)655、325、310、245、178和110 kbp。 结论 我国肠炎沙门菌临床分离株存在PFGE优势型别,PFGE对于肠炎沙门菌XbaⅠ单酶切分型能力有限,用于暴发确认或发现时需多种酶切联合使用,提高分辨能力。
内容大纲
-
1 材料与方法
2 结果
- 2.1 肠炎沙门菌PFGE分子分型特征
- 2.1.1 肠炎沙门菌PFGE的带型统计
- 2.1.2 主要优势带型的年代及地区分布
- 2.2 肠炎沙门菌PFGE的特征性条带
- 2.3 肠炎沙门菌暴发相关带型
脉冲场凝胶电泳(pulse-field gel electrophoresis,PFGE)已广泛应用于沙门菌的分子分型研究,在沙门菌暴发调查、溯源工作中发挥着重要作用 ,对于肠炎沙门菌,目前已有成熟的国际通用标准化PFGE分型方案, PulseNet China是以PFGE等为技术手段,对中国细菌性传染病病原体的分型分析和信息交流的实验室监测网络体系。沙门菌分离株的PFGE数据库为PulseNet China的重要组成,通过网络实验室对监测中分离菌株进行PFGE分析,并上传图谱至中心数据库,形成实时监测数据,进行对比分析,发现成簇带型的菌株,提示暴发调查及预警。
本研究中,对多年来PulseNet China中的肠炎沙门菌分子分型结果进行总结,对肠炎沙门菌的流行病学分布规律及PFGE分型特点进行分析。
1 材料与方法
1.1 菌株来源 肠炎沙门菌共981株,来源于北京、上海、四川、广东、河南、湖北等13个省(直辖市),2004—2012年菌株数量分别为6、13、78、113、216、239、212、83和21株。
1.2 PFGE分型
所有肠炎沙门菌依据国际实验室分子分型监测网络PulseNet中沙门菌PFGE分型标准化方案进行操作。使用限制性内切酶XbaⅠ进行酶切,获得的菌株PFGE图像录入BioNumerics(Version 5.1,Applied maths,Inc.)软件包进行处理,经校准后,使用非加权配对算术平均法(unweighted pair group average method,UPGMA)进行聚类,构建聚类树,分析菌株间的相似性。
1.3 D值计算
在 评价某种分型方法的分辨能力时通常采用基于Simpson的分辨系数 ,即D值来表示。其公式是:D=1- 1 N(N-1) ∑ s j=1 nj(nj-1), 其中N表示菌株总数,Nj为某一型别的菌株数。
2 结果
本次研究的981株肠炎沙门菌,经限制性内切酶XbaⅠ进行酶切,PFGE获得126种带型,D值为0.8336,说明XbaⅠ单酶切分型分辨能力较低。
2.1 肠炎沙门菌PFGE分子分型特征
2.1.1 肠炎沙门菌PFGE的带型统计
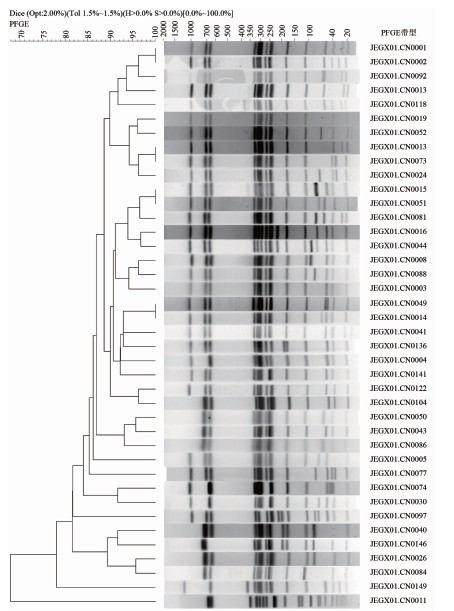
本次研究的981株肠炎沙门菌,共分为126个PFGE带型,总的相似度为55.51%,不同带型之间的差异较大。不同带型的菌株数量差异亦较大,菌株数从1~333不等。PFGE优势带型(包含菌株数n≥10)及包含的菌株数见图1。
图1 肠炎沙门菌优势带型PFGE聚类结果
Figure 1 Clustering results of predominant PFGE patterns of S. enteritidis strains
优势带型即包含10株菌株以上的带型共计11种,763株菌,占菌株总数的77.78%,前5位的优势带型共计653株,其中JEGX01.CN0003,333株;JEGX01.CN0001,206株; JEGX01.CN0002型,43株;JEGX01.CN0005,37株;JEGX01.CN0016,34株。
2.1.2 主要优势带型的年代及地区分布
前5位的优势带型分别为JEGX01.CN0003、JEGX01.CN0001、JEGX01.CN0002、JEGX01.CN0005和JEGX01.CN0016。在肠炎沙门菌的数据库中前5位优势带型所占的比例分别为33.94%、21.00%、4.38%、3.77%和3.47%。表1结果表明,JEGX01.CN0003遍布2004—2012年,优势带型主要集中在2006—2011年。主要优势带型在各地区分布不均,主要集中在上海、河南及北京等地区,见表2。
表1 主要优势带型的年代分布
Table 1 Time distribution of predominant PFGE patterns
| 主要优势带型 | 菌株数 | 2004年 | 2005年 | 2006年 | 2007年 | 2008年 | 2009年 | 2010年 | 2011年 | 2012年 | ||||||||||||||||||
| 菌株数 | 构成比(%) | 菌株数 | 构成比(%) | 菌株数 | 构成比(%) | 菌株数 | 构成比(%) | 菌株数 | 构成比(%) | 菌株数 | 构成比(%) | 菌株数 | 构成比(%) | 菌株数 | 构成比(%) | 菌株数 | 构成比(%) | |||||||||||
| JEGX01.CN0003 | 333 | 4 | 1.20 | 5 | 1.50 | 27 | 8.11 | 38 | 11.41 | 66 | 19.82 | 81 | 24.33 | 77 | 23.12 | 24 | 7.21 | 11 | 3.30 | |||||||||
| JEGX01.CN0001 | 206 | 0 | 0.00 | 0 | 0.00 | 23 | 11.17 | 21 | 10.19 | 42 | 20.39 | 73 | 35.44 | 39 | 18.93 | 7 | 3.40 | 1 | 0.48 | |||||||||
| JEGX01.CN0002 | 43 | 0 | 0.00 | 0 | 0.00 | 0 | 0.00 | 7 | 16.28 | 4 | 9.30 | 1 | 2.33 | 6 | 13.95 | 22 | 51.16 | 3 | 6.98 | |||||||||
| JEGX01.CN0005 | 37 | 2 | 5.40 | 2 | 5.40 | 6 | 16.22 | 4 | 10.81 | 7 | 18.92 | 3 | 8.11 | 13 | 35.14 | 0 | 0.00 | 0 | 0.00 | |||||||||
| JEGX01.CN0016 | 34 | 0 | 0.00 | 0 | 0.00 | 6 | 17.65 | 4 | 11.76 | 9 | 26.47 | 6 | 17.65 | 7 | 20.59 | 2 | 5.88 | 0 | 0.00 | |||||||||
表2 主要优势带型的地区分布
Table 2 Geographic distribution of predominant PFGE patterns
| 优势带型 | 菌株数 | 不同地区PFGE带型构成比分布(%) | ||||||||||||
| 浙江 | 重庆 | 深圳 | 四川 | 北京 | 福建 | 广东 | 广西 | 河南 | 河北 | 湖北 | 湖南 | 上海 | ||
| JEGX01.CN0003 | 333 | 0.00 | 0.60 | 1.80 | 7.51 | 18.62 | 2.10 | 6.61 | 3.91 | 22.52 | 0.00 | 1.80 | 0.60 | 33.93 |
| JEGX01.CN0001 | 206 | 0.00 | 0.00 | 1.46 | 5.83 | 34.95 | 0.00 | 7.28 | 1.46 | 21.36 | 0.00 | 0.97 | 1.94 | 24.75 |
| JEGX01.CN0002 | 43 | 0.00 | 0.00 | 4.65 | 0.00 | 0.00 | 0.00 | 2.33 | 4.65 | 60.46 | 0.00 | 6.98 | 2.33 | 18.60 |
| JEGX01.CN0005 | 37 | 0.00 | 0.00 | 0.00 | 8.11 | 18.92 | 0.00 | 8.11 | 2.70 | 24.32 | 0.00 | 2.70 | 0.00 | 35.14 |
| JEGX01.CN0016 | 34 | 0.00 | 0.00 | 0.00 | 5.88 | 17.65 | 0.00 | 0.00 | 5.88 | 29.41 | 0.00 | 0.00 | 20.59 | 20.59 |
2.2 肠炎沙门菌PFGE的特征性条带
图2中我们对40种肠炎沙门菌的优势带型,条带大小进行比对,得出肠炎沙门菌优势带型中普遍存在的共同条带,以便在PFGE条带比对时提供参考。分别为(±0.8%)655、325、310、245、178和110 kbp [12]。
2.3 肠炎沙门菌暴发相关带型
暴发相关带型指实验室PFGE带型相同,流行病学调查确定为点源性暴发的菌株,尚未发现跨地域流行。暴发相关的带型主要为JEGX01.CN0003、JEGX01.CN0001和JEGX01.CN0150,分别与三次家庭内聚餐暴发有关。其中JEGX01.CN0003、JEGX01.CN0001是中国肠炎沙门菌的优势带型,处于第1、2位,因此在今后的实验室监测中应加强这几种带型的跨地域流行病学调查,避免大范围暴发发生。
3 讨论
全球化及食品贸易国际化带来的食品分发途径的改变,使食源性疾病发生逐渐由暴发转为多点散发, 单纯血清型的分型鉴定已不能满足在复杂的食源性疾病诊断中对病原体进行溯源的需求。肠炎沙门菌是非伤寒沙门菌性腹泻或食物中毒中最常见的血清型之一,PFGE作为大多数细菌分子分型的金标准在暴发的发现及确认中得到了广泛的关注及应用。通过PFGE图谱分析,并与以往本底资料比较,发现新的聚集性PFGE图谱,确认暴发。因此,沙门菌引起的食源性疾病的暴发在很大程度上依赖于分子分型。分子分型方法区分能力采用Simpsom指数的D值计算方法,了解肠炎沙门菌在我国的流行病学分布规律及PFGE分型特点,对于肠炎沙门菌的监测、暴发预警及调查至关重要。
本次研究的菌株共计981株,年代从2004—2012年。由于中美EID项目子项目6全球食源性疾病监测网络(GFN)工作的开展,2004—2012年,肠炎沙门菌的菌株量呈倍数增长(表1)。尤其是2006年之后,收集到的肠炎沙门菌菌株的数量也明显增加。2012年肠炎沙门菌PFGE结果很多省份还未能及时提交,因此数据量较少。分布在全国13个省(市),从2株到288株不等,主要分布地区有上海、河南、北京、四川、广东等地区,这些省份都是GFN项目的主要参与成员。
优势带型JEGX01.CN0003分布自2004—2012年,集中分布于2008—2010年。JEGX01.CN0001和JEGX01.CN0002带型分布自2006—2012年,在2004年和2005年未见分离到此带型菌株。而JEGX01.CN0005带型出现在2005—2010年,JEGX01.CN0016带型出现自2006—2011年,提示这种带型的病原菌在近两年已不存在。优势带型的地区分布分析,提示在浙江、河北省未分离到该带型菌株。各个带型在不同省份的分布不均,其中以JEGX01.CN0003带型的分布范围最广。优势带型在时间及地区分布的广泛性提示,感染源的持续存在,实验室应加强菌株PFGE分析的及时性,结合流行病学调查,预防可能的跨区域暴发。
图2 肠炎沙门菌PFGE优势带型的共有条带
Figure 2 Common bands of predominant PFGE patterns of S. enteritidis strains 挑选肠炎沙门菌40种优势带型菌株,利用BioNumerics软件中的电泳条带的比对计算功能,将PFGE条带标记大小,通过比对条带值,可以看出,其中6条带为肠炎菌株的共有条带,分别为655、325、310、245、178和110 kbp。如需进一步确认肠炎沙门菌的特异性条带,一方面需较大的样本数量,另一方面,需对比其他血清型菌株,此条带在其他血清型菌株的PFGE图谱中频率较低,才可确认为此血清型的特征性条带[12]。
肠炎沙门菌是非伤寒的沙门菌的主要血清型之一,在我国分布较广,PFGE是肠炎沙门菌监测工作中的重要工具,但对于肠炎沙门菌XbaⅠ单酶切分型能力有限,本次研究中981株肠炎沙门菌分型的D值仅为0.8336,需多种酶切联合使用,提高分型能力,增加暴发发现的概率 。
此外,在PulseNet China的沙门菌的数据库中,肠炎沙门菌共计1044株,本次研究的菌株量为981株,其余的63株由于缺少详细的流行病学调查数据,缺少具体的暴发地点、来源、分离时间,失去进行流行病学及PFGE分型结果分析的意义。沙门菌的监测工作的目的是通过日常的监测数据,实验室的实验结果结合详细的流行病学资料,为暴发的预警及调查工作提供依据,加强实验室与流行病学调查部门的沟通,才能在食源性疾病暴发的早期发现、早期干预方面有所突破,促进疾病的预防控制工作。
参考文献
[1] Scaria J, Palaniappan RU, Chiu D, et al. Microarray for molecular typing of Salmonella enterica serovars[J]. Mol Cell Probes,2 008, 22(4):238-243.
[2] Pang JC, Chiu TH, Chiou CS, et al.Pulsed-field gel electrophoresis, plasmid profiles and phage types for the human isolates of Salmonella enterica serovar Enteritidis obtained over 13 years in Taiwan[J]. J Appl Microbiol, 2005, 99(6): 1472-1483.
[3] de Jong B, Ekdahl K. The comparative burden of salmonellosis in the European Union member states, associated and candidate countries[J]. BMC Public Health, 2006, 6:4.
[4] Boxrud D, Pederson-Gulrud K, Wotton J, et al. Comparison of multiple-locus variable-number tandem repeat analysis, pulsed-field gel electrophoresis, and phage typing for subtype analysis of Salmonella enterica serotype Enteritidis[J]. J Clin Microbiol, 2007, 45(2):536-543.
[5] Beranek A, Mikula C, Rabold P, et al. Multiple-locus variable-number tandem repeat analysis for subtyping of Salmonella enterica subsp enterica serovar Enteritidis[J]. Int J Med Microbiol, 2009, 299(1):43-51.
[6] Chiou CS, Hung CS, Torpdahl M, et al. Development and evaluation of multilocus variable number tandem repeat analysis for fine typing and phylogenetic analysis of Salmonella enterica serovar Typhimurium[J]. Int J Food Microbiol, 2010, 142 (1/2): 67-73.
[7] Torpdahl M, Sorensen G, Lindstedt BA, et al. Tandem repeat analysis for surveillance of human Salmonella typhimurium infections[J]. Emerg Infect Dis, 2007,13(3):388-395.
[8] Lindstedt BA, Heir E, Gjernes E, et al. DNA fingerprinting of Salmonella enterica subsp. enterica serovar typhimurium with emphasis on phage type DT104 based on variable number of tandem repeat loci[J]. J Clin Microbiol, 2003,41(4): 1469-1479.
[9] Garaizar J, Lopez-Molina N, Laconcha I, et al. Suitability of PCR fingerprinting, infrequent-restriction-site PCR, and pulsed-field gel electrophoresis, combined with computerized gel analysis, in library typing of Salmonella enterica serovar enteritidis[J]. Appl Environ Microbiol, 2000, 66(12): 5273-5281.
[10] Gaul SB, Wedel S, Erdman MM, et al. Use of pulsed-field gel electrophoresis of conserved Xba Ⅰ fragments for identification of swine Salmonella serotypes[J]. J Clin Microbiol, 2007, 45(2):472-476.
[11] Liebana E, Guns D, Garcia-Migura L, et al. Molecular typing of Salmonella serotypes prevalent in animals in England: assessment of methodology[J]. J Clin Microbiol, 2001, 39(10):3609-3616.
[12] Gaul SB, Wedel S, Erdman MM, et al. Use of pulsed-field gel electrophoresis of conserved Xba Ⅰ fragments for identification of swine Salmonella serotypes[J]. J Clin Microbiol, 2007, 45(2):472-476.
[13] Hyytia-Trees E, Smole SC, Fields PA, et al. Second generation subtyping: a proposed PulseNet protocol for multiple-locus variable-number tandem repeat analysis of Shiga toxin-producing Escherichia coli O157 (STEC O157) [J]. Foodborne Pathog Dis, 2006,3(1):118-131.
[14] Lukinmaa S, Nakari UM, Eklund M, et al. Application of molecular genetic methods in diagnostics and epidemiology of food-borne bacterial pathogens[J]. APMIS, 2004,112(11/12):908-929.
[15] Swaminathan B, Barrett TJ, Hunter SB, et al. PulseNet: the molecular subtyping network for foodborne bacterial disease surveillance, United States[J]. Emerg Infect Dis, 2001,7(3):382-389.
[16] Zheng J, Keys CE, Zhao S, et al. Simultaneous analysis of multiple enzymes increases accuracy of pulsed-field gel electrophoresis in assigning genetic relationships among homogeneous Salmonella strains[J]. J Clin Microbiol, 2011,49(1):85-94.
[2] Pang JC, Chiu TH, Chiou CS, et al.Pulsed-field gel electrophoresis, plasmid profiles and phage types for the human isolates of Salmonella enterica serovar Enteritidis obtained over 13 years in Taiwan[J]. J Appl Microbiol, 2005, 99(6): 1472-1483.
[3] de Jong B, Ekdahl K. The comparative burden of salmonellosis in the European Union member states, associated and candidate countries[J]. BMC Public Health, 2006, 6:4.
[4] Boxrud D, Pederson-Gulrud K, Wotton J, et al. Comparison of multiple-locus variable-number tandem repeat analysis, pulsed-field gel electrophoresis, and phage typing for subtype analysis of Salmonella enterica serotype Enteritidis[J]. J Clin Microbiol, 2007, 45(2):536-543.
[5] Beranek A, Mikula C, Rabold P, et al. Multiple-locus variable-number tandem repeat analysis for subtyping of Salmonella enterica subsp enterica serovar Enteritidis[J]. Int J Med Microbiol, 2009, 299(1):43-51.
[6] Chiou CS, Hung CS, Torpdahl M, et al. Development and evaluation of multilocus variable number tandem repeat analysis for fine typing and phylogenetic analysis of Salmonella enterica serovar Typhimurium[J]. Int J Food Microbiol, 2010, 142 (1/2): 67-73.
[7] Torpdahl M, Sorensen G, Lindstedt BA, et al. Tandem repeat analysis for surveillance of human Salmonella typhimurium infections[J]. Emerg Infect Dis, 2007,13(3):388-395.
[8] Lindstedt BA, Heir E, Gjernes E, et al. DNA fingerprinting of Salmonella enterica subsp. enterica serovar typhimurium with emphasis on phage type DT104 based on variable number of tandem repeat loci[J]. J Clin Microbiol, 2003,41(4): 1469-1479.
[9] Garaizar J, Lopez-Molina N, Laconcha I, et al. Suitability of PCR fingerprinting, infrequent-restriction-site PCR, and pulsed-field gel electrophoresis, combined with computerized gel analysis, in library typing of Salmonella enterica serovar enteritidis[J]. Appl Environ Microbiol, 2000, 66(12): 5273-5281.
[10] Gaul SB, Wedel S, Erdman MM, et al. Use of pulsed-field gel electrophoresis of conserved Xba Ⅰ fragments for identification of swine Salmonella serotypes[J]. J Clin Microbiol, 2007, 45(2):472-476.
[11] Liebana E, Guns D, Garcia-Migura L, et al. Molecular typing of Salmonella serotypes prevalent in animals in England: assessment of methodology[J]. J Clin Microbiol, 2001, 39(10):3609-3616.
[12] Gaul SB, Wedel S, Erdman MM, et al. Use of pulsed-field gel electrophoresis of conserved Xba Ⅰ fragments for identification of swine Salmonella serotypes[J]. J Clin Microbiol, 2007, 45(2):472-476.
[13] Hyytia-Trees E, Smole SC, Fields PA, et al. Second generation subtyping: a proposed PulseNet protocol for multiple-locus variable-number tandem repeat analysis of Shiga toxin-producing Escherichia coli O157 (STEC O157) [J]. Foodborne Pathog Dis, 2006,3(1):118-131.
[14] Lukinmaa S, Nakari UM, Eklund M, et al. Application of molecular genetic methods in diagnostics and epidemiology of food-borne bacterial pathogens[J]. APMIS, 2004,112(11/12):908-929.
[15] Swaminathan B, Barrett TJ, Hunter SB, et al. PulseNet: the molecular subtyping network for foodborne bacterial disease surveillance, United States[J]. Emerg Infect Dis, 2001,7(3):382-389.
[16] Zheng J, Keys CE, Zhao S, et al. Simultaneous analysis of multiple enzymes increases accuracy of pulsed-field gel electrophoresis in assigning genetic relationships among homogeneous Salmonella strains[J]. J Clin Microbiol, 2011,49(1):85-94.
|
扩展功能
|
|
| 本文信息 | |
| PDF全文 | |
| HTML全文 | |
| 参考文献 | |
| 服务与反馈 | |
| 加入引用管理器 | |
| 引用本文 | |
| Email Alert | |
| 本文作者相关文章 | |
| 娄静 | |
| 刁保卫 | |
| 李杰 | |
| 阚飙 | |
| 闫梅英 | |
| PubMed | |
| Article by LOU Jing | |
| Article by DIAO Bao-wei | |
| Article by LI Jie | |
| Article by KAN Biao | |
| Article by YAN Mei-ying | |