Establishment of rRT-PCR assay for identification of influenza C virus and preparation of reference RNA
LAN Yu, ZHAO Xiang, LI Xiao-dan, WEI He-jiang, WANG Da-yan, SHU Yue-long
China National Influenza Center, Institute for Viral Disease Control and Prevention, Chinese Center for Disease Control and Prevention, WHO Collaborating Center for Reference and Research on Influenza, Beijing 102206, China
Abstract
Objective To establish a real-time quantitative RT-PCR assay to detect influenza C virus and prepare reference RNA of influenza C virus. Methods The sequence of influenza C virus was obtained from GenBank. The specific primers and TaqMan probes targeting conserved regions were designed with Beacon Designer 8 software. The HE, NP, MP and NS gene sequences of the virus were synthesized, then the synthesized genes were cloned into pBluescript Ⅱ SK (+) plasmid respectively. The reference RNA was prepared and the detection limit and specificity of the assay were tested with in vitro transcription of RNA. Results The rapid, reliable real-time quantitative RT-PCR assay for the identification of HE, NP, MP and NS gene of influenza C virus was established and reference RNA was prepared. Conclusion The real time quantitative RT-PCR assay can be used in the routine respiratory virus surveillance and clinical rapid detection of influenza C virus.
丙型流感病毒RNA标准品的制备和实时荧光定量-反转录聚合酶链反应检测方法的建立
蓝雨, 赵翔, 李晓丹, 隗合江, 王大燕, 舒跃龙
中国疾病预防控制中心病毒病预防控制所 国家流感中心 世界卫生组织流感参比和研究合作中心, 北京 102206
摘要
目的 通过分析GenBank中丙型流行性感冒(流感)病毒的序列,建立丙型流感病毒实时荧光定量-反转录聚合酶链反应(rRT-PCR)检测方法,并制备丙型流感病毒的RNA标准品。 方法 下载GenBank中丙型流感病毒的序列,使用Beacon Designer 8软件在保守区域设计特异性引物和TaqMan荧光探针。人工合成病毒HE、NP、MP和NS基因序列,克隆到pBluescript Ⅱ SK(+)质粒载体,进一步通过体外转录获得RNA模板,并验证检测方法的检测极限值和特异性。 结果 建立了针对丙型流感病毒HE、NP、MP和NS基因的快速、可靠的实时荧光定量RT-PCR检测方法,同时制备了RNA标准品。 结论 本文建立的丙型流感病毒实时荧光定量RT-PCR检测方法,可以在日常监测和临床鉴定中快速、准确地检测丙型流感病毒。
内容大纲
-
1 材料与方法
- 1.1 引物、探针的设计及合成
- 1.2 病毒RNA标准品的制备
- 1.2.1 模板的筛选和质粒构建
- 1.2.2 体外转录和标准品的制备
- 1.3 rRT-PCR反应体系的优化
- 1.4 rRT-PCR标准曲线的建立
- 1.5 检测方法的特异性评价
丙型流感病毒可以通过鸡胚或者细胞分离,但分离率比较低,灵敏度不高。已有的普通RT-PCR检测技术容易引起环境污染,检测灵敏度低,也无法对标本中的病毒进行定量检测。为了填补中国目前对丙型流感病毒监测的空白,本文建立了丙型流感病毒rRT-PCR检测方法,以便在日常监测和临床鉴定中快速、准确地检测丙型流感病毒。
1 材料与方法
1.1 引物、探针的设计及合成从GenBank下载所有丙型流感病毒的HE、NP、MP和NS序列,使用ClustalW进行比对,去掉完全一致和较短的序列,最后获得126条HE序列,85条NP序列,96条MP序列,120条NS序列。使用Beacon Designer 8软件设计特异性引物和TaqMan荧光探针,由大连宝生物工程有限公司合成。
1.2 病毒RNA标准品的制备
1.2.1 模板的筛选和质粒构建
由于本实验室没有当前中国大陆人丙型流感病毒的毒株,所以需要人工合成模板。使用1.1步骤的序列分别构建HE、NP、MP和NS四个基因的进化树。为了使模板序列跟中国当前流行的毒株更接近,本研究从进化树上筛选与80年代初中国首次报道的C/pig/Beijing/115/1981毒株处于同一分支,年代最近,而且包含完整开放阅读框(open reading frame, ORF)毒株的序列作为模板序列。
获得的HE、NP、MP和NS基因完整ORF序列由大连宝生物工程有限公司合成,并将合成的DNA模板克隆到含有T7启动子的pBluescript Ⅱ SK(+)质粒上,通过对质粒测序确认合成DNA序列的准确性。
1.2.2 体外转录和标准品的制备
将质粒转化DH5a感受态细胞,用Amp抗性LB平板筛选阳性克隆,挑选阳性克隆,Amp抗性LB培养基37 ℃,200 r/min振荡培养过夜。使用TaKaRa质粒小提试剂盒提取质粒,用限制性内切酶(ScaⅠ和NotⅠ)双酶切质粒。然后用Promega公司的RiboMAXTM Express Large Scale RNA-T7试剂盒进行体外转录,向体外转录产物中加入Dnase酶,37 ℃,15 min,去除DNA模板。再用QIAGEN公司的RNeasy Mini Kit试剂盒纯化RNA,对纯化后的RNA使用Invitrogen公司的Qubit 核酸蛋白定量仪进行RNA定量。
根据核酸分子拷贝数的计算公式:拷贝数=(6.02×1023拷贝/摩尔)×(浓度)/(平均分子量),得出单位体积RNA拷贝数的计算公式(拷贝/ml): 拷贝/ml=6.02×1023×(Xg/ml)/(RNA碱基数×340),把Qubit 核酸蛋白定量仪测得的RNA浓度分别代入上述公式,计算出体外转录RNA模板每微升拷贝数,调整该RNA模板为109拷贝/μl,并继续进行10倍梯度稀释获得100~109拷贝/μl的RNA标准品。
1.3 rRT-PCR反应体系的优化
本研究的反应体系为:①2×RT-PCR buffer 12.5 μl; ②25×RT-PCR Enzyme Mix 1μl;③上游引物、下游引物和探针各0.5 μl ;④检测标本5 μl;⑤RNase Free Water 5 μl,最终的反应体系为25 μl。RT-PCR程序为45 ℃ 10 min;95 ℃ 10 min;95 ℃ 15 s,60 ℃ 45 s,40个循环。将引物和探针的工作浓度分别稀释为10、20、30、40和50 μmol/L,然后使用矩阵法筛选引物和探针的最佳浓度。
1.4 rRT-PCR标准曲线的建立
使用优化好的体系,分别对100~109拷贝/μl的RNA标准品进行荧光定量RT-PCR扩增,利用Stratagene的公司的MxPro软件进行分析,建立标准曲线。
1.5 检测方法的特异性评价
用2009年香港分离到的1株丙型流感病毒(V09-2216483)和其他90份呼吸道病毒或标本,共11种病毒对引物和探针的特异性进行评价。90份其他病毒包括本实验室常规监测得到的乙型流感病毒(N=10)、H3N2和H1N1亚型流感病毒(N=30),以及由多病原检测试剂盒鉴定为阳性的呼吸道标本[3],包括鼻病毒(N=10)、副流感病毒(N=10)、呼吸道合胞病毒A(N=10)和B(N=10)和人偏肺病毒(N=10)。阴性对照和阳性对照分别为EASY Dilution和本研究制备的107拷贝/μl的RNA标准品。
2 结果
2.1 引物和探针特性使用Beacon Designer 8软件在HE、NP、MP和NS的保守区域设计各个片段的特异性引物和探针,探针的5′端用FAM标记,3′端用BHQ1标记,由大连宝生物工程有限公司合成,引物和探针序列及特性见表1。
2.2 病毒RNA标准品的制备
为了验证提取质粒的准确性,并保证体外转录获得等长度的RNA模板,因此对质粒进行ScaⅠ和NotⅠ双酶切,其中ScaⅠ内切酶位于T7启动子的上游,NotⅠ内切酶位于目的片段的下游,酶切电泳图谱见图1。
图1显示HE、NP和MP质粒的双酶切电泳图形成明显双条带,NS质粒形成单一条带(NS质粒双
表1 特异性引物和探针特性
Table 1 Characteristics of primer and probe sets
| 名称 | 长度 | 序列 (5′~3′) | 长度 (bp) | 退火 温度 |
| FluCHE-F105 | HE | YGG AGG AAA TTT RTA TGC | 18 | 59.9 |
| FluCHE-R201 | GCC RAT CCA TGT ACT TTG | 18 | 59.9 | |
| FluCHE-P182 | FAM-TTC AAG ACR GAR GCT CCA GC-BHQ1 | 20 | 67.0 | |
| FluCNP-F201 | NP | RAG GGA AAA GAA AGC AAG | 18 | 59.7 |
| FluCNP-R316 | CTG CRT GGT AGG TTA TAT TG | 20 | 59.6 | |
| FluCNP-P231 | FAM-AGC RGA CAG YAA CTT CAA CGC-BHQ1 | 21 | 69.1 | |
| FluCMP-F984 | MP | GAC CAC AAT TAT GCC TGA A | 19 | 61.7 |
| FluCMP-R1089 | TGG TGA GTT GTC GGT TTC | 18 | 63.7 | |
| FluCMP-P1058 | FAM-TCT CCC AGG TCA AGT CTC TCC C-BHQ1 | 22 | 69.9 | |
| FluCNS-F249 | NS | CGT AGA AAT GAA GGG RAA G | 19 | 59.4 |
| FluCNS-R394 | CTC CAC GAT GAT GAT ACA | 18 | 59.6 | |
| FluCNS-P287 | FAM-CTC CAA GCA ACA TAG CAC CAA TT-BHQ1 | 23 | 66.8 |
图1 质粒经ScaⅠ和NotⅠ双酶切鉴定
Figure 1 Identification of plasmid by ScaⅠ and NotⅠ digestion
酶切后2个片段大小正好相等),而且片段大小也正确,表明提取的质粒是正确的。
酶切后的产物进行体外转录,然后向体外转录产物中加入Dnase酶消化DNA模板,对纯化后的RNA进行定量,再用EASY Dilution 分别对HE、NP、MP和NS的RNA模板进行10倍梯度稀释,获得100~109拷贝/μl的RNA标准品。每个浓度分装6 μl/管(保存20份)作为rRT-PCR的标准品,另外对107拷贝/μl分装50 μl/管作为阳性对照(保存5份),所有RNA产物立即保存于-80 ℃冰箱中。
2.3 反应体系的优化
本研究rRT-PCR反应试剂使用Ambion公司的Ag Path-IDTM One-step RT-PCR试剂盒,通过多批次的重复试验,证明在25 μl反应体系中,当引物和探针的浓度分别为40 μmol/L和20 μmol/L时,上游引物、下游引物和探针各加0.5 μl,最后得到的结果本底噪音最低,Ct值最小,扩增效率最高。
2.4 标准曲线的确定
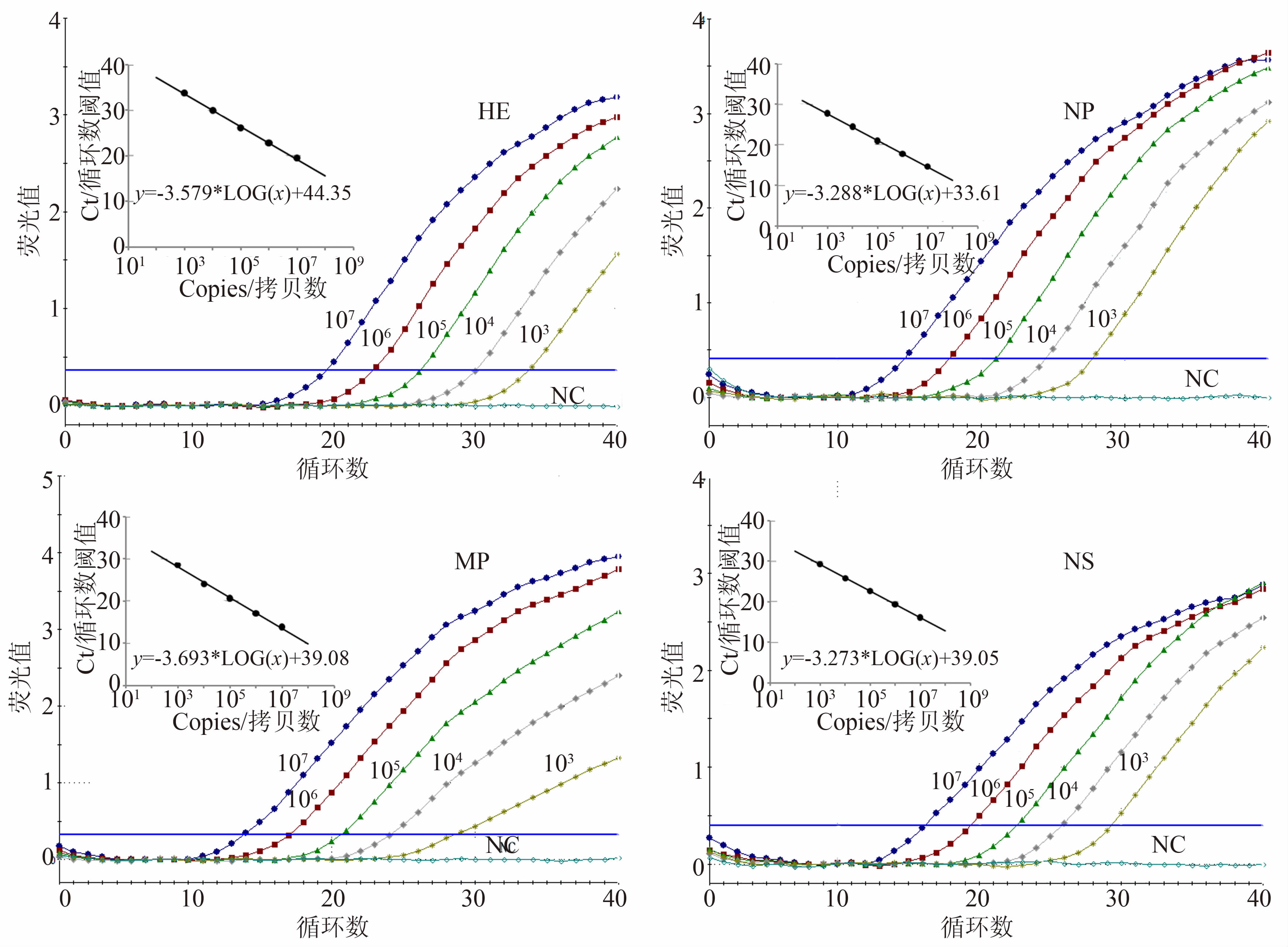
分别取HE、NP、MP和NS的103~107拷贝/μl的RNA模板,以上述优化反应的条件进行RT-PCR反应,采用Stratagene公司随机附带的MxPro软件进行分析,获得定量标准曲线,见图2。重复5批次,获得的RT-PCR扩增曲线的基线均平整,标准曲线的相关系数(r2)都在0.996以上,扩增效率(Eff.)都大于0.883。可见本文RNA标准品的稳定性和标准曲线可靠性很好。
图2 RNA标准品RT-PCR扩增曲线和标准曲线
Figure 2 Amplification plots and Standard curve of reference RNA
2.5 检测极限
为了确定各个引物的检测极限值,分别取HE、NP、MP和NS的100~102拷贝/μl的RNA标准品,以上述优化反应的条件进行RT-PCR反应,重复5批次,得到HE引物/探针的检测极限为500拷贝/反应,NP引物/探针的检测极限为5拷贝/反应,MP引物/探针的检测极限为50拷贝/反应,NS引物/探针的检测极限为50拷贝/反应。
2.6 特异性评价
本方法对香港分离到的丙型流感病毒V09-2216483鉴定为阳性,对其他90份呼吸道病毒和标本均鉴定为阴性,同时阴性对照和阳性对照都成立,表明本研究建立的方法对检测丙型流感病毒具有很高的特异性。
3 讨论
自1947年在美国分离到第1株人丙型流感病毒(C/Taylor/1233/1947)以来,日本、希腊、英国等地都有报道丙型流感病毒的相关研究和文献,特别是日本的某些地区一直持续监测丙型流感病毒的活动情况。但在中国大陆,丙型流感病毒的研究和监测没有像甲型和乙型流感病毒那样得到重视,目前只有1981年和1982年郭元吉等[2]从猪群中分离到2株丙型流感病毒的文献报道。为了掌握丙型流感病毒在中国的流行情况,了解其造成的疾病负担,有必要在中国或者中国部分地区开展丙型流感病毒的监测。
流感病毒是分节段的RNA病毒,与甲型和乙型流感病毒基因组由8个RNA片段构成不同,丙型流感病毒基因组由7个RNA片段组成,分别为:PB2、PB1、P3、HE、NP、MP和NS片段。据文献报道,基因重配并非甲型或者乙型流感病毒的专利,丙型流感病毒的基因重配也时常发生[4],本研究为了最大限度地保证检测引物的正常工作,所以设计了HE、NP、MP和NS四个片段的引物,以防病毒的重配导致漏检,反过来,如果病毒发生重配也能及时被发现。另外,由于中国人群中丙型流感病毒的活动情况缺少监测数据,病毒的抗原性和基因的进化情况也没有掌握,因此本文根据流感病毒进化特点,假定中国人群中当前流行的丙型流感病毒由C/pig/Beijing/115/1981毒株或其类似株进化而来,并且已经进化到与最近分离到的毒株同源,所以分别以4株最近分离到的毒株序列作为人工合成的模板(HE:C/Yamagata/15/2004;NP:C/Yamagata/2/2010;MP:C/Miyagi/5/2000;NS:C/Hiroshima/251/2000),并进而制备了病毒RNA标准品,一方面可用于本研究核酸检测方法的评价,另一方面构建的重组完整ORF序列质粒也可用于蛋白表达等后期相关研究工作。
因为普通方法对丙型流感病毒检测和分离的灵敏度不高,费时又费力,所以本文设计了rRT-PCR检测丙型流感病毒的方法。在提高了检测灵敏度的同时,又节约了时间,也最大限度地避免实验室的污染,为中国开展丙型流感病毒监测和临床鉴定做了技术准备。
( 志谢: 感谢香港公共卫生实验中心罗懿之医生提供丙型流感病毒毒株)
参考文献
[1] Matsuzaki Y,Abiko C,Mizuta K, et al. A nationwide epidemic of influenza C virus infection in Japan in 2004[J]. J Clin Microbiol,2007,45(3):783-788.
[2] Guo YJ, Jin FG, Wang M, et al. Isolated influenza C virus from swine in China[J]. Chinese Science Bulletin, 1982,3:186-188.(in Chinese)
郭元吉,金粉根,王敏, 等. 从我国猪中分离到丙型流感病毒[J]. 科学通报, 1982,3:186-188.
[3] Li J,Mao NY, Zhang C, et al. The development of a GeXP-based multiplex reverse transcription-PCR assay for simultaneous detection of sixteen human respiratory virus types/subtypes[J]. BMC Infect Dis, 2012,12:189.
[4] Peng G,Hongo S,Kimura H, et al. Frequent occurrence of genetic reassortment between influenza C virus strains in nature[J]. J Gen Virol, 1996,77(Pt 7):1489-1492.
[2] Guo YJ, Jin FG, Wang M, et al. Isolated influenza C virus from swine in China[J]. Chinese Science Bulletin, 1982,3:186-188.(in Chinese)
郭元吉,金粉根,王敏, 等. 从我国猪中分离到丙型流感病毒[J]. 科学通报, 1982,3:186-188.
[3] Li J,Mao NY, Zhang C, et al. The development of a GeXP-based multiplex reverse transcription-PCR assay for simultaneous detection of sixteen human respiratory virus types/subtypes[J]. BMC Infect Dis, 2012,12:189.
[4] Peng G,Hongo S,Kimura H, et al. Frequent occurrence of genetic reassortment between influenza C virus strains in nature[J]. J Gen Virol, 1996,77(Pt 7):1489-1492.
|
扩展功能
|
|
| 本文信息 | |
| PDF全文 | |
| HTML全文 | |
| 参考文献 | |
| 服务与反馈 | |
| 加入引用管理器 | |
| 引用本文 | |
| Email Alert | |
| 本文作者相关文章 | |
| 蓝雨 | |
| 赵翔 | |
| 李晓丹 | |
| 隗合江 | |
| 王大燕 | |
| 舒跃龙 | |
| PubMed | |
| Article by LAN Yu | |
| Article by ZHAO Xiang | |
| Article by LI Xiao-dan | |
| Article by WEI He-jiang | |
| Article by WANG Da-yan | |
| Article by SHU Yue-long | |