Relationship between interleukin-18 gene polymorphisms and chronic hepatitis B virus infection
LIAO Yan1, ZHOU Qing-xi1, LIAO Wei-jia2, HE Song-qing2, YUAN Sheng-guang2, QIN Li-ling2, CHEN Qian2, YU Ya-qun2, Mei Ming-hui2
Guilin Center for Disease Control and Prevention, Guilin 541001, Guangxi, China
Abstract
Objective To investigate the relationship between the single nucleotide polymorphisms (SNP)-137G/C (rs187238) and-607C/A (rs1946518) in the promoter region of Interleukin-18 (IL-18) gene and chronic hepatitis B virus infection. Methods Case-control study was conducted among 264 chronic hepatitis B cases and 300 healthy controls seeking medical care in Guilin Municipal Center for Disease Control and Prevention and Affiliated Hospital of Guilin Medical College from March 2007 to October 2010. The SNP at the position of IL-18 gene promoter-137G/C (rs187238) and-607C/A (rs1946518) were detected by sequence specific primer-polymerase chain reaction (SSP-PCR). The genotype and allele frequencies were analyzed for both case group and control group. Results At rs1946518, the difference in the genotype and allele frequencies of IL-18 gene polymorphism between case group and control group was not significant (P>0.05).but,at rs187238, G allele frequency was significantly higher (OR=1.353, 95% CI:1.009-1.815,P=0.043) in case group. Patients who carried rs187238 GG genotype had higher risk of suffering from chronic hepatitis B (OR=1.629,95% CI:1.152-2.305,P=0.006). Stratified analysis showed that rs187238 GC genotypes and C allele frequency were significantly associated in chronic hepatitis B patients who were HBeAg positive (P=0.022, P=0.011, respectively). Conclusion IL-18 gene promoter-137G/C (rs187238) polymorphism is correlated with chronic hepatitis B, and rs187238 GC genotypes and C allele frequency are respectively associated with chronic hepatitis B patients who are HBeAg positive.
白细胞介素-18基因多态性与慢性乙型病毒性肝炎病毒感染关系的研究
廖艳1, 周清喜1, 廖维甲2, 何松青2, 袁晟光2, 覃理灵2, 陈谦2, 喻亚群2, 梅铭惠2
1. 桂林市疾病预防控制中心, 广西 桂林 541001;
2. 桂林医学院附属医院肝胆外科研究室, 桂林医学院肝病防治研究所
2. 桂林医学院附属医院肝胆外科研究室, 桂林医学院肝病防治研究所
摘要
目的 探讨白介素-18(IL-18)基因启动子区-137G/C(rs187238)和-607C/A(rs1946518)单核苷酸多态性(SNP)与慢性乙型病毒性肝炎(乙肝)病毒感染之间的关系。 方法 选取2007年3月至2010年10月期间就诊于桂林市疾病预防控制中心和桂林医学院附属医院的慢性乙肝患者作为病例组,共264例;另外选取同期在上述两个单位进行体检,均无慢性乙肝病史的健康者作为对照组,共300例。采用序列特异性引物-聚合酶链反应技术(SSP-PCR),检测上述二组研究对象IL-18基因启动子-137G/C(rs187238)、-607C/A(rs1946518)单核苷酸多态性位点基因型,分析病例组和对照组基因型和等位基因频率分布。 结果 病例组IL-18基因多态性位点rs1946518的基因型和等位基因的频率与对照组相比差异无统计学意义(P>0.05)。但是,病例组IL-18基因SNP位点rs187238 G等位基因的频率高于对照组(OR=1.353,95%CI:1.009~1.815,P=0.043)。携带rs187238 GG基因型的患者发生慢性乙肝的风险较高(OR=1.629,95%CI:1.152~2.305,P=0.006)。分层分析发现,rs187238位点上GC基因型和C等位基因的频率与慢性乙肝的关联在乙肝e抗原(HBeAg)阳性组的患者中更加显著(P=0.022,P=0.011)。 结论 IL-18基因启动子区-137G/C(rs187238)多态性与慢性乙肝易感性具有相关性,而且,rs187238位点上GC基因型和C等位基因的频率与HBeAg阳性的慢性乙肝患者具有相关性。
内容大纲
-
1 材料与方法
- 1.1 研究对象
- 1.2 基因组DNA提取
- 1.3 PCR引物设计与合成
- 1.4 PCR反应条件
- 1.5 统计学分析
- 2.1 病例组和对照组的基本情况
- 2.2 SSP-PCR方法检测
- 2.3 IL-18基因SNP位点等位基因在病例组与对照组的分布频率
- 2.4 IL-18基因SNP位点基因型与慢性乙肝易感性的关系
- 2.5 慢性乙肝患者IL-18基因启动子区位点基因型和等位基因分布频率在HBeAg阴性组和阳性组的关系
近年研究表明,单核苷酸多态性(single nucleotide polymorphism, SNP)的研究对人类疾病的预防、病因探寻、诊断和治疗等各方面具有重大的意义 。
包括白介素-18(interleukin-18, IL-18)在内的许多细胞因子SNP与HBV清除有关,影响着HBV感染后不同的临床结局 。IL-18基因启动子区有多个SNP位点,其中,-137G/C(rs187238)位点在组蛋白H4转录因子结合区,而-607C/A(rs1946518)位点在3′,5′-环磷酸腺苷反应元件结合蛋白区,这两个位点是IL-18发挥细胞调节功能的重要区域[6]。基于此,本实验选择IL-18基因启动子区-137G/C和-607C/A的两个SNP位点,并采用序列特异性引物-聚合酶链反应技术(SSP-PCR)进行分析,探讨广西桂北地区汉族人群IL-18基因SNP与慢性HBV感染预后的关联,并分析其与乙肝e抗原(HBeAg)的关系,期待为慢性HBV感染的防治提供理论依据。
1 材料与方法
1.1 研究对象 随机选取的外周血样本包括:慢性乙肝组(病例组)共264例,均来源于2007年3月至2010年10月期间就诊于桂林市疾病预防控制中心(CDC)和桂林医学院附属医院的患者;另外选取同期在上述两个单位进行体检的300名健康者(对照组),均无乙肝病史。所有研究对象均为汉族人群,对年龄和性别没有特别限制。本研究的实施通过了上述两个单位的医学伦理委员会批准,所取样本都是在患者或健康者签署了书面知情同意书之后收集的。
1.2 基因组DNA提取
所有观察对象均采集静脉血2 ml,经枸橼酸钠抗凝,以蛋白酶K消化-饱和氯化钠盐析法抽提外周血白细胞DNA,并经1.5%琼脂糖凝胶电泳检测DNA的完整性和纯度,放置-80 ℃冰箱保存备用。
1.3 PCR引物设计与合成
依据IL-18基因启动子-137G/C(rs187238)和-607C/A(rs1946518)位点多态性和已知的DNA序列,PCR引物设计参照文献[6](表1),委托上海生工生物技术有限公司合成。其中特异性引物的3′端碱基依据多态性序列与其严格互补原则,通过特定的PCR反应体系扩增各等位基因的特异性DNA片段,产生相对应的特异性扩增条带;设计的内参照引物扩增包含有特异性突变位点的DNA片段,用于判定整个PCR体系的成功与否。
表1 SSP-PCR法分析IL-18基因启动子多态性的引物及扩增产物长度
Table 1 Primer sequences and product sizes of SSP-PCR for polymorphism of IL-18 gene promoter region
| SNP位点 | 引物序列5′ ~3′ | 特异性产物 大小(bp) | 内参照产物 大小(bp) |
| rs187238 | |||
| Specific F1 | CCC CAA CTT TTA CGG AAG AAA AG | 261 | 446 |
| Specific F2 | CCC CAA CTT TTA CGG AAG AAA AC | ||
| Control F | CCA ATA GGA CTG ATT ATT CCG CA | ||
| Common R | AGG AGG GCA AAA TGC ACT GG | ||
| rs1946518 | |||
| Specific F1 | GTT GCA GAA AGT GTA AAA ATT ATT AC | 196 | 301 |
| Specific F2 | GTT GCA GAA AGT GTA AAA ATT ATT AA | ||
| Control F | CTT TGC TAT CAT TCC AGG AA | ||
| Common R | TAA CCT CAT TCA GGA CTT CC |
1.4 PCR反应条件
PCR反应体系总体积 20 μl,包括 2 μl基因组DNA(约20 ng),2 μl 10×PCR buffer,0.5 μl dNTPs,通用反向引物和特异性正向引物1(或2)各 0.15 μl,质控正向引物0.15 μl,0.5 μl Taq DNA聚合酶,使用灭菌超纯水补充至20 μl体系,试剂均为TaKaRa公司产品。PCR反应条件依次是:94 ℃预变性4 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸40 s,10个循环;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸 40 s,25个循环;最后 72 ℃延伸7 min。以上反应均在Applied Biosystem 公司基因扩增仪上完成。PCR产物经2%的琼脂糖凝胶中电泳,紫外线凝胶成像系统观察结果并鉴定基因型。
1.5 统计学分析
应用χ2检验比较基因型在不同分组中的分布差异,用比值比(OR)及其95%的可信区间(95%CI)表示相对危险度,数据均用SPSS 13.0软件进行统计分析,以P<0.05为差异具有统计学意义。
2 结果
2.1 病例组和对照组的基本情况 两组在年龄、性别、饮酒史和吸烟史等分布差异均无统计学意义(P>0.05),表明选取两组样本的数据比较均衡,见表2。
表2 病例组和对照组的基本情况
Table 2 Basic information of case group and control group
| 变量 | 病例组 | 对照组 | |||
| 例数 | 构成比/% | 例数 | 构成比/% | ||
| 年龄组(岁) | |||||
| <55 | 178 | 67.42 | 191 | 63.67 | |
| ≥55 | 86 | 32.58 | 109 | 36.33 | |
| 性别 | |||||
| 男 | 201 | 76.14 | 221 | 73.67 | |
| 女 | 63 | 23.86 | 79 | 26.33 | |
| 饮酒史 | |||||
| 否 | 198 | 75.00 | 237 | 79.00 | |
| 是 | 66 | 25.00 | 63 | 21.00 | |
| 吸烟史 | |||||
| 否 | 241 | 91.29 | 274 | 91.33 | |
| 是 | 23 | 8.71 | 26 | 8.67 | |
2.2 SSP-PCR方法检测
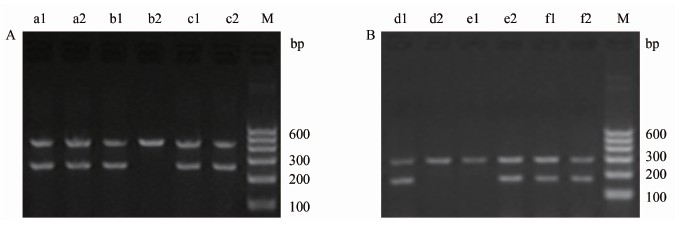
IL-18基因启动子区-137G/C(rs187238)、-607C/A(rs1946518)位点多态性,基因分型见图1。
图1 IL-18基因多态性基因分型
Figure 1 Genotyping for the interleukin-18 gene polymorphisms
nbsp; 注:A泳道a、b、c分别为rs187238基因型GG、GC、CC;B泳道d、e、f分别为rs1946518基因型CC、AA、CA;M为DNA分子质量Marker。
2.3 IL-18基因SNP位点等位基因在病例组与对照组的分布频率
如表3所示,IL-18基因启动子区-137G/C(rs187238)位点的等位基因G和C分布频率在病例组分别为82.2%和17.80%,对照组分别为77.33%和22.67%,G和C等位基因频率在病例组和对照组的分布差异有统计学意义(P=0.043),携带rs187238位点等位基因G的个体发生慢性乙肝的风险高于携带rs187238 位点等位基因 C的个体(OR=1.353,95%CI:1.009~1.815);-607C/A(rs1946518) 位点的等位基因C和A分布频率在两组间基本一致(51.14%,53.67%;48.86%,46.33%),C和A等位基因频率在病例组和对照组的分布差异无统计学意义(P=0.396)。提示,rs187238位点的G等位基因可能是慢性乙肝的遗传易感基因。
2.4 IL-18基因SNP位点基因型与慢性乙肝易感性的关系
IL-18基因rs187238和rs1946518位点的各基因型在病例组和对照组中的分布情况见表4。IL-18基因 SNP rs1946518位点的各基因型在
表3 IL-18基因SNP位点等位基因在病例组与对照组中频率分布比较
Table 3 Allele frequencies of IL-18 promoter gene in case group and control group
| IL-18等位基因 | 病例组 | 对照组 | |||
| 例数(N=264) | 构成比/% | 例数(N=300) | 构成比/% | ||
| rs187238 | |||||
| G | 434 | 82.20 | 464 | 77.33 | |
| C | 94 | 17.80 | 136 | 22.67 | |
| rs1946518 | |||||
| C | 270 | 51.14 | 322 | 53.67 | |
| A | 258 | 48.86 | 278 | 46.33 | |
两组中的分布差异无统计学意义(P=0.183);而rs187238位点的各基因型在两组中的分布差异有统计学意义(P=0.006),病例组位点GG、GC和CC基因型分布频率分别为68.94%、25.76%和5.30%, 对照组三种基因型的分布频率分别为57.67%、38.33%和4.00%。携带rs187238 GG基因型的个体发生慢性乙肝的风险高于携带rs187238 GC/CC基因型的个体(OR=1.629,95%CI: 1.152~2.305)。结果提示,
表4 IL-18基因多态性基因型在病例组和对照组分布
Table 4 Genotype distribution of IL-18 gene polymorphisms in case group and control group
| 组别 | rs187238基因型频率 | rs1946518基因型频率 | |||||||||||||||
| GG | GC | CC | CC | CA | AA | ||||||||||||
| 例数 | 构成比/% | 例数 | 构成比/% | 例数 | 构成比/% | 例数 | 构成比/% | 例数 | 构成比/% | 例数 | 构成比/% | ||||||
| 病例组 | 182 | 68.94 | 68 | 25.76 | 14 | 5.30 | 63 | 23.86 | 145 | 54.92 | 56 | 21.21 | |||||
| 对照组 | 173 | 57.67 | 115 | 38.33 | 12 | 4.00 | 61 | 20.33 | 156 | 52.00 | 83 | 27.67 | |||||
慢性乙肝易感性与IL-18基因启动子区-137G/C(rs187238)位点多态性存在关联。
2.5 慢性乙肝患者IL-18基因启动子区位点基因型和等位基因分布频率在HBeAg阴性组和阳性组的关系
本实验结合病例组中HBeAg阴性和阳性情况,对rs1946518和rs187238位点各基因型的效应进行分析。结果发现,IL-18基因rs187238位点基因型和等位基因分布频率在HBeAg阴性组和阳性组差异都有统计学意义(χ2=7.645, P=0.022;χ2=6.503,P=0.011),而且rs187238位点上GC基因型与慢性乙肝的关联在HBeAg阳性患者中差异有统计学意义(χ2=7.645,P=0.022);rs1946518位点基因型和等位基因分布频率在HBeAg阴性组和阳性组差异均无统计学意义(χ2=1.981,P=0.371;χ2=1.213,P=0.271),见表5。
表5 IL-18基因多态性基因频率分布与HBeAg阴性和阳性的关系
Table 5 Gene frequency distribution of IL-18 gene polymorphisms in HBeAg-negative and positive patients
| 特征 | HBeAg阴性组 | HBeAg阳性组 | |||
| 例数(N=186) | 构成比/% | 例数(N=78) | 构成比/% | ||
| rs187238 | |||||
| 基因型 | |||||
| GG | 137 | 73.66 | 45 | 57.69 | |
| GC | 39 | 20.97 | 29 | 37.18 | |
| CC | 10 | 5.37 | 4 | 5.13 | |
| 等位基因 | |||||
| G | 316 | 84.95 | 118 | 75.64 | |
| C | 56 | 15.05 | 38 | 24.36 | |
| rs1946518 | |||||
| 基因型 | |||||
| CC | 40 | 21.50 | 23 | 29.49 | |
| CA | 106 | 57.00 | 39 | 50.00 | |
| AA | 40 | 21.50 | 16 | 20.51 | |
| 等位基因 | |||||
| C | 196 | 52.69 | 74 | 47.44 | |
| A | 176 | 47.31 | 82 | 52.56 | |
3 讨论
HBV感染不同患者会造成不同的临床转归,其HBV感染慢性化进程的可能机制是Th1/Th2等细胞失衡。而维持Th1/Th2等细胞之间的平衡需要肝脏组织免疫微环境中的细胞因子来调节。也就是说,如能维持良好的平衡关系,HBV感染者,HBV可能自然被清除,反之亦然。总之,Th1/Th2等细胞失衡是因为其调节因子失衡所造成。这类调节因子的失衡与否存在着个体差异,比如某些细胞因子,特别是重要的细胞因子(如IL-18)发生突变,或其基因型不同。IL-18属于Th1类细胞因子,是一种参与机体强有力的细胞免疫调节因子,又名干扰素-γ诱导因子(IGIF)[7],主要是由巨噬细胞、单核细胞、Kuffer细胞和树突细胞等分泌[8]。已有研究证实,IL-18能诱导Th1、NK等细胞增殖和促进Th1类细胞产生IL-2、GMCSF等细胞因子[9],并能增强Th1、NK细胞表达Fas配体以及介导细胞毒反应 ,在机体免疫调节、抗细胞内病毒感染等方面具有广阔的应用前景[7]。慢性乙肝患者病情程度与血清IL-18水平密切相关[12]。提示,IL-18基因可能是慢性乙肝有效的治疗靶标,也可能与其易感性有关。
本实验通过分析IL-18基因启动子区-137G/C(rs187238)和-607C/A(rs1946518)位点的各基因型在病例组和对照组中的分布频率,发现rs1946518位点的各等位基因频率分布和基因型比例在病例组和对照组间其差异无统计学意义(P>0.05)。提示,在慢性乙肝患者和正常人群之间存在相同的遗传背景,rs1946518位点的SNP与慢性乙肝的易感性之间没有必然的联系。而rs187238位点的等位基因(G、C)频率分布和基因型(GG、GC和CC)比例在病例组和对照组间差异有统计学意义(P<0.05)。提示,IL-18基因rs187238位点基因多态性与慢性乙肝易感性相关联,其中,G等位基因可能是慢性乙肝的遗传易感基因;慢性乙肝患者更多的携带rs187238 GG基因型,推测携带rs187238 GG基因型的个体发生慢性乙肝的风险高于携带rs187238 GC/CC基因型的个体;也可能是携带rs187238 GG基因型的HBV感染个体,其HBV不容易被自然清除。
本实验将HBV感染的慢性乙肝者按HBeAg阴性组和阳性组进行比较和分析。结果显示,IL-18基因启动子-137G/C(rs187238)位点基因型和等位基因分布频率在HBeAg阴性组和阳性组差异都有统计学意义(P<0.05),HBeAg阳性是HBV复制活跃的表现之一,推测携带rs187238 GC基因型的慢性乙肝患者的HBV病毒载量较高,HBV复制活跃程度高,传染性强;而-607C/A(rs1946518)位点基因型和等位基因分布频率在HBeAg阴性组和阳性组差异均无统计学意义(P>0.05)。Hirankarn等[12]发现IL-18基因-607位点AA基因型在泰国人群HBV感染组出现频率明显高于健康对照组,推测该位点AA基因型可作为泰国人群中预测HBV感染的指标;Li等[13]发现rs1946518的C等位基因频率在慢性乙肝组与对照组相比差异有统计学意义(P=0.017),与本项研究结果存在差异,可能与不同地区分布有关。而Migita等[14]发现rs1946518位点上AA基因型和rs187238位点上C等位基因频率与HBV复制能力有关联;也有研究者发现IL-18基因启动子区的其他位点,-148位点C等位基因,+8925位点G等位基因和+13925位点C等位基因等与HBV易感性相关[15]。鉴于所选取有限的样本量及不同地区的人群,结果存在差异,尚需要进一步增加样本量进行研究,并验证结果。
慢性乙肝是受多因素共同作用的长期过程,其中HBV与机体环境,尤其是肝脏免疫微环境的相互作用非常关键。我们初步探索了IL-18基因启动子区多态性与慢性乙肝易感性之间的关系,发现rs187238位点基因型与慢性乙肝易感性存在关联性;rs187238位点上GC基因型和C等位基因的频率与HBeAg阳性的慢性乙肝的患者具有相关性,但仍需扩大样本量进行深入研究。期待进一步详细的研究将为阐明慢性乙肝遗传易感性的关联机制提供可能,为临床防治慢性乙肝提供新的、合理的策略。
参考文献
[1] Zhang H, Zhai Y, Hu Z, et al. Genome-wide association study identifies 1p36.22 as a new susceptibility locus for hepatocellular carcinoma in chronic hepatitis B virus carriers [J].Nat Genet,2010,42(9):755-758.
[2] Jiang DK, Sun J, Cao G, et al. Genetic variants in STAT4 and HLA-DQ genes confer risk of hepatitis B virus-related hepatocellular carcinoma [J].Nat Genet,2013,45(1):72-75.
[3] Zhang PA, Wu JM, Li Y, et al. Association of polymorphisms of interleukin-18 gene promoter region with chronic hepatitis B in Chinese Han population [J].World J Gastroenterol,2005,11(11):1594-1598.
[4] Hu L, Zhai X, Liu J, et al. Genetic variants in human leukocyte antigen/DP-DQ influence both hepatitis B virus clearance and hepatocellular carcinoma development [J].Hepatology,2012,55(5):1426-1431.
[5] Li Y, Shi Y, Chen J, et al. Association of polymorphisms in interleukin-18 and interleukin-28B with hepatitis B recurrence after liver transplantation in Chinese Han population [J].Int J Immunogenet,2012,39(4):346-352.
[6] Giedraitis V, He B, Huang WX, et al. Cloning and mutation analysis of the human IL-18 promoter:a possible role of polymorphisms in expression regulation [J].J Neuroimmunol,2001,112(1-2):146-152.
[7] Sun Y, Chen HY, Xin SJ. Effect of IL-18 on peripheral blood mononuclear cells of chronic hepatitis B and hepatitis B virus DNA released by HepG2.2.15 cell lines [J].Hepatobil Pancreat Dis Int,2004,3(2):230-234.
[8] Chaudhry UI, Kingham TP, Plitas G, et al. Combined stimulation with interleukin-18 and CpG induces murine natural killer dendritic cells to produce IFN-gamma and inhibit tumor growth [J].Cancer Res,2006,66(21):10497-10504.
[9] Lian H, Jin N, Li X, et al. Induction of an effective anti-tumor immune response and tumor regression by combined administration of IL-18 and Apoptin [J].Cancer Immunol Immunoth,2007,56(2):181-192.
[10] Subleski JJ, Hall VL, Back TC, et al. Enhanced antitumor response by divergent modulation of natural killer and natural killer T cells in the liver[J].Cancer Res,2006,66(22):11005-11012.
[11] Yokoyama A, Mori S, Takahashi HK, et al. Effect of amodiaquine, a histamine N-methyltransferase inhibitor, on, Propionibacterium acnes and lipopolysaccharide-induced hepatitis in mice [J].Eur J Pharmacol,2007,558(1-3):179-184.
[12] Hirankarn N, Manonom C, Tangkijvanich P. Association of interleukin-18 gene polymorphism(-607A/A genotype) with susceptibility to chronic hepatitis B virus infection [J].Tissue Antigens,2007,70(2):160-163.
[13] Li N, Gao YF, Zhang TC, et al. Relationship between interleukin 18 polymorphisms and susceptibility to chronic hepatitis B virus infection [J].World J Hepatol,2012,4(3):105-109.
[14] Migita K, Sawakami-Kobayashi K, Maeda Y, et al. Interleukin-18 promoter polymorphisms and the disease progression of hepatitis B virus-related liver disease [J].Transl Res,2009,153(2):91-96.
[15] Cheong JY, Cho SW, Oh B, et al. Association of interleukin-18 gene polymorphisms with hepatitis B virus clearance [J].Dig Dis Sci,2010,55(4):1113-1119.
[2] Jiang DK, Sun J, Cao G, et al. Genetic variants in STAT4 and HLA-DQ genes confer risk of hepatitis B virus-related hepatocellular carcinoma [J].Nat Genet,2013,45(1):72-75.
[3] Zhang PA, Wu JM, Li Y, et al. Association of polymorphisms of interleukin-18 gene promoter region with chronic hepatitis B in Chinese Han population [J].World J Gastroenterol,2005,11(11):1594-1598.
[4] Hu L, Zhai X, Liu J, et al. Genetic variants in human leukocyte antigen/DP-DQ influence both hepatitis B virus clearance and hepatocellular carcinoma development [J].Hepatology,2012,55(5):1426-1431.
[5] Li Y, Shi Y, Chen J, et al. Association of polymorphisms in interleukin-18 and interleukin-28B with hepatitis B recurrence after liver transplantation in Chinese Han population [J].Int J Immunogenet,2012,39(4):346-352.
[6] Giedraitis V, He B, Huang WX, et al. Cloning and mutation analysis of the human IL-18 promoter:a possible role of polymorphisms in expression regulation [J].J Neuroimmunol,2001,112(1-2):146-152.
[7] Sun Y, Chen HY, Xin SJ. Effect of IL-18 on peripheral blood mononuclear cells of chronic hepatitis B and hepatitis B virus DNA released by HepG2.2.15 cell lines [J].Hepatobil Pancreat Dis Int,2004,3(2):230-234.
[8] Chaudhry UI, Kingham TP, Plitas G, et al. Combined stimulation with interleukin-18 and CpG induces murine natural killer dendritic cells to produce IFN-gamma and inhibit tumor growth [J].Cancer Res,2006,66(21):10497-10504.
[9] Lian H, Jin N, Li X, et al. Induction of an effective anti-tumor immune response and tumor regression by combined administration of IL-18 and Apoptin [J].Cancer Immunol Immunoth,2007,56(2):181-192.
[10] Subleski JJ, Hall VL, Back TC, et al. Enhanced antitumor response by divergent modulation of natural killer and natural killer T cells in the liver[J].Cancer Res,2006,66(22):11005-11012.
[11] Yokoyama A, Mori S, Takahashi HK, et al. Effect of amodiaquine, a histamine N-methyltransferase inhibitor, on, Propionibacterium acnes and lipopolysaccharide-induced hepatitis in mice [J].Eur J Pharmacol,2007,558(1-3):179-184.
[12] Hirankarn N, Manonom C, Tangkijvanich P. Association of interleukin-18 gene polymorphism(-607A/A genotype) with susceptibility to chronic hepatitis B virus infection [J].Tissue Antigens,2007,70(2):160-163.
[13] Li N, Gao YF, Zhang TC, et al. Relationship between interleukin 18 polymorphisms and susceptibility to chronic hepatitis B virus infection [J].World J Hepatol,2012,4(3):105-109.
[14] Migita K, Sawakami-Kobayashi K, Maeda Y, et al. Interleukin-18 promoter polymorphisms and the disease progression of hepatitis B virus-related liver disease [J].Transl Res,2009,153(2):91-96.
[15] Cheong JY, Cho SW, Oh B, et al. Association of interleukin-18 gene polymorphisms with hepatitis B virus clearance [J].Dig Dis Sci,2010,55(4):1113-1119.