1. Department of Clinical Laboratory, The First Affiliated Hospital of Wenzhou Medical University, Wenzhou 325000, Zhejiang China;
2. Department of Respiratory Medicine, The First Affiliated Hospital of Wenzhou Medical University, Wenzhou 325000, Zhejiang, China;
3. Department of Clinical Laboratory, The First Hospital of Jiaxing, Jiaxing 314000, Zhejiang, China
Detection of plasmid-mediated quinolone resistance determinants in clinical non-fermentative bacteria and ciprofloxacin sensitive Enterobacteriaceae strains
ZHANG Xue-qing1, LOU Dan-ping2, XU Chun-quan1, MAO Min-jie3, CHEN Shu-ying1, LIN Chun-chan1, YU Fang-you1, WANG Liang-xing2
Abstract
Objective To investigate the prevalence of three types of plasmid-mediated quinolone resistance (PMQR) determinants in clinical non-fermentative bacteria and ciprofloxacin. sensitive Enterobacteriaceae strains Methods Totally 600 clinical strains isolated from January 2008 to June 2013 were used in this study, including 419 non-fermentative bacteria strains and 181 Enterobacteriaceae strains sensitive to ciprofloxacin. The non-fermentative bacteria strains consisted of 130 strains of Acinetobacter baumannii complex, 100 strains of Pseudomonas aeruginosa, 80 strains of Burkholderia cepacia, 60 strains of Stenotrophomonas maltophilia, 25 strains of Elizabethkingia meningoseptica, 16 strains of Acinetobacter lwoffii and 8 strains of Chryseobacterium gleum. The Enterobacteriaceae strainss sensitive to ciprofloxacin included 69 strains of Enterobacter cloacae complex, 62 strains of Escherichia coli, 35 strains of Klebsiella pneumoniae and 15 strains of Citrobacter freundii. The qnrA,qnrB,qnrS,aac(6')-Ib and qepA genes were detected by PCR, and qnr gene type and aac(6')-Ib-cr were identified by DNA sequencing. Conjugation experiment was conducted to study the transmission of antimicrobial resistance. Results The qnr,aac(6')-Ib and qepA genes were not identified in 419 non-fermentative bacteria strains. The qepA gene was not identified in 181 Enterobacteriaceae strains. Four qnr-positive strains were detected among 181 Enterobacteriaceae strains, including 2 qnrA1-positive strains (1 Klebsiella pneumoniae strain and 1 Escherichia coli strain) and 2 qnrB4-positive strains (2 Enterobacter cloacae complex strains). 12 strains among 181 Enterobacteriaceae strains harbored aac(6')-Ib-cr gene, including 6 Escherichia coli strains, 3 Enterobacter cloacae complex strains and 3 Klebsiella strains. Conjugation experiments demonstrated that the antimicrobial determinants could be transferred to the recipient strains in 7 of 16 strains harboring qnr or aac(6')-Ib-cr gene. Conclusion In Enterobacteriaceae strains, the PMQR genes existed not only in clinical strains resistant to ciprofloxacin,but also in clinical strains sensitive to ciprofloxacin. The PMQR genes might not exist in non-fermentative bacteria stains in Wenzhou.
Keywords:
ciprofloxacin
Enterobacteriaceae
non-fermentative bacteria
qnr genes
aac(6’)-Ib-cr gene
qepA gene
非发酵菌和环丙沙星敏感肠杆菌科细菌中质粒介导喹诺酮类耐药基因的检测
张雪青1, 楼丹萍2, 徐春泉1, 毛敏婕3, 陈舒影1, 林纯婵1, 余方友1, 王良兴2
1. 温州医科大学附属第一医院实验诊断中心,浙江 温州 325000;
2. 温州医科大学附属第一医院呼吸内科,浙江 温州 325000;
3. 嘉兴第一医院检验科,浙江 嘉兴 314000
2. 温州医科大学附属第一医院呼吸内科,浙江 温州 325000;
3. 嘉兴第一医院检验科,浙江 嘉兴 314000
收稿日期:2013-10-31
通讯作者:王良兴,Tel:0577-88069268,Email:38805@163.com
摘要
目的 了解3类质粒介导的喹诺酮类耐药基因在浙江省温州地区分离的非发酵菌和环丙沙星敏感肠杆菌科细菌中的分布情况。方法 收集2008年1月至2013年6月温州地区分离的非发酵菌和环丙沙星敏感肠杆菌科细菌,共600株,其中非发酵菌419株,环丙沙星敏感肠杆菌科细菌181株。采用聚合酶链反应(PCR)检测qnrA、qnrB、qnrS、aac(6)-Ib以及qepA 基因,并通过DNA测序确定qnr基因型和aac(6’)-Ib-cr基因变异体;通过接合转移实验研究细菌质粒介导的耐药性传递情况。结果 419株非发酵菌临床株中未检测到qnrA、qnrB、qnrS、aac(6’)-Ib和qepA等耐药基因。181株环丙沙星敏感的肠杆菌科细菌临床株中未检到qepA,检到2株qnrA1阳性株,分别为1株肺炎克雷伯菌和1株大肠埃希菌;2株qnrB4阳性株,均为阴沟肠杆菌复合菌;12株检测到aac(6’)-Ib,分别为6株大肠埃希菌、3株阴沟肠杆菌复合菌和3株克雷伯菌属细菌。接合转移试验中,2株qnrA1阳性株和1株qnrB4阳性株接合转移成功,12株携带aac(6’)-Ib-cr基因的阳性株中4株接合转移成功。结论 温州地区,质粒介导的喹诺酮类耐药基因不仅存在于环丙沙星耐药株中,而且还存在于环丙沙星敏感的肠杆菌科细菌中,可能不存在于非发酵菌中。
内容大纲
-
1 材料与方法
- 1.1 材料
- 1.2 方法
- 1.2.1 菌株鉴定和药敏试验
- 1.2.2 超广谱β-内酰胺酶(Extended Spectrum Beta-Lactamases,ESBLs)检测
- 1.2.3 聚合酶链反应(PCR)模板的制备
- 1.2.4 PCR
- 1.2.5 DNA序列分析
- 1.2.6 微量肉汤稀释法
- 1.2.7 质粒接合转移试验
- 2.1 药敏结果
- 2.2 qnr基因的检测结果
- 2.3 aac(6′)-Ib的检测结果
- 2.4 qepA的检测结果
- 2.5 ESBLs检测
- 2.6 质粒接合转移试验
1 材料与方法
1.1 材料 1.1.1 菌株来源
收集2008年1月至2013年6月间温州地区临床分离的600株非发酵菌和环丙沙星敏感肠杆菌科细菌,其中非发酵菌419株,包括130株鲍曼不动杆菌复合群、100株铜绿假单胞菌、80株洋葱假单胞菌、60株嗜麦芽寡养假单胞菌、25株脑膜败血伊丽莎白金菌、16株鲁氏不动杆菌和8株粘金黄杆菌;181株环丙沙星敏感肠杆菌科细菌包括69株阴沟肠杆菌复合菌、62株大肠埃希菌、35株肺炎克雷伯菌和15株弗氏柠檬酸杆菌。菌株主要分离自尿液、痰液、脓液、血液、胆汁、引流液、胸水和腹水等临床标本,所有细菌均选择患者的初次分离株,剔除重复菌株。质控菌株包括大肠埃希菌ATCC25922、肺炎克雷伯菌ATCC700603(产SHV-12型ESBLs);qnrA基因阳性对照株为含质粒pMG252的菌株UABl,qnrB基因、qnrS基因以及aac(6′)-Ib-cr基因阳性对照株分别为含质粒pHS7、pHS8及pHS6的菌株J53,由复旦大学附属华山医院抗生素研究所王明贵教授惠赠。接合转移实验受体菌为利福平抗性大肠埃希菌E.coli 600(Nalr,Rifr,LacZ-),由浙江大学医学院附属第一医院俞云松教授惠赠。
1.1.2 抗生素纸片和抗生素标准品
环丙沙星药敏纸片购自OXOID公司,而头孢噻肟(30 μg)、头孢噻肟/克拉维酸(30 μg/10 μg)、头孢他啶(30 μg)和头孢他啶/克拉维酸(30 μg/10 μg)药敏纸片购自北京天坛药物生物技术开发公司。利福平、氯霉素、四环素、环丙沙星、左旋氧氟沙星、庆大霉素、阿米卡星等抗生素标准品购自中国药品生物制品检定所。
1.1.3 培养基
LB李氏增菌肉汤、MH水解酪蛋白琼脂、MH水解酪蛋白肉汤和TSA胰酶胨大豆酵母浸膏琼脂购自杭州天和微生物试剂有限公司。
1.1.4 基因检测试剂
Taq DNA聚合酶购自MBI生物工程有限公司,PCR相关材料和Goldview核酸染料购自上海桑尼生物工程有限公司,DNA Ladder Marker购自大连宝生物工程有限公司。
1.1.5 主要仪器
VITEK-compact型全自动微生物分析仪(法国生物梅里埃公司产品),电子分析天平(瑞士METTLER Toledo公司, 型号AE200),小型高速离心机(德国SIGMA3k18),旋涡混合器(上海医科大学仪器厂,型号XW-80A),多用振荡器 (江苏姜堰市新康仪器厂,型号XK95-I),PCR扩增仪(美国ABI公司,型号ABI Therml cycler 2720),琼脂糖凝胶电泳仪(上海医用分析仪器厂),凝胶成像系统 (Pharamacin Biotech公司,型号Bisoens SC620)测序仪(美国ABI公司,型号3730XL)。
1.1.6 引物序列
引物由上海桑尼生物工程有限公司合成。参照文献分别合成qnrA、qnrB、qnrS基因[4]以及aac(6′)-Ib基因[5]检测所需引物,采用DNA-star软件设计qepA基因引物,引物序列及合成片段长度见表1。
1.2 方法
1.2.1 菌株鉴定和药敏试验
应用VITEK-compact型全自动微生物分析仪进行菌种鉴定和药敏试验,对环丙沙星敏感的菌株用K-B法检测确认。
表1 qnrA、qnrB、qnrS、aac(6′)-Ib和qepA基因的PCR检测所需引物
Table 1 Primers for PCR detection of qnrA, qnrB, qnrS, aac(6′)-Ib and qepA genes
| 引物 | 引物序列(5′→3′) | 检测基因 | 目的片段大小(bp) |
| QnrAm-F | AGA GGA TTT CTC ACG CCA GG | qnrA1 to qnrA6 | 580 |
| QnrAm-R | TGC CAG GCA CAG ATC TTG AC | ||
| QnrBm-F | CCT GAG CGG CAC TGA ATT TAT | qnrB1 to qnrB6 | 408 |
| QnrBm-R | GTT TGC TGC TCG CCA GTC GA | ||
| QnrSm-F | CAA TCA TAC ATA TCG GCA CC | qnrS1 to qnrS2 | 641 |
| QnrSm-R | TCA GGA TAA ACA ACA ATA CCC | ||
| Aac-F | TTG CGA TGC TCT ATG AGT GGC TA | aac(6′)-Ib | 482 |
| Aac-R | CTC GAA TGC CTG GCG TGT TT | ||
| qepA-F | GGA CAT CTA CGG CTT CTT CG | qepA | 544 |
| qepA-R | GGT GAT GAT GAT CTC GTT GC |
1.2.2 超广谱β-内酰胺酶(Extended Spectrum Beta-Lactamases,ESBLs)检测
采用CLSI推荐的双纸片扩散法,用头孢噻肟(30 μg)、头孢噻肟/克拉维酸(30 μg/10 μg)和头孢他啶(30 μg)、头孢他啶/克拉维酸(30 μg/10 μg)两对纸片进行ESBLs筛选,严格按照CLSI推荐的操作和结果判断标准进行[6]: 挑取血琼脂平板上新鲜的菌落,经肉汤稀释至0.5麦氏浊度菌悬液,涂布与M-H琼脂平板,贴上两对纸片,35 ℃孵育18~24 h后,判读结果为两组中任何一组药物,加克拉维酸与不加克拉维酸的抑菌圈相比,增大值≥5 mm时即判定为产ESBLs。以肺炎克雷伯菌ATCC700603为阳性对照。
1.2.3 聚合酶链反应(PCR)模板的制备
采用煮沸法。挑取3~5个菌落到加有150 μl无菌双蒸水的微量离心管,充分混匀,在100 ℃煮沸 15 min,13 000 r/min高速离心10 min,冷却后,吸取上清液(含细菌DNA),装入另一微量离心管,-80 ℃保存。
1.2.4 PCR
普通PCR方法, 检测质粒介导喹诺酮类药物耐药相关基因qnr、aac(6′)-Ib-cr和qepA。PCR反应体系:总体积为50 μl,其中 10×Taq buffer 5 μl,MgCl2(25 mmol/L)3 μl, dNTP(10 mmol/L)1 μl,Taq DNA聚合酶(1 U/μl)2 μl, 引物-F、R (30 μmol/L)各1 μl, DNA模板3 μl,加水补足至50 μl。PCR扩增:94 ℃ 5 min预变性,循环变性 94 ℃ 40 s,复性55 ℃ 40 s,延伸65 ℃ 40 s,共30个循环;最后72 ℃ 5 min延伸。PCR产物在1.0%琼脂糖凝胶中电泳约40 min,紫外灯下观察结果, 凝胶成像系统Bisoens SC620(Pharamacin Biotech公司)拍照。
1.2.5 DNA序列分析
将 qnr、aac(6′)-Ib阳性株的PCR扩增产物送上海桑尼生物工程有限公司进行DNA测序,DNA序列利用NCBI网站(www.ncbi.nlm.nih.gov/)的Blastn程序进行比对确定基因型。
1.2.6 微量肉汤稀释法
CLSI推荐用阳离子校正的M-H培养基。抗生素经精确称量稀释后定量加入微量板各凹孔中,在凹孔中进行连续稀释,使微量板凹孔中含有梯度浓度的抗生素,末孔为对照。用生长法或直接调制菌悬液制备浓度为0.5麦氏浊度的被测菌株菌液,再将菌液稀释200倍,定量加入每凹孔,微量板加盖或置湿盒中,以防水分蒸发,35 ℃孵育16~20 h后查看结果。
1.2.7 质粒接合转移试验
参照张雪青等[7]的方法,以利福平抗性大肠埃希菌(E.coli 600,Nalr,Rifr,LacZ-)为接合实验受体菌,临床分离株为供体菌进行实验。取对数生长期的供体菌及受体菌各0.5 ml加到4 ml新鲜LB肉汤中振荡培养过夜,然后取适量培养液,涂布含利福平(512 mg/L)、氯霉素(50 mg/L)和四环素(20 mg/L)的胰大豆琼脂(TSA)平板筛选接合转移子。以微量肉汤稀释法测定供体株、受体株和接合转移子对喹诺酮类和氨基糖甙类的最小抑菌浓度值(minimum inhibitory concentration,MIC),进行耐药性分析;对接合转移子进行耐药基因的PCR扩增,并将产物送上海桑尼生物工程有限公司进行DNA序列分析,以确证接合转移子中耐药基因的存在状况。
2 结果
2.1 药敏结果 419株非发酵菌对头孢呋辛、头孢噻肟、庆大霉素、阿米卡星、头孢吡肟和环丙沙星的敏感率分别为20.8%(87/481)、36.8%(154/481)、61.8%(259/481)、63.2%(265/481)、65.2%(273/481)和66.8%(280/481)。181株环丙沙星敏感肠杆菌科细菌对头孢噻肟、头孢吡肟、头孢呋辛、庆大霉素和阿米卡星敏感率分别为60.8%(110/181)、61.9%(112/181)、67.4%(122/181)、77.3%(140/181)和97.2%(176/181)。
2.2 qnr基因的检测结果
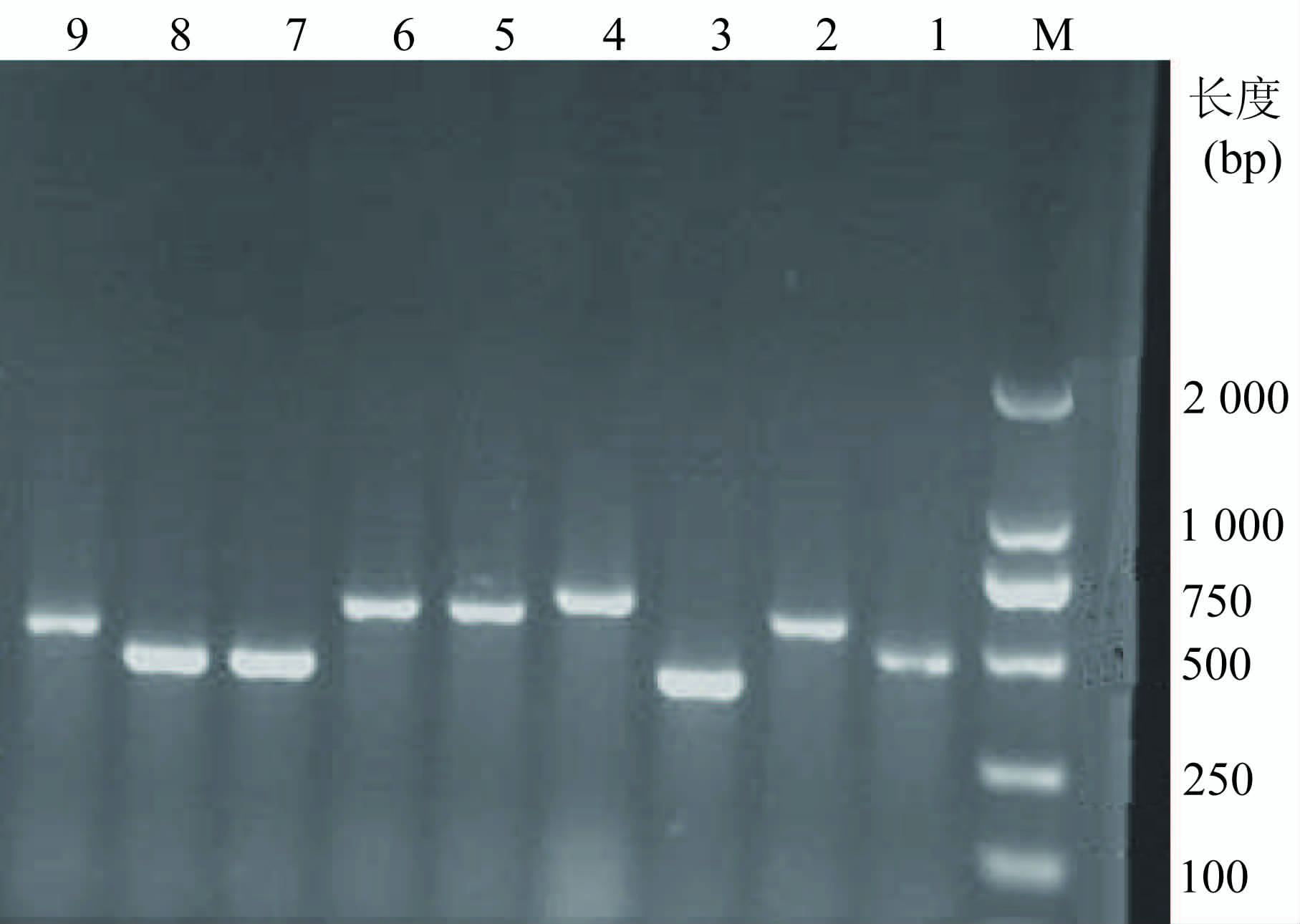
419株非发酵菌临床株中未检测到qnrA和qnrB阳性株,有2株洋葱假单胞菌检到与qnrS基因条带相似产物,经测序证实为非qnrS基因;181株环丙沙星敏感的肠杆菌科细菌临床株中检测到2株qnrA阳性株(1株肺炎克雷伯菌和1株大肠埃希菌)和2株qnrB阳性株(2株阴沟肠杆菌复合菌),未检测到qnrS阳性株。经DNA序列分析证实,2株qnrA基因阳性菌株均为qnrA1亚型,2株qnrB基因阳性菌株均为qnrB4亚型。扩增产物电泳条带见图1。
图1 qnrA、qnrB、qnrS和aac(6′)-Ib基因的PCR扩增产物电泳图
Figure 1 Electrophoresis of PCR amplification products of qnrA,qnrB, qnrS and aac(6′)-Ib genes 注:1~4:分别为aac(6′)-Ib,qnrA,qnrB,qnrS基因阳性质控菌株; 5,6:qnrA基因阳性的临床株; 7,8:qnrB基因阳性的临床株; 9:aac(6′)-Ib基因阳性的临床株;M:DNA Ladder Marker。
2.3 aac(6′)-Ib的检测结果
419株非发酵菌临床株中未检测到aac(6′)-Ib阳性株,181株环丙沙星敏感的肠杆菌科细菌临床株中检测到15株aac(6′)- Ib阳性株。15株aac(6′)-Ib阳性菌的PCR扩增产物进行DNA序列分析,经DNA测序证实12株为aac(6′)-Ib-cr,包括6株大肠埃希菌、3株阴沟肠杆菌复合菌和3株克雷伯菌属细菌。扩增产物电泳条带见图1。
2.4 qepA的检测结果
419株非发酵菌和181株环丙沙星敏感的肠杆菌科细菌中未检测出qepA基因。
2.5 ESBLs检测
181株环丙沙星敏感的肠杆菌科细菌中91株ESBLs阳性,阳性率为50.3%(91/181)。2株qnrA阳性株(1株肺炎克雷伯菌和1株大肠埃希菌) ESBLs阴性,2株qnrB阳性的阴沟肠杆菌复合菌ESBLs阳性。12株aac(6′)-Ib-cr阳性株中的4株大肠埃希菌、1株阴沟肠杆菌复合菌和1株肺炎克雷伯菌ESBLs阳性。
2.6 质粒接合转移试验
利福平抗性大肠埃希菌(E.coli 600,Nalr,Rifr,LacZ-)为接合实验受体菌,2 株qnrA1阳性株(1株肺炎克雷伯菌F1和1株大肠埃希菌E2)、2株qnrB4阳性株(2株阴沟肠杆菌复合菌Y3、Y4)和12株aac(6′)-Ib-cr阳性株(6株大肠埃希菌、3株阴沟肠杆菌复合菌和3株克雷伯菌属细菌A1-A12)为供体菌。2株qnrA1阳性株(F1和E2)、1株qnrB4阳性株(Y3)和4株aac(6′)-Ib-cr基因的阳性株(A2、A5为大肠埃希菌,A9为阴沟肠杆菌复合菌,A10为肺炎克雷伯菌)在TSA筛选平板上筛选到接合转移子。微量肉汤稀释法药敏结果表明:与受体菌相比,7株接合转移子对环丙沙星、左旋氧氟沙星的MIC提高了1~4个稀释度;对阿米卡星和庆大霉素的MIC提高了0~4个稀释度,见表2。对接合转移子进行qnr、aac(6′)-Ib的PCR扩增和DNA测序,结果显示与供体株的耐药基因类型一致。
表2 供体菌、接合转移子和受体菌对喹诺酮和氨基糖甙类药物的耐药情况
Table 2 Drug resistance of donors, transconjugants and recipients against quinolones and aminoglycosides
| 菌株 | 菌株 编号 | 环丙 沙星 | 左旋氧 氟沙星 | 阿米 卡星 | 庆大 霉素 |
| 供体菌 | F1 | 0.250 | 0.250 | 8.000 | 4.000 |
| E2 | 0.500 | 0.500 | 2.000 | 2.000 | |
| Y3 | 0.250 | 0.500 | 16.000 | 4.000 | |
| A2 | 1.000 | 2.000 | 2.000 | 2.000 | |
| A5 | 0.500 | 1.000 | 4.000 | 4.000 | |
| A9 | 0.250 | 0.250 | 2.000 | 1.000 | |
| A10 | 1.000 | 2.000 | 2.000 | 4.000 | |
| 结合株 | Tf1 | 0.250 | 0.125 | 0.125 | 0.250 |
| Te2 | 0.125 | 0.250 | 0.125 | 0.125 | |
| Ty3 | 0.125 | 0.125 | 0.250 | 0.250 | |
| Ta2 | 0.250 | 0.500 | 0.500 | 0.250 | |
| Ta5 | 0.250 | 0.250 | 0.500 | 1.000 | |
| Ta9 | 0.125 | 0.250 | 0.500 | 0.500 | |
| Ta10 | 0.250 | 1.000 | 0.250 | 0.250 | |
| 受体菌(1) | EC600 | 0.063 | 0.063 | 0.125 | 0.250 |
3 讨论
1998年Martinez-Martinez等[8]首次证实肺炎克雷伯菌中存在质粒介导的喹诺酮类耐药机制,并将该耐药基因命名为qnr(现命名为qnrA),2005年从福氏志贺菌中发现了一种与qnrA基因有59%同源性的qnrS基因[9],Jacoby等在2006年发现了与qnrA基因有<40%同源性的qnrB基因。qnr基因可能是细菌对喹诺酮类药物耐药性形成过程中较早出现的耐药机制,研究显示携带qnr基因的细菌在喹诺酮类药物的选择压力下产生靶位突变而导致高度耐药的概率比不携带qnr基因的菌株高100倍以上[8]。Robicsek等[5]于2006年报道了氨基糖苷乙酰化酶突变体 能使环丙沙星、诺氟沙星侧链的氨基氮乙酰化而使其抗菌活性下降;2007年Yamane等[10]报道在大肠埃希菌中发现质粒介导的外排泵机制引起细菌对喹诺酮类药物敏感性下降,该质粒由qepA 基因编码,近年来对这三类质粒介导喹诺酮类药物耐药基因研究日趋详细深入。
本次研究显示,温州地区419株非发酵菌中未检到这三类质粒介导喹诺酮类耐药基因,而2008年Cattoir等[11]在法国水样标本的斑点产气单胞菌中检测到qnrS2(此为首次在非发酵菌中发现qnr基因的存在),中国四川杨仁国等[12]在115株鲍曼不动杆菌中检到2株qnrB1,这可能与地域差别有关,也可能与我们选取的非发酵菌菌种、数量有关。
质粒介导的喹诺酮类耐药主要为低水平耐药,可引起喹诺酮类药物的MIC值升高,但并不一定在耐药范围。赵旭等[13]的研究也表明临床的环丙沙星耐药株和敏感株中都可检测出qnr基因,因此,我们在以往的研究基础上对环丙沙星敏感肠杆菌科细菌的三类质粒介导喹诺酮类药物耐药基因的流行状况做进一步调查研究,181株菌中发现4株qnr和12株aac(6′)-Ib-cr阳性株,阳性率分别为2.2%和6.6%,而我们前期对环丙沙星耐药肠杆菌科细菌的检测结果显示,qnr和aac(6′)-Ib-cr阳性率分别为3.2%(15/461)和11.3%(52/461)[7],明显高于环丙沙星敏感菌。4株qnr阳性菌(3株结合成功)的环丙沙星MIC为0.25~0.5 mg/L,这些细菌对喹诺酮类药物敏感性的下降可能仅由qnr基因所致。 aac(6′)-Ib-cr是普通氨基糖甙类乙酰转移酶基因aac(6′)-Ib的变异体,两次碱基的突变赋予了其修饰喹诺酮类的特性,第一处在223位(T→C或T→A),导致的氨基酸改变为Trp→Arg(色氨酸→精氨酸);第二处在454位(G→T),导致的氨基酸改变为Asp→Tyr(天冬氨酸→酪氨酸)。aac(6′)-Ib-cr是第一个被发现的可以同时对两类药物呈现耐药的酶。本次调查中发现该基因在环丙沙星敏感肠杆菌科细菌中可以存在,主要因为aac(6′)-Ib-cr编码的酶可以提高喹诺酮类药物对肠杆菌科细菌的MIC值,但并不一定达到耐药水平。含aac(6′)-Ib-cr的接合转移子对喹诺酮类、氨基糖苷类抗生素的敏感性都发生了变化,提示aac(6′)-Ib-cr基因对这两类药物敏感性下降起一定的作用。
qepA基因系近年发现的质粒介导的喹诺酮类药物耐药基因,相关细菌中分子流行病学调查资料比较少。本次研究中未检测到qepA基因,可能由于样本量偏小,也提示肠杆菌科中qepA 基因介导的喹诺酮类药物耐药可能比较少见,如2002 2006年日本140家医院临床分离的751株大肠埃希菌中仅检到2株携带qepA基因,检出率为0.3%[14]。有研究表明,qepA基因在动物分离的肠杆菌科细菌较为普遍,而在临床分离的肠杆菌科细菌中较少见[15]。181株肠杆菌科中91株产ESBLs菌株,qnr和aac(6′)-Ib-cr基因的阳性率为8.79%(8/91),90株非产ESBLs菌株中阳性率为8.89%(8/90),两组细菌基因阳性率经χ2检验,差异无统计学意义(P=0.60),提示在肠杆菌科细菌非产ESBLs菌株中qnr和aac(6′)-Ib-cr基因仍有较高的检出率。
16株试验菌株质粒接合转移试验中有7株成功,提示qnr、aac(6′)-Ib-cr基因在有些菌株里可能位于非接合转移性质粒上,而位于可接合转移耐药质粒上的喹诺酮类耐药基因可以在不同菌株间传播,应引起临床关注。
综上所述,在温州地区质粒介导喹诺酮类药物耐药基因不仅存在于环丙沙星耐药株中而且存在于环丙沙星敏感的肠杆菌科细菌中,但是,这些耐药基因可能不存在于非发酵菌中。
参考文献
[1] Hooper DC. Mechanisms of action and resistance of older and newer fluoroquinolone [J]. Clin Infect Dis, 2000, 31 (Suppl 2):S24-28.
[2] RodriguezMartinez JM, Velasco C, Pascual A, et al. Correlation of quinolone resistance levels and differences in basal and quinoloneinduced expression from three qnrA containing plasmids[J]. Clin Microbiol Infect, 2006, 12(5):440-445.
[3] Park CH, Robicsek A, Jacoby GA, et al. Prevalence in the United States of aac (6') Ibcr encoding a ciprofloxacinmodifying enzyme[J]. Antimicrob Agents Chemother, 2006, 50(11):3953-3955.
[4] Wu JJ, Ko WC, Tsai SH, et al. Prevalence of plasmidmediated quinolone resistance determinants QnrA, QnrB, and QnrS among clinical isolates of Enterobacter cloacae in a Taiwanese hospital[J]. Antimicrob Agents Chemoth, 2007, 51(4):1223-1227.
[5] Robicsek A, Strahilevitz J, Jacoby GA, et al. Fluoroquinolone modifying enzyme: a new adaptation of a common aminoglycoside acetyltransferase[J]. Nat Med, 2006, 12(1):83-88.
[6] CLSI. M100S17 Performance standards for antimicrobial susceptibility testing; 17th informational. Supplement. Wayne, PA: Clinical and Laboratory Standards Institute, 2012.
[7] Zhang XQ, Chen ZQ, Chen J, et al. The examination of qnr and aac (6′) Ibcr genes of clinical isolates resistant to ciprofloxacin in Enterobacteriaceae[J]. Chinese Journal of Microbiology and Immunology, 2009, 29(3):265-270.(in Chinese)张雪青, 陈增强, 陈坚, 等. 环丙沙星耐药肠杆菌科细菌临床株 qnr 和 aac (6') Ibcr 基因的检测[J].中华微生物学和免疫学杂志, 2009, 29(3):265-270.
[8] MartinezMartinez I, Pascua1 A, Jacoby GA.Quinolone resistance from a transferable plasmid[J]. Lancet, 1998, 351(9105):797-799.
[9] Hata M, Suzuki M, Matsumoto M, et al.Cloning of a novel gene for quinolone resistance from a transferable plasmid in Shigella flexneri 2b[J]. Antimicrob Agents Chemother, 2005, 49(2):801-803.
[10] Yamane K, Wachino J, Suzuki S, et al.New plasmidmediated f1uoroquinolone efflux pump, QepA, found in an Escherichia coli clinical isolate[J]. Antimierob Agents Chemother, 2007, 51(9):3354-3360.
[11] Cattoir V, Poiret L, Auhert C, et al.Unexpected occurrence of plasmidmediated quinolone resistance determinants in environmental Aeromonas spp[J]. Emergence Infect Disese, 2008, 14(2):231-237.
[12] Yang RG, Yu RJ, Gao YY, et al. Detection of qnr gene and drug resistance in clinical isolates of Acinetobacter baumannii [J]. Chinese Journal of Antibiotics, 2009, 34(12):747-749.(in Chinese)杨仁国, 俞汝佳, 高燕渝, 等. 临床分离鲍曼不动杆菌耐药性及 qnr 基因检测 [J]. 中国抗生素杂志, 2009, 34(12):747-749.
[13] Zhao X, Xu XG, Zhu DM, et al. Detection of plasmidmediated quinolone resistance determinants in clinical isolates of Enterobacter cloacae[J]. Chinese Journal of Infection and Chemotherapy, 2009, 9(3):210-215.(in Chinese)赵旭, 徐晓刚, 朱德妹, 等. 阴沟肠杆菌临床分离株中3类质粒介导喹诺酮类药物耐药基因的检测[J]. 中国感染与化疗杂志, 2009, 9(3):210-215.
[14] Yamane K, Wachino J, Suzuki S, et al.Plasmidmediated qepA gene among Escherichia coli clinical isolates from Japan[J]. Antimierob Agents Chemother, 2008, 52(4);1564-1566.
[15] Liu JH, Deng YT, Zeng ZL, et al.Coprevalence of plasmidmediated quinolone resistance determinants QepA, Qnr and aac (6′) Ibcr nmong 16S rRNA methylase RmtBprodueing Escherichia coli isolates from pigs[J]. Antimicrob Agents Chemother, 2008, 52(8):2992-2993.
[2] RodriguezMartinez JM, Velasco C, Pascual A, et al. Correlation of quinolone resistance levels and differences in basal and quinoloneinduced expression from three qnrA containing plasmids[J]. Clin Microbiol Infect, 2006, 12(5):440-445.
[3] Park CH, Robicsek A, Jacoby GA, et al. Prevalence in the United States of aac (6') Ibcr encoding a ciprofloxacinmodifying enzyme[J]. Antimicrob Agents Chemother, 2006, 50(11):3953-3955.
[4] Wu JJ, Ko WC, Tsai SH, et al. Prevalence of plasmidmediated quinolone resistance determinants QnrA, QnrB, and QnrS among clinical isolates of Enterobacter cloacae in a Taiwanese hospital[J]. Antimicrob Agents Chemoth, 2007, 51(4):1223-1227.
[5] Robicsek A, Strahilevitz J, Jacoby GA, et al. Fluoroquinolone modifying enzyme: a new adaptation of a common aminoglycoside acetyltransferase[J]. Nat Med, 2006, 12(1):83-88.
[6] CLSI. M100S17 Performance standards for antimicrobial susceptibility testing; 17th informational. Supplement. Wayne, PA: Clinical and Laboratory Standards Institute, 2012.
[7] Zhang XQ, Chen ZQ, Chen J, et al. The examination of qnr and aac (6′) Ibcr genes of clinical isolates resistant to ciprofloxacin in Enterobacteriaceae[J]. Chinese Journal of Microbiology and Immunology, 2009, 29(3):265-270.(in Chinese)张雪青, 陈增强, 陈坚, 等. 环丙沙星耐药肠杆菌科细菌临床株 qnr 和 aac (6') Ibcr 基因的检测[J].中华微生物学和免疫学杂志, 2009, 29(3):265-270.
[8] MartinezMartinez I, Pascua1 A, Jacoby GA.Quinolone resistance from a transferable plasmid[J]. Lancet, 1998, 351(9105):797-799.
[9] Hata M, Suzuki M, Matsumoto M, et al.Cloning of a novel gene for quinolone resistance from a transferable plasmid in Shigella flexneri 2b[J]. Antimicrob Agents Chemother, 2005, 49(2):801-803.
[10] Yamane K, Wachino J, Suzuki S, et al.New plasmidmediated f1uoroquinolone efflux pump, QepA, found in an Escherichia coli clinical isolate[J]. Antimierob Agents Chemother, 2007, 51(9):3354-3360.
[11] Cattoir V, Poiret L, Auhert C, et al.Unexpected occurrence of plasmidmediated quinolone resistance determinants in environmental Aeromonas spp[J]. Emergence Infect Disese, 2008, 14(2):231-237.
[12] Yang RG, Yu RJ, Gao YY, et al. Detection of qnr gene and drug resistance in clinical isolates of Acinetobacter baumannii [J]. Chinese Journal of Antibiotics, 2009, 34(12):747-749.(in Chinese)杨仁国, 俞汝佳, 高燕渝, 等. 临床分离鲍曼不动杆菌耐药性及 qnr 基因检测 [J]. 中国抗生素杂志, 2009, 34(12):747-749.
[13] Zhao X, Xu XG, Zhu DM, et al. Detection of plasmidmediated quinolone resistance determinants in clinical isolates of Enterobacter cloacae[J]. Chinese Journal of Infection and Chemotherapy, 2009, 9(3):210-215.(in Chinese)赵旭, 徐晓刚, 朱德妹, 等. 阴沟肠杆菌临床分离株中3类质粒介导喹诺酮类药物耐药基因的检测[J]. 中国感染与化疗杂志, 2009, 9(3):210-215.
[14] Yamane K, Wachino J, Suzuki S, et al.Plasmidmediated qepA gene among Escherichia coli clinical isolates from Japan[J]. Antimierob Agents Chemother, 2008, 52(4);1564-1566.
[15] Liu JH, Deng YT, Zeng ZL, et al.Coprevalence of plasmidmediated quinolone resistance determinants QepA, Qnr and aac (6′) Ibcr nmong 16S rRNA methylase RmtBprodueing Escherichia coli isolates from pigs[J]. Antimicrob Agents Chemother, 2008, 52(8):2992-2993.