-
1 材料与方法
- 1.1 菌株
- 1.2 SRL、Tn7耐药岛序列比较方法
在发展中国家,福氏志贺菌(Shigella flexneri)仍然是引起细菌性腹泻的主要病原菌[1]。由于抗生素的广泛使用,志贺菌的耐药现象越来越严重[2-4]。1997年,Rajakumar等[5]在分离自20世纪60年代的日本福氏志贺菌YSH600(2a血清型)的染色体上发现了1个多重耐药岛,命名为志贺菌耐药岛(Shigella resistance locus,SRL)。SRL岛携带多个耐药基因,包括编码氨基糖苷类药物腺苷酰基转移酶(aminoglycoside adenyltransferase)的aadA2,编码β-内酰胺酶(β-lactamases)的 oxa-1 基因,编码氯霉素乙酰转移酶(chloramphenicol acetyltransferase)的cat基因和编码四环素耐药蛋白的tet基因簇(tetABCD)[5]。与之前在质粒上发现的耐药岛相比,YSH600菌株染色体上的SRL岛缺失了位于 cat和tet之间汞抗性基因和磺胺类药物(sulfonamide, SUL)的抗性基因,插入了1个编码β-内酰胺酶、与氨苄西林(ampicillin,AMP)耐药相关的基因oxa-1[6-8]。我国分离的福氏志贺菌序列型91(sequence type,ST)菌株2002017(Xv血清型)的基因组中也有SRL岛[9]。其携带的4个耐药基因aadA2、 oxa-1, cat和tet与YSH600菌株相同[9]。但是,中国菌株的SRL耐药岛还有两个特征,一是在YSH600菌株SRL岛上插入了1个编码四环素耐药蛋白的tet基因簇(tetABCD),所以有2个编码四环素耐药的基因簇;二是没有YSH600菌株PAI岛(pathogenicity island,PAI)上编码铁转运系统(ferric transport system)的fec基因簇(fecABCDE)和转录调控基因fecR, fecI[9-10]。
除了SRL岛外,志贺菌的质粒上有1个编码多种耐药基因的Tn7耐药岛[5,11-12]。Tn7岛携带编码氨基糖苷类药物腺苷酰基转移酶(aminoglycoside adenyltransferase)的 aadA1、 编码链丝菌素乙酰转移酶(streptothricin acetyltransferase)的 sat1 和编码二氢叶酸还原酶(dihydrofolate reductase)的 dfrA1 基因[13]。携带这个耐药岛的菌株,对链霉素(streptomycin, STR)、链丝菌素(streptothricin)、甲氧苄啶(trimethoprim, TMP)耐药[11-12]。2002017菌株的Tn7岛与2457T(2a血清型,ST86序列型)、Sf301(2a血清型,ST18序列型)和Sf8401(5b血清型,ST93序列型)菌株的基本相同,序列较为保守[9]。
我国福氏志贺菌Xv血清型是2000年首先在河南出现的,分离率逐年升高,于2002 2006年间取代2a成为河南省的优势血清型[9]。随后Xv血清型在中国的其他省份也相继出现,也一度成为优势血清型[9]。研究发现,我国分离的Xv血清型菌株为ST91序列型 。ST91是我国福氏志贺菌的优势序列型,由于编码不同群抗原、型抗原的噬菌体、质粒的转移,形成了多种血清型[14]。本研究主要探讨我国优势序列型ST91菌株的SRL、Tn7耐药岛的分布及变化特征。
59株福氏志贺菌覆盖7个序列型,包括ST91(50株),ST109(1株),ST18(3株),ST142(1株),ST143(1株),ST103(2株),ST16(1株);覆盖14个血清型,包括 Xv(25株),X(9株),1a(7株),1b(1株),1d(1株),2a(5株),2b(1株),3a(3株),3b(1株),4a(1株),4av(1株),4b(1株),Y(2株)和Yv(1株)。菌株来源于6个省份,其中河南(38株),甘肃(8株),安徽(7株),山西(3株),贵州(2株),四川(1株)。分离年代,55株分离自1997 2006年,4株分离自20世纪80年代。菌株主要来源于中国疾病预防控制中心(CDC)的志贺菌感染监测项目(2004 2006年),均分离自腹泻患者,并经生化鉴定及血清型复核后-80 ℃保存于本实验室。59株菌基因序列号(GenBank accession number)为AZOG00000000~AZQM00000000, 版本号为AZOG00000000.1~AZQM00000000.1,Bioproject ID为 PRJNA230538。
通过对59株菌的基因组序列进行扫描,找出每株菌的SRL和Tn7岛核酸序列。使用序列比对软件Artimes(Release 8)将每株菌的SRL和Tn7岛的序列与参考菌株2002017(Genbank accession number CP001383.1)和YSH600(Genbank accession number AF326777.3)的序列进行比较,查找插入和缺失(insertion, duplication and deletion, Indels)片段。大、小片段的Indels分别使用mauve软件和BWA(Burrows-Wheeler Alignment)软件[15]进行查找。最后对插入和缺失片段的核酸序列进行注释。
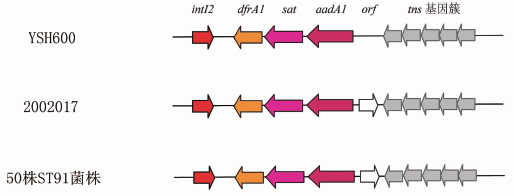
我们分析的所有50株ST91菌株均携带SRL和Tn7耐药岛。所分析的1株ST109(Shi06HN159)和1株ST142(2002091)也携带这2个岛。1株ST18菌株(菌株号51581)仅携带2个耐药岛上的3个耐药基因(SRL岛上的 aadA2和cat 基因,Tn7岛上的 aadA1 基因)(图1)。2株ST18菌株(菌株号2002007,2002028),1株ST143菌株(菌株号1997005),2株ST103菌株(菌株号51576,51577)和1株ST16菌株(菌株号51575)的基因组序列未发现SRL和Tn7岛。所有50株ST91菌株,1株ST109和1株ST142菌株携带的Tn7耐药岛的核酸序列及编码基因与参考菌株2002017相同(图2)。携带SRL和Tn7耐药岛的50株ST91的分离时间为2000 2006年,提示我国福氏志贺菌ST91最迟在2000年左右已经获得上述2个耐药岛。
注:参考菌株为YSH600(GenBank accession number AF326777.3)。基因名称及编码ORF名称标注在上面,不同的编码基因使用不同颜色表示。
图1 YSH600,2002017,51581,51575的SRL岛上基因及编码ORF比较
Figure 1 Comparison of open reading frame of SRL island genes of YSH600,2002017, 51581 and 51575
注:参考菌株为YSH600(GenBank accession number AF326777.3)。基因名称及编码ORF名称标注在上面, 不同的编码基因使用不同颜色表示。菌株号标在左边。
图2 ST91菌株的Tn7岛上基因及编码ORF比较
Figure 2 Comparison of open reading frame of Tn7 island genes of ST91 isolates
在50株ST91菌株中,有9株的SRL耐药岛发生了插入或缺失。其中,有7株ST91菌株缺失了四环素耐药基因簇(tetABCD)。包括2005051菌株缺失了位于ORF SFxv_1138-SFxv_1140 之间的tet基因簇,缺失片段长度为2340 bp;2002106菌株缺失了位于ORF SFxv_1151-SFxv_1155之间的tet基因簇,缺失片段长度为4644 bp;Shi06GS43和Shi06AH130菌株缺失了长度为870 bp的insB和tetD基因;2003035和2001048菌株在ORF SFxv_1138和SFxv_1138a的位置缺失了insB、tetC和tetD基因,缺失片段长为1194 bp;Shi06GS48菌株在2个tet基因簇位置上都缺失了一个416 bp的tetD基因。另外,2005AH264菌株缺失了编码β-内酰胺酶的 oxa-1 和编码氯霉素乙酰转移酶的cat耐药基因,缺失片段长度为1004 bp。2001042菌株在SRL岛上的1,122,571位置(ORF SFxv_1155,insB)插入了一段编码广谱β-内酰胺酶的bla TEM-1基因。其bla TEM-1核酸序列同源性与E.coli 13-5(GenBank accession number HQ174461.1)相比达到100%。见图3和表1。
与2002017相比,日本菌株YSH600的SRL岛下游还存在一个PAI岛。其携带的铁转运相关的基因簇就位于这个岛上。铁转运基因簇包括操纵子fecABCDE和发挥转录调控作用的fecI、fecR基因。在我们分析的59株中国分离菌株中,只有1株分离自20世纪80年代的ST16菌株(菌株号51575,3a血清型)携带fec基因簇,包括操纵子和调控基因(图1)。其核酸序列的同源性和日本菌株YSH600相比达100%。
注:参考菌株为2002017(GenBank accession number CP0013831)。基因名称及编码ORF名称标注在上面, 不同的编码基因使用不同颜色表示。SRL岛两端的保守基因分别用红色和绿色表示。菌株号标在左边。
图3 ST91菌株的SRL岛上基因及编码ORF比较
Figure 3 Comparison of open reading frame of SRL island genes of ST91 isolates
表1 中国分离的福氏志贺菌SRL耐药岛插入和缺失分析
Table 1 Insertion or deletions on SRL island of S. flexneri strains isolated in China

|
笔者实验室最近的研究发现,福氏志贺菌ST91序列型是1993年左右在我国出现的[14],最迟在2000年前后获得SRL和Tn7耐药岛,最终成为我国的优势序列型。SRL岛是一个整合子,可以不断丢失和捕获耐药基因,这和面临的抗生素选择压力有关[7-8]。本研究中国菌株的SRL耐药岛和日本菌株发现的SRL岛不同,发生了四环素耐药基因一些拷贝的缺失以及其他耐药基因的缺失和插入。而我国菌株携带的Tn7耐药岛和日本菌株的基本一致,没有明显差异。另外,分离自20世纪80年代的1株ST16,虽然没有SRL和Tn7耐药岛,但携带fec基因簇。其基因序列和分离自60年代的日本菌株YSH600的核酸序列完全一致。说明fec基因簇在ST91成为中国优势序列型及Xv新血清型流行之前,就已经存在。
近年来在中国的不同地区Xv血清型逐渐取代2a一度成为当地的优势血清型[9]。我国20世纪80年代分离的福氏2a血清型志贺菌301菌株属于ST18序列型,没有获得耐药岛[14]。本实验室的1株ST18菌株没有获得完整的耐药岛,仅携带岛上的3个耐药基因,而其他2株2a血清型的ST18菌株未获得2个岛。提示在ST91成为优势序列型之前,ST18序列型可能在我国已经广泛存在,而且在获得耐药岛的过程中发挥重要作用。另外,我们的前期研究结果显示,ST109和ST91基因组序列很相似,但在进化上晚于ST91[14,16]。本研究提示,不能排除ST109从携带耐药岛的ST91菌株进化而来的可能性。
[2] Watanabe T. Infective heredity of multiple drug resistance in bacteria[J]. Bacteriol Rev, 1963,27:87-115.
[3] Nakaya R, Nakamura A, Murata Y. Resistance transfer agents in Shigella[J]. Biochem Biophys Res Commun, 1960,3:654-659.
[4] Womble DD, Rownd RH. Genetic and physical map of plasmid NR1: comparison with other IncFII antibiotic resistance plasmids[J]. Microbiol Rev, 1988,52(4):433-451.
[5] Rajakumar K, Bulach D, Davies J, et al. Identification of a chromosomal Shigella flexneri multi-antibiotic resistance locus which shares sequence and organizational similarity with the resistance region of the plasmid NR1[J]. Plasmid, 1997,37(3):159-168.
[6] Tanaka M, Matsushita K, Yamamoto T. Genesis of a complex transposon encoding the OXA-1 (type Ⅱ)beta-lactamase gene[J]. Antimicrob Agents Chemother, 1985,28(2):227-234.
[7] Turner SA, Luck SN, Sakellaris H, et al. Molecular epidemiology of the SRL pathogenicity island[J]. Antimicrob Agents Chemother, 2003,47(2):727-734.
[8] Turner SA, Luck SN, Sakellaris H, et al. Nested deletions of the SRL pathogenicity island of Shigella flexneri 2a[J]. J Bacteriol, 2001,183(19):5535-5543.
[9] Ye C, Lan R, Xia S, et al. Emergence of a new multidrug-resistant serotype X variant in an epidemic clone of Shigella flexneri[J]. J Clin Microbiol, 2010,48(2):419-426.
[10] Luck SN, Turner SA, Rajakumar K, et al. Ferric dicitrate transport system (Fec)of Shigella flexneri 2a YSH600 is encoded on a novel pathogenicity island carrying multiple antibiotic resistance genes[J]. Infect Immun, 2001,69(10):6012-6021.
[11] Sundstrom L, Radstrom P, Swedberg G, et al. Site-specific recombination promotes linkage between trimethoprim and sulfonamide resistance genes[J]. Mol Gen Genet, 1988,213 (2/3): 191-201.
[12] Sundstrom L, Skold O. The dfrI trimethoprim resistance gene of Tn7 can be found at specific sites in other genetic surroundings[J]. Antimicrob Agents Chemother, 1990,34(4):642-650.
[13] Sundstrom L, Roy PH, Skold O. Site-specific insertion of three structural gene cassettes in transposon Tn7[J]. J Bacteriol, 1991,173(9):3025-3028.
[14] Zhang N, Lan R, Sun Q, et al. Genomic portrait of evolution and epidemic spread of a recently emerged multidrug resistant Shigella flexneri clone in China[J]. J Clin Microbiol, 2014,52(4):1119-1126.
[15] Li H, Durbin R. Fast and accurate long-read alignment with Burrows-Wheeler transform[J]. Bioinformatics, 2010,26(5):589-595.
[16] Luo X, Sun Q, Lan R, et al. Emergence of a novel Shigella flexneri serotype 1d in China[J]. Diagn Microbiol Infect Dis, 2012,74(3):316-319.
|
扩展功能
|
|
| 本文信息 | |
| PDF全文 | |
| HTML全文 | |
| 参考文献 | |
| 服务与反馈 | |
| 加入引用管理器 | |
| 引用本文 | |
| Email Alert | |
| 本文作者相关文章 | |
| 张楠 | |
| 孙强正 | |
| 王建平 | |
| 代航 | |
| 徐建国 | |
| PubMed | |
| Article by ZHANG Nan | |
| Article by SUN Qiang-zheng | |
| Article by WANG Jian-ping | |
| Article by DAI Hang | |
| Article by XU Jian-guo | |