扩展功能
文章信息
- 龚林, 袁敏, 陈霞, 禹蕙兰, 卢金星, 李娟
- GONG Lin, YUAN Min, CHEN Xia, YU Hui-lan, LU Jin-xing, LI Juan
- 氨基糖苷类药物耐药基因armA实时荧光定量聚合酶链反应检测方法的建立
- Establishment of real-time PCR assay to detect resistance gene armA
- 疾病监测, 2014, 29(11): 901-904
- Disease Surveillance, 2014, 29(11): 901-904
- 10.3784/j.issn.1003-9961.2014.11.015
-
文章历史
- 收稿日期:2014-06-25
氨基糖苷类抗生素作为一类高效、广谱的抗菌药物,一直是临床上治疗革兰阴性菌所致感染的重要药物。但是随着此类药物的广泛使用,对其耐药的菌株不断出现,耐药率逐渐上升,严重影响了该类抗生素在临床中的使用[1]。革兰阴性菌对氨基糖苷类抗生素耐药机制主要体现在如下几点:(1)细菌产生针对药物的修饰酶。(2)外膜孔蛋白的缺失或表达下降以及药物外排泵的激活和过度表达。(3)核糖体结合位点的改变[2]。近年来出现了一种新的由质粒介导的耐药机制——16S rRNA甲基化酶,导致革兰阴性杆菌对阿米卡星等多种临床常用氨基糖苷类药物高度耐药。该酶修饰的是所有氨基糖苷类共同的作用位点,一旦修饰发生,几乎所有的氨基糖苷类抗生素都将失去作用[3, 4]。
目前,编码16S rRNA甲基化酶的基因主要包括rmtA、rmtB、rmtC、rmtD和armA[5, 6, 7],其中armA基因在世界各地广泛分布,并且该基因在不同国家和地区的不同细菌中分布大相径庭。我国也鲜有关于armA分布的报道,因此有必要进行较大规模的armA耐药基因监测,以明确各地区armA在氨基糖苷类抗生素耐药中的地位和作用。目前国内对于armA基因的检测主要使用的是聚合酶链反应(polymerase chain reaction,PCR),但是该方法检测存在耗时过长、步骤繁琐、不能定量等不足。实时荧光定量PCR(quantitive real-time PCR)是一种将常规PCR与荧光检测相结合的核酸定量检测技术,具有操作简便、特异性强、敏感性高、重复性好、用时短和可自动定量分析等优点,已成为病原体检测的重要方法[8, 9]。本研究拟建立一种快速、灵敏、特异的armA基因 real-time PCR检测方法,以期为探索armA基因的流行特征提供技术支持。 1 材料与方法 1.1 材料 1.1.1 实验菌株
携带armA基因的肺炎克雷伯菌SY230 1株,鲍曼不动杆菌、铜绿假单胞菌、阴沟肠杆菌、大肠埃希菌各2株,携带rmtB基因的大肠埃希菌和肺炎克雷伯菌各2株,携带aac(6′)-Ⅱ基因(氨基糖苷类乙酰化酶基因)的铜绿假单胞菌2株,携带aac(3′)-Ⅱ基因(氨基糖苷类乙酰化酶基因)的阴沟肠杆菌2株,对氨基糖苷类药物敏感,不携带上述耐药基因的菌株(包括肺炎克雷伯菌、大肠埃希菌、铜绿假单胞菌、产气肠杆菌、阴沟肠杆菌、粘质沙雷菌、弗氏柠檬酸杆菌、鲍曼不动杆菌、雷氏普罗威登斯菌、奇异变形杆菌各2株),均来自中国疾病预防控制中心(CDC)传染病预防控制所。 1.1.2 引物及探针
根据GenBank上公布的armA的基因序列,使用Primer Premier 5.0 软件设计引物和探针,见表 1。引物和探针均为上海基康生物技术有限公司合成。
| 引物名称 | 序列(5′~3′) | 扩增片段 长度(bp) |
| armA |
上游引物-F:TCA AAA ACC TAT ACT TTA TCG TCG TCT T | 160 |
| 下游引物-R:TAT TTT AGA TTT TGG TTG TGG CTT CA | ||
| TaqMan探针-P: FAM-AAC TTC CCA ATA ATG CTA C-MGB |
Rotor-Gene Q real time PCR仪、QIAamp DNA Stool Mini Kit 9 和QIAGEN Plasmid Mini Kit 来自德国QIAGEN公司,Peasy-t1 simple cloning vector、Trans1-T1感受态细胞、real-time PCR反应试剂Premix Ex TaqTM,均购自大连宝生物工程有限公司,DNA基因组提取试剂盒购自康为世纪生物科技有限公司,NanoDrop ND-1000分光光度计为美国Thermo公司产品。 1.2 方法 1.2.1 DNA的提取
按照说明书,用DNA基因组提取试剂盒提取实验菌株基因组DNA,-20 ℃保存。 1.2.1 DNA的提取
Premix(TaKaRa) Ex TaqTM(2×)10 μl,armA基因上下游引物各0.4 μmol/L,探针0.2 μmol/L,1 μl DNA模板,补充去离子水至20 μl。反应条件采用两步法PCR扩增程序标准:95 ℃ 3 min;95 ℃ 5 s,55 ℃ 20 s,40个循环。 1.2.3 特异度评价
使用携带armA基因的肺炎克雷伯菌、鲍曼不动杆菌、铜绿假单胞菌、阴沟肠杆菌和大肠埃希菌;携带rmtB基因的大肠埃希菌和肺炎克雷伯菌;携带aac(6′)-Ⅱ基因的铜绿假单胞菌;携带aac(3)-Ⅱ基因的阴沟肠杆菌;以及不携带氨基糖苷类耐药基因的菌株(肺炎克雷伯菌、大肠埃希菌、铜绿假单胞菌、产气肠杆菌、阴沟肠杆菌、粘质沙雷菌、弗氏柠檬酸杆菌、鲍曼不动杆菌、雷氏普罗威登斯菌、奇异变形杆菌)的基因组核酸同时进行反转录-聚合酶链反应(RT-PCR),以确定该方法的特异性。 1.2.4 含armA重组质粒的制备
(1)目的基因的扩增:以携带armA的肺炎克雷伯菌SY230基因组DNA为模板,应用设计的引物对armA基因进行扩增,电泳检测产物片段;(2)纯化PCR产物;(3)目的片段与载体Peasy-t1 simple cloning vector连接;(4)将连接产物转入到Trans1-T1感受态细胞中;(5)PCR筛选阳性克隆子并测序确认;(6)提取质粒,-20 ℃保存,含有重组子的阳性菌-80 ℃保存[10]。 1.2.5 质粒拷贝数的计算
根据质粒的分子质量将质粒标本浓度换算为拷贝数浓度:每微升标本中检测基因的拷贝数=浓度(ng/μl)×6.02×1023×10-9/(660×重组质粒碱基数),其中重组质粒碱基数=Peasy-t1 simple cloning vector载体碱基数+目的基因碱基数[11]。用NanoDrop ND-1000分光光度计测定重组质粒浓度为178 ng/μl,经计算得出质粒标准品的拷贝数为4.07×1010拷贝/ml。 1.2.6 标准曲线的制备及灵敏度评价
将含有armA基因的重组质粒进行10×倍比稀释,使浓度梯度为4.07×108~4.07×100拷贝/ml,同时做3个平行标本,real-time PCR扩增,并制作相应的标准曲线,用于定量分析及灵敏度评价。 1.2.7 粪便模拟标本的制备以及检测
(1)粪便模拟标本的制备:挑取携带armA基因的肺炎克雷伯菌SY230,接种至脑心液体培养基,37 ℃恒温摇床过夜培养,移取50 μl细菌原液至5 ml新鲜脑心液体培养中,37 ℃恒温摇床培养至A600值达0.6,取菌悬液1 ml,10×倍比稀释至浓度为10-2~10-8的菌液。分别称取8管健康人粪便(约0.2 g/管),其中一管加100 μl磷酸盐缓冲液(PBS)作为空白对照,其他管分别加入100 μl 10-2~10-8的10×倍比稀释菌液。同时分别取100 μl 10-4~10-7的10×倍比稀释菌液,采取平板计数法,对各管所加的细菌进行计数,粪便中的菌浓度以cfu/ml表示。(2)粪便中肺炎克雷伯菌SY230的DNA提取:按QIAamp DNA Stool Mini Kit 操作提取。(3)real-time PCR检测,同时做3个平行样本[12]。 2 结果 2.1 引物和探针特异性的确定
对携带armA基因的肺炎克雷伯菌进行PCR扩增,电泳结果表明得到的片段与目的基因大小相符,DNA测序结果显示扩增产物序列与目的序列一致。real-time PCR方法对上述9株携带armA基因的菌株检测结果均为阳性,28株不携带armA基因的菌株检测结果均为阴性。 2.2 标准曲线的制备及灵敏度评价
对armA基因的重组质粒进行10×倍比稀释,使浓度达到4.07×108~4.07×100拷贝/ml共9个浓度梯度。real-time PCR结果显示,本方法质粒标准品检测下限为4.07×101拷贝/ml,由质粒拷贝浓度对数值与其对应Ct值作图显示质粒拷贝浓度对数值与其对应Ct值呈线性关系,标准曲线公式为:y=-3.438x + 14.020,R2>0.99,扩增效率为0.95,见图 1,2。
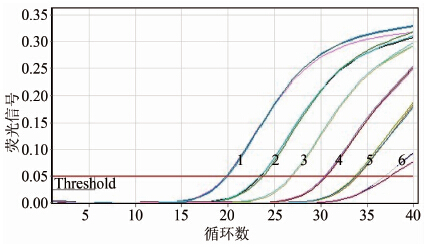
|
| 图 1 armA基因阳性质粒标准品荧光信号图 Figuer 1 Fluorescent graph of armA-positive standard plasmid 注:1~6分别为4.07×106~4.07×101拷贝/ml阳性质粒(空白对照图中未显示) |
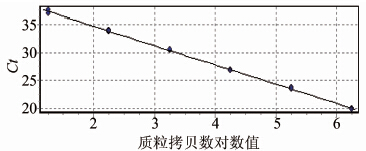
|
| 图 2 含armA基因的质粒拷贝数对数值及其对应Ct值的标准曲线 Figuer 2 Standard curve of armA-positive standard plasmid |
平板计数法的结果显示,10-2~10-8拷贝/ml的粪便菌液对应的浓度分别是1.3×106~1.3×100 cfu/ml。当粪便模拟标本的含菌量为1.3×106~1.3×104 cfu/ml时,所有标本的扩增荧光信号强度均高于检测阈值;当标本含菌量 为≤1.3×103 cfu/ml时,3个平行样本中均未出现特异性扩增,因此,本研究建立的TaqMan real-time PCR 方法对粪便模拟标本的检测下限为1.3×104 cfu/ml。见表 2。
| 样本名称 | 菌落浓度(cfu/ml) | |||
| 1.3×106 | 1.3×105 | 1.3×104 | 1.3×103 | |
| 平行样本1 | 27.55 | 30.30 | 33.99 | |
| 平行样本2 | 27.55 | 30.17 | 33.40 | |
| 平行样本3 | 27.56 | 30.19 | 33.24 | |
| 平均值 | 27.55 | 30.22 | 33.54 | |
armA作用于细菌30S核糖体亚单位中16S rRNA A位点,引起某个或某几个碱基甲基化,使氨基糖苷类药物不能与其作用靶点结合,从而导致细菌对氨基糖苷类抗生素耐药[13]。该编码基因通常位于接合性质粒上,因此具有高度可移动性,可在菌株间广泛交换和转移,使越来越多的临床分离菌产生对氨基糖苷类抗生素的高水平耐药[14]。建立快速准确的检测方法加强对armA基因的检测对了解armA的流行趋势,阻断其传播具有重要意义。
荧光定量PCR方法克服了普通PCR中耗时长、 工作量大等缺陷,充分发挥了自身灵敏度高、特异 性强、耗时短、自动化程度高等特点[15],大大提高了微生物的检出和鉴定效率。本研究中,选取armA基因作为靶基因设计引物和探针,建立了能检测耐药基因armA的real-time PCR方法,并在粪便标本中进行了模拟实验。
在特异性评价中,不携带armA基因的菌株并未显示扩增曲线,说明该方法的特异性好。通过对荧光扩增曲线和标准曲线的分析发现,其对质粒标准品的检测下限为4.07×101拷贝/ml,表明本研究建立的方法灵敏度非常高。为了临床应用,制作了粪便模拟标本,通过对不同菌浓度的模拟标本的检测证实本方法的检测下限为1.3×104 cfu/ml。目前国内尚无armA耐药基因的荧光定量PCR检测技术的报道,该方法在临床实践中进一步验证后,有望在耐药基因监测中推广使用。
| [1] | Wang QT,Xu YC,Wang H,et al. Antimicrobial resistant characteristics in Enterococciand antibiotics for Enterococcal infections[J].Chinese Journal of Laboratory Medicine,1999,22(3):154-156.(in Chinese) 王清涛,徐英春,王辉,等. 肠球菌耐药现状调查及抗感染用药探讨[J]. 中华检验医学杂志,1999,22(3):154-156. |
| [2] | Shen YQ,Zhao M. Resistance mechanism of aminoglycoside antibiotics and strategy of controlling the drug resistance[J]. World Notes on Antibiotics,2002,23(3):118-120.(in Chinese) 沈依群,赵敏.氨基糖苷类抗生素的耐药机制及控制耐药性的策略[J].国外医药抗生素分册,2002,23(3):118-120. |
| [3] | Yamane K, Wachino J, Suzuki S, et al.16S rRNA methylase-producing,gram-negative pathogens, Japan[J].Emerg Infect Dis,2007,13(4):642-646. |
| [4] | Pan YF, Zhou H, Yu YS.The research advanceof 16S rRNA methylase conferring highlevel resistance to aminoglycosides[J].Chinese Journal of Laboratory Medicine,2007,30(6):699-701. (in Chinese) 潘韵峰,周华,俞云松. 导致高水平氨基糖苷类抗生素耐药的新型16SrRNA 甲基化酶基因研究进展[J]. 中华检验医学杂志,2007,30(6):699-701. |
| [5] | Karah N,Haldorsen B,Hermansen NO, et al. Emergence of OXA-carbapenemase-and16S rRNA methylase-producing international clones of Acinetobacter umannii in Norway[J]. J Med Microbiol,2011,60(4):515-521. |
| [6] | Gurung M, Moon DC, Tamang MD, et al.Emergence of 16S rRNA methylase gene armA and cocarriage of bla(IMP-1) in Pseudomonas aeruginosa isolates from South Korea[J].Diagn Microbiol Infect Dis,2010,68(4):468-470. |
| [7] | Galimand M,Courvalin P, Lambert T.Plasmid-mediated highlevel resistance toaminoglycosides in Enterobacteriaceaedue to 16S rRNA methylation[J]. Antimicrob Agents Chemother,2003,47(8):2565-2671. |
| [8] | Guo GJ, Lyu SF, Xiao YQ,et al. The research advance of detection methods of Pseudorabies virus[J]. Progress in Veterinary Medicine,2011,32(12):84-88.(in Chinese) 郭广君,吕素芳,肖跃强,等.猪伪狂犬病毒检测方法研究进展[J].动物医学进展,2011,32(12):84-88. |
| [9] | Zheng M,Mao N, Huang MQ, et al.Establishment and application of TaqMan-MGB fluorescence quantitative PCR for detection of Pseudorabies virus[J].Chinese Agricultural Science Bulletin,2013,29(17):37-41. (in Chinese) 郑敏,毛凝,黄梅清,等.猪伪狂犬病毒TaqMan-MGB荧光定量PCR检测方法的建立及应用[J].中国农学通报,2013,29(17):37-41. |
| [10] | Wang P, Hu Y, Yang HR, et al. Multiplex fluorescent real-time PCR for detection of methicillin-resistance gene and enterotoxin gene A in Staphylococcus aureus[J]. Disease Surveillance,2011,26(12):999-1003. (in Chinese) 王娉,胡玥,杨海荣,等.多重实时荧光定量聚合酶链反应检测携带耐药基因及肠毒素A基因的金黄色葡萄球菌[J].疾病监测,2011,26(12):999-1003. |
| [11] | Wang YD, He LH, Zhang JZ, et al.Establishment of multiplex real-time PCR assay to detect AIDS-assoiated Mycoplasmas[J]. Disease Surveillance,2013,28(2):148-152. (in Chinese) 王艳东,何利华,张建中,等.艾滋病相关支原体多重实时荧光定量荧光聚合酶链式反应检测方法的初步建立[J].疾病监测,2013,28(2):148-152. |
| [12] | Xu HQ, Wang Y, Wang Y, et al.Novel multiplex real-time TaqMan PCR assay for detection of Listeria ivanovii in simulated fecal samples[J]. Disease Surveillance,2013,29(3):228-233. (in Chinese) 许华青,王艳,王毅,等.模拟粪便标本单增李斯特菌和伊氏李斯特菌TanMan双重实时荧光定量-聚合酶链反应检测方法[J].疾病监测,2013,29(3):228-233. |
| [13] | Wu R, Zhang L, Dai JH, et al. Study on the determination and its influence of 16S rRNA methylase gene in gram negative bacteria[J].Laboratory Medicine,2010,25(6):423-426.(in Chinese) 吴蓉,张隆,戴俊华,等.革兰阴性杆菌16S rRNA甲基化酶基因检测及作用研究[J].检验医学,2010,25(6):423-426. |
| [14] | Gonzalez-zorn B, Catalan A, Escudero JA, et al. Geneticbasis for dissemination of armA [J]. J Antimicrob Chemother,2005, 56(3):583-585. |
| [15] | Jin D, Yu B, Ye CY, et al. TaqMan real-time PCR detection of Enterococcus faecium[J].Chinese Journal of Zoonoses,2013,29(6):551-555.(in Chinese) 金东,于波,叶长芸,等. 屎肠球菌TaqMan 荧光定量PCR 检测方法的研究[J]. 中国人兽共患病学报,2013,29(6):551-555. |
 2014, Vol. 29
2014, Vol. 29


