扩展功能
文章信息
- 刘彩霞, 严春, 李方去, 张盼
- LIU Cai-xia, YAN Chun, LI Fang-qu, ZHANG Pan
- 儿科耐碳青霉烯类肠杆菌科细菌携带β-内酰胺酶基因的调查
- Survey of the genotypes of beta-lactamases carried by carbapenems resistant Enterobacteriaceae isolated from children
- 疾病监测, 2015, 30(1): 67-70
- Disease Surveillance, 2015, 30(1): 67-70
- 10.3784/j.issn.1003-9961.2015.01.017
-
文章历史
- 收稿日期:2014-07-30
2. 温州市儿童医院, 浙江 温州325000
2. Wenzhou Children's Hospital, Wenzhou 325000, Zhejiang, China
肠杆菌科细菌分布广泛,是社区获得性感染和院内感染的重要病原菌,容易引起肺炎、脓毒症、尿路感染等。碳青霉烯类抗菌药物是目前临床治疗多重耐药肠杆菌科细菌引起感染的最有效的抗菌药,但随着该类药物的广泛应用及不合理使用,对该类药物耐药的肠杆菌科细菌(carbapenem-resistant enterobacteriaeae,CRE)的分离率呈逐渐上升趋势。根据中国CHINET细菌耐药性监测显示,2005年肠杆菌科细菌对碳青霉烯类抗菌药物的耐药率为0~3%,至2012年上升到4.4%~6.3%[1],给临床抗感染治疗带来极大挑战。虽然近10年来CRE感染已在成年人引起局部流行和暴发[2, 3, 4],但有关儿童CRE感染的文献较少。本研究对温州医科大学附属第二医院儿科分离的CRE菌株进行β-内酰胺酶基因携带情况和其耐药情况进行检测分析,为临床合理使用抗菌药物提供借鉴。
1 材料与方法 1.1 菌株来源2011年1月至2012年12月温州医科大学附属第二医院儿科分离到的CRE菌株,同一患者的分离株不重复留取。
1.2 主要仪器和试剂VITEK 2 COMPACT全自动微生物分析仪及其配套的GN+卡和GN13卡(法国生物梅里埃公司),PCR仪(德国Eppendorf公司),引物由上海生工生物工程技术服务有限公司合成,PCR试剂盒购自大连宝生生物工程有限公司。
1.3 方法 1.3.1 菌株鉴定及药敏所有菌株均经VITEK 2 COMPACT全自动微生物分析仪及配套卡片进行鉴定和药敏。可疑CRE菌株均经K-B法验证,同时进行改良Hodge试验。
1.3.2 菌株产碳青霉烯酶检测按照CLSI推荐的方法,用改良Hodge试验检测菌株产碳青霉烯酶情况。将0.5麦氏比浊度的大肠埃希菌ATCC25922进行10倍稀释后均匀涂布在MH平板表面,在平板的中心贴厄他培南纸片,然后将待测菌沿纸片边缘向平板边缘划线。将平板置35 ℃孵箱中培养16~18 h观察结果。若抑菌圈和接种线之间出现矢状线则为阳性结果,表明该菌株产碳青霉烯酶。
1.3.3 β-内酰胺酶基因的PCR扩增检测 1.3.4 核酸提取采用煮沸裂解法。挑取单个纯菌落置入内含250 μl双蒸水的0.5 ml ependorf(EP)管内,置沸水中10 min,取出后立即置冰浴盒中放置10 min,然后13 000 r/min离心5 min,上清液即为提取出的核酸DNA。
1.3.5 引物序列参照文献[4, 5, 6, 7]合成以下各种PCR检测用引物序列,见表 1。
| 基因型 | 引物序列(5′~3′) | 产物长度(bp) |
| KPC | F:CGG CTT GCT GGA CAC AC R:TTG ACG CCC AAT CCC TCG | 912 |
| IMP-1 | F:ACG GGC GGA ATA GAG TGG R:TAA CCG CCT GCT CTA ATG T | 397 |
| IMP-2 | F:GTT TTA TGT GTA TGC TTC C R:AGT TAC TTG GCT GTG ATG GT | 724 |
| VIM-1 | F:ATG GTG TTT GGT CGC ATA TC R:TGG GCC ATT CAG CCA GAT C | 920 |
| VIM-2 | F:AAA GTT ATG CCG CAC TCA CC R:TGC AAC TTC ATG TTA TGC CG | 865 |
| TEM | F:TTT CCG TGT CGC CCT TAT R:GCA ACT TTA TCC GCC TCC | 630 |
| SHV | F:TCT CCC TGT TAG CCA CCC TG R:CCA CTG CAG CAG CTG CCG TT | 593 |
| CTXM-1 | F:ATT AGA GCG GCA GTC GG R:CCA CAA CCC AGG AAG CAG | 587 |
| CTXM-2 | F:ACG CTA CCC CTG CTA TT R:TGT GCC CGC TGA GTT TC | 581 |
| CTXM-9 | F:ATG GTG ACA AAG AGA GTG CA R:CCC TTC GGC GAT GAT TCT C | 870 |
| NDM-1 | F:GGT TTG GCG ATC TGG TTT TC R:CGG AAT GGC TCA TCA CGA TC | 754 |
整个扩增体系为25 μl,扩增参数为94 ℃预变性5 min,然后94 ℃解链30 s,51~60 ℃(根据引物不同选择合适的退火温度)退火30 s,72 ℃延伸1 min,进行30个循环,最后72 ℃再延伸10 min。
1.3.7 产物检测产物在1.2%的琼脂糖凝胶中电泳30 min(200 V,75 mA),再用凝胶成像系统观察并记录。
2 结果 2.1 菌株分布共分离13株CRE,6株阴沟肠杆菌,3株产酸克雷伯菌,3株肺炎克雷伯菌,1株产气肠杆菌。其中8株来源于儿童重症监护室(ICU),2株来源于神经内科,1株来源于肾内科,1株来源于消化内科,1株来源于抢救室。
2.2 药敏情况13株CRE菌株对常用的β-内酰胺类及其与酶抑制剂复方制剂(包括氨苄西林、头孢唑啉、头孢呋辛、头孢噻肟、头孢曲松、头孢吡肟、氨曲南、阿莫西林/棒酸、氨苄西林/舒巴坦、哌拉西林、哌拉西林/他唑巴坦、替卡西林/棒酸、厄他培南)均耐药,对复方新诺明耐药率100%,对阿米卡星的耐药率最低,为7.69%,其次为妥布霉素15.38%,但细菌对该药物尚有23.08%的中介情况。环丙沙星和左旋氧氟沙星的耐药率为38.46%,庆大霉素为69.23%,具体药敏结果见表 2。
| 抗菌药物 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
| 阴沟 | 阴沟 | 阴沟 | 阴沟 | 阴沟 | 产酸 | 肺克 | 产酸 | 阴沟 | 肺克 | 产酸 | 产气 | 肺克 | |
| 注:S:敏感;R:耐药,I:中介。 | |||||||||||||
| 丁胺卡那 | S | S | S | S | S | S | S | S | S | R | S | S | S |
| 庆大霉素 | R | R | R | R | R | S | R | S | R | R | S | S | R |
| 环丙沙星 | S | S | S | S | S | R | R | R | S | R | S | S | R |
| 左旋氧氟沙星 | S | S | S | S | S | R | R | R | S | R | S | S | R |
| 妥布霉素 | I | S | S | S | I | S | R | S | I | S | S | S | R |
| 亚胺培南 | R | R | R | R | R | R | R | I | I | R | R | R | R |
| 厄他培南 | R | R | R | R | R | R | R | R | R | R | R | R | R |
Hodge试验结果显示共有11株菌阳性,阳性率84.62%,提示这些菌株产碳青霉烯酶。
2.4 PCR扩增结果13株菌共扩增出6种β-内酰胺酶基因,分别为TEM、KPC、SHV、CTXM-1、CTXM-9、VIM-1型基因,阳性率分别为100%、84.62%、46.15%、38.46%、15.38%和7.69%,而VIM-2、IMP-1、IMP-2、CTXM-2、NDM-1均未检出。各菌株的β-内酰胺酶基因携带情况见表 3。各种基因PCR扩增阳性结果见图 1、2。
| 基因型 | 标本号 | 阳性菌株数 | 阳性率(%) | ||||||||||||
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | |||
| TEM | + | + | + | + | + | + | + | + | + | + | + | + | + | 13 | 100.00 |
| 注:(1) “+”表示阳性;“-”表示阴性。 | |||||||||||||||
| KPC | + | + | + | + | + | + | + | - | + | + | - | + | + | 11 | 84.62 |
| SHV | - | - | - | - | - | - | + | + | + | + | + | - | + | 6 | 46.15 |
| CTXM-1 | - | - | - | - | - | + | - | + | + | - | + | + | + | 6 | 46.15 |
| CTXM-9 | - | - | - | - | - | + | - | - | - | - | - | - | + | 2 | 15.38 |
| CTXM-2 | - | - | - | - | - | - | - | - | - | - | - | - | - | 0 | 0.00 |
| VIM-1 | - | - | - | - | - | - | - | - | - | - | - | + | - | 1 | 7.69 |
| VIM-2 | - | - | - | - | - | - | - | - | - | - | - | - | - | 0 | 0.00 |
| IMP-1 | - | - | - | - | - | - | - | - | - | - | - | - | - | 0 | 0.00 |
| IMP-2 | - | - | - | - | - | - | - | - | - | - | - | - | - | 0 | 0.00 |
| NDM-1 | - | - | - | - | - | - | - | - | - | - | - | - | - | 0 | 0.00 |
| Hodge试验 | + | + | + | + | + | + | + | - | + | + | - | + | + | 11 | 84.62 |
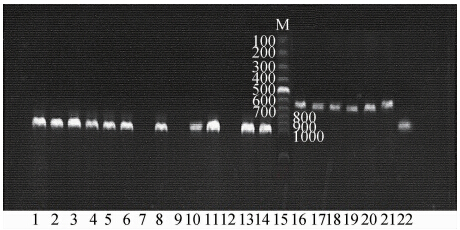
|
| 注:1~14泳道为blaKPC基因扩增产物,其中7号泳道为阴性对照(大肠埃希菌ATCC25922); 15泳道为100 bp的Marker;16~21泳道为blaSHV基因扩增产物; 22泳道为blaVIM-1基因扩增产物。 图 1 blaKPC、blaSHV和blaVIM-1基因扩增产物电泳结果 Fig. 1 Agarose gel electrophoresis of PCR products of blaKPC,blaSHV and blaVIM-1 |

|
| 注:1~14泳道为blaTEM基因扩增产物,其中7号泳道为阴性对照(大肠埃希菌ATCC25922); 15泳道为100 bp的Marker; 16~21泳道为blaCTXM-1基因扩增产物; 22~23泳道blaCTXM-9基因扩增产物。 图 2 blaTEM、blaCTXM-1和blaCTXM-9基因扩增产物电泳结果 Fig. 2 Agarose gel electrophoresis of PCR products of blaTEM,blaCTXM-1 and blaCTXM-9 |
Logan[8]综合统计了5个国家报道的63例儿童CRE感染病例,CRE对氨苄西林/舒巴坦、哌拉西林/他唑巴坦、复方新诺明的耐药率分别为100%、82%和68%。北京儿童医院报道的46株CRE菌株对头孢哌酮/舒巴坦、哌拉西林/他唑巴坦和环丙沙星的耐药率分别为87.00%、69.60%和26.00%[9]。本组资料显示13株CRE菌株对常用的β-内酰胺类抑制剂及其与酶抑制剂复方制剂均耐药,对复方新诺明100%耐药,对阿米卡星的耐药率最低,为7.69%,其次为妥布霉素15.38%,但细菌对该药物尚有23.08%的中介情况。环丙沙星和左旋氧氟沙星的耐药率为38.46%,庆大霉素为69.23%,较国内外其他地区分离株耐药性强。
Little等[10]检测了西雅图儿童医院20022010年分离的6株CRE,携带的耐药基因包括blaKPC、blaIMP、blaSME,同时检测到1株携带blaCTXM-15伴ompK36基因缺陷致孔蛋白改变的CRE。而北京儿童医院的CRE主要携带的是IMP型金属酶[9]。温州医科大学附属第二医院儿科分离的CRE主要产KPC型碳青霉烯酶,但还同时携带1~3种其他β-内酰胺酶基因,可能与该院分离的CRE菌株表现出对β-内酰胺类抗菌药物高度耐药有关。本次实验发现所有改良Hodge试验阳性的CRE菌株均携带blaKPC,说明改良Hodge试验与产KPC型碳青霉烯酶的菌株之间有良好的相关性。改良Hodge试验阴性,blaKPC检测阴性的2株产酸克雷伯菌,均同时携带blaTEM、blaSHV 和blaCTXM-1三种耐药基因,它们对碳青霉烯类抗菌药物耐药机制中可能还存在外膜蛋白的缺失或改变,导致膜通透性降低,从而仅引起菌株对碳青霉烯类抗菌药物的敏感性降低[10]。当然也有可能存在未检测或未知的其他酶基因。在本组数据中有5株阴沟肠杆菌(标本1~5)均分离至儿童ICU,其耐药谱和携带的耐药基因型均相近,可能是同一克隆株,有待进一步研究。
总之,CRE因其高传播率、高死亡率、高耐药率已成为全球住院患者面临的严重威胁,美国CDC于2012年6月发布了针对CRE感染的防控指南[11],2013年,又将CRE列为危险级别为紧迫、需采取紧急行动应对的3种病原微生物之一[12]。由于目前治疗手段匮乏,积极筛查和有效的院感防控是防止CRE蔓延的重要手段。微生物检验工作者与感染管理科、临床医生的积极有效沟通是必要的。
| [1] | Wang F, Zhu DM, Hu FP, et al.2012 CHINET surveillance of bacterial resistance in China[J].Chinese Journal of Infection and Chemotherapy,2013,13(5):321-330.(in Chinese) 汪复,朱德妹,胡付品,等. 2012年中国CHINET细菌耐药性监测[J]. 中国感染与化疗杂志,2013,13(5):321-330. |
| [2] | Bratu S, Brooks S, Burney S, et al. Detection and spread of Escherichia coli possessing the plasmid-borne carbapenemase KPC-2 in Brooklyn, New York[J]. Clin Infect Dis,2007,44(7):972-975. |
| [3] | Lomaestro BM, Tobin EH, Shang W, et al. The spread of Klebsiella pneumoniae carbapenemase-producing K. pneumoniae to upstate New York[J]. Clin Infect Dis,2006,43(3):e26-e28. |
| [4] | Navon-Venezia S, Chmelnitsky I, Leavitt A, et al. Plasmid-mediated imipenem-hydrolyzing enzyme KPC-2 among multiple carbapenem-resistant Escherichia coli clones in Israel[J]. Antimicrob Agents Chemother,2006,50(9):3098-3101. |
| [5] | Zhang X, Song SD. The mechanism of two clinical isolates of Enterobacter cloacae resistant to Carbopenems[J].Chinese Journal of Antibiotics,2011,36(4):303-306.(in Chinese) 张肖,宋诗铎.2株阴沟肠杆菌临床分离株对碳青霉烯类抗生紊耐药机制的研究[J].中国抗生素杂志2011,36(4):303-306. |
| [6] | Hisaaki N, Masaru K. Metallo-lactamase-producing gram-negative bacilli:laboratory-based surveillance in cooperation with 13 clinical laboratories in the Kinki Region of Japan[J]. J Clin Microbiol,2004,42(11):5256-5263. |
| [7] | Nordmann P, Poirel L, Carrër A,et al. How to detect NDM-1 producers[J]. J Clin Microbiol,2011,49(2):718-721. |
| [8] | Logan LK. Carbapenem-resistant enterobacteriaceae:an emerging problem in children[J]. Clin Infect Dis,2012,55(6):852-859. |
| [9] | Dong F, Xu XW,Song WQ,et al. Characterization of antibiotic resistance and cargapenemase-producing in carbapenem no-susceptibility Enterobacteriaceae from pediatric patients[J].Chinese Journal of Microbiology and Immunology,2011,31(9):787-791.(in Chinese) 董方,徐樨巍,宋文琪,等. 儿科患者中对碳青霉烯类不敏感肠杆菌科细菌耐药性和耐药基因的研究[J]. 中华微生物学和免疫学杂志,2011,31(9):787-791. |
| [10] | Little ML, Qin X, Zerr DM, et al. Molecular diversity in mechanisms of carbapenem resistance in paediatric Enterobacteriaceae[J]. Int J Antimicrob Agents,2012,39(1):52-57. |
| [11] | CDC. 2012 CRE Toolkit-Guidance for Control of Carbapenem-resistant Enterobacteriaceae (CRE)[EB/OL].(2012-01-01)[2014-05-22]. http://www.cdc.gov/hai/organisms/cre/cre-toolkit/. |
| [12] | CDC. Antibiotic resistance threats in the United States[EB/OL].(2013-01-01)[2014-05-20]. http://www.cdc.gov/drug resistance/threat-report-2013/. |
 2015, Vol. 30
2015, Vol. 30


