扩展功能
文章信息
- 顾一心, 陶晓霞, 刘立雍, 张建中, 赵飞
- GU Yi-xin, TAO Xiao-xia, LIU Li-yong, ZHANG Jian-zhong, ZHAO Fei
- TaqMan-MGB探针实时荧光定量-聚合酶链反应检测肺炎支原体方法的建立
- Establishment of TaqMan-MGB real-time PCR assay to detect Mycoplasma pneumoniae
- 疾病监测, 2015, 30(3): 236-239
- Disease Surveillance, 2015, 30(3): 236-239
- 10.3784/j.issn.1003-9961.2015.03.017
-
文章历史
- 收稿日期:2014-12-24
肺炎支原体是引起人类呼吸道感染的重要病原菌之一,每年约有10%~40%社区获得性肺炎是由其感染引起的[1, 2]。以实时荧光定量-聚合酶链反应(real-time PCR)为代表的快速核酸检测技术,凭借其快速、特异、灵敏的特点逐步成为肺炎支原体检测的“金标准”而被广泛应用。目前已报道或应用的肺炎支原体real-time PCR方法有多种,其检测灵敏度和特异度均较好,检测限一般在几个至十几个cfu[3, 4]。上述检测肺炎支原体方法的靶基因均为单拷贝的保守基因,研究表明,肺炎支原体基因中存在许多重复序列[5](包括RepMP1 a-n,RepMP23 a-j,RepMP4 a-h,RepMP5 a-j),这些重复序列在肺炎支原体中都是多拷贝。理论上说,以多拷贝重复序列基因作为靶序列检测可将相同条件下荧光PCR检测灵敏度相应提高几倍至十几倍。但是目前由于肺炎支原体的这些重复序列并非完全一致,只是部分序列一致,且重复序列的稳定性也没有通过大量的测序数据进行验证。因此,对于重复序列中寻找保守且稳定的靶序列,并设计探针引物存在巨大困难。因此,课题组通过对前期大量测序数据比对,希望寻找出RepMP序列中的保守区域,在此多拷贝区域设计 real-time PCR检测体系,力求在现有水平上进一步提高将肺炎支原体检测能力。
1 材料与方法 1.1 菌株肺炎支原体ATCC39505、ATCC29342、ATCC29343、ATCC15531、ATCC29085、ATCC15492、ATCC29894、ATCC15293,人型支原体ATCC23114,生殖支原体ATCC33530,解脲脲原体ATCC27618,穿透支原体ATCC55252,梨支原体ATCC25960,发酵支原体ATCC19989,猪鼻支原体ATCC17981,唾液支原体ATCC23064,口腔支原体ATCC23714,咽喉支原体ATCC25293,金黄色葡萄球菌ATCC29213,鲍曼不动杆菌ATCC17978,A族链球菌ATCC700294,均购自美国ATCC菌种保存库并由传染病所诊断室保存;肺炎衣原体、结核分枝杆菌、流感嗜血杆菌、脑膜炎双球菌、嗜肺军团菌、肺炎克雷伯菌、大肠埃希菌、铜绿假单胞菌、草绿色链球菌、肺炎链球菌,均为中国疾病预防控制中心传染病预防控制所其他科室惠赠,所有菌株均使用QIAamp DNA Mini Kit (QIAGEN 51306) 试剂盒提取核酸。
1.2 主要试剂和仪器Platinum Taq DNA polymerase和Platinum quantitative PCR SuperMix-UDG为Invitrogen公司产品;Nucleotide mix和Nuclease-free water为Promega公司产品;QIAamp DNA Mini Kit为QIAGEN公司产品;Real-time PCR仪C1000 Thermal Cycler为BIO RAD公司仪器。
1.3 引物与探针比对课题组前期20株肺炎支原体全基因测序株与NCBI数据库已报道的肺炎支原体所有RepMP基因序列,确认一段在8个RepMP23重复序列中的保守区域,使用Beacon Designer 7.0软件设计特异性扩增引物RepMP23-F(5′-GTG GAT GAT ATA ACC GCG CC-3′)和RepMP23-R(5′-GAA CGC GAA CCA CTT GTG TTC-3′)及TaqMan-MGB荧光探针RepMP23-P 。引物和探针由Invitrogen公司合成。
1.4 TaqMan-MGB real-time PCR检测体系与扩增条件优化对体系镁离子浓度、引物和探针浓度以及退火温度进行了梯度优化,最终优化的25 μl 检测体系包含: 0.25 μl Platinum Taq DNA polymerase (Invitrogen C10966-034),12.5 μl Platinum quantitative PCR SuperMix-UDG (Invitrogen C11730-025),1.0 μl MgCl2(50 mmol/L),0.3 μl上游引物RepMP23-F(25 μmol/L),0.3 μl下游引物RepMP23-R(25 μmol/L),0.2 μl探针RepMP23-P(25 μmol/L),1.5 μl PCR nucleotide mix (PromegaC1141),3.0 μl DNA模板,用Nuclease-free water (Promega P1193)补至25 μl。混合体系使用BIO-RAD C1000 Thermal Cyclerreal-time PCR仪进行扩增。扩增条件为:95 ℃ 2 min;然后95 ℃ 15 s,59 ℃ 15 s,45个循环。
1.5 TaqMan-MGB real-time PCR体系灵敏度及特异度检测对已优化的体系使用8株肺炎支原体ATCC标准株(详见1.1菌株清单)和50株临床分离株核酸验证其检测灵敏度;另外使用10种其他支原体种属标准菌株核酸和13种呼吸道常见致病菌核酸及人类染色体(详见1.1菌株清单)进行体系特异度验证。
1.6 TaqMan-MGB real-time PCR体系检测效率及检测限测定本使用含0.162 fg/μl~1.62 ng/μl的肺炎支原体核酸对优化后体系进行标准曲线建立,评价该方法的检测效率及检测限。real-time PCR扩增效率(E)为每个循环扩增模板的百分比,计算公式为:E=(10-1/斜率-1)×100%。检测限为方法能检测到的最低分析物浓度,是检测方法的重要性能指标之一。
1.7 TaqMan-MGB real-time PCR与MpP1 real-time PCR[6]两种方法检测限比较使用含0.162 fg/μl~1.62 ng/μl的肺炎支原体核酸对TaqMan-MGB real-time PCR与MpP1 real-time PCR两种方法分别建立标准曲线,每种检测方法均使用各浓度梯度模板3 μl扩增,比较两种方法的理论检测限。
2 结果 2.1 RepMP23保守区域靶位点筛选与TaqMan-MGB real-time PCR体系优化通过比对课题组前期20株肺炎支原体全基因测序株与NCBI数据库已报道的肺炎支原体所有RepMP基因序列,分别筛选到一段在8个RepMP23重复序列中的保守区域和一段在6个RepMP4重复序列中的保守区域,共设计了4套检测引物和探针,通过对各体系镁离子浓度、引物和探针浓度以及退火温度的梯度优化,最终以RepMP23重复序列为靶基因建立了本研究报道的检测方法。
2.2 TaqMan-MGB real-time PCR体系的灵敏度和特异度使用该方法对8株ATCC标准株和50株临床分离株共计58株肺炎支原体核酸检测结果均为阳性,因此在使用肺炎支原体纯菌模板情况下体系的检测灵敏度为100%。用该体系对10种其他种属支原体(生殖支原体、人型支原体、解脲脲原体、猪鼻支原体、穿透支原体、梨支原体、发酵支原体、唾液支原体、口腔支原体、咽喉支原体)以及13种常见呼吸道病原菌(肺炎衣原体、肺炎链球菌、鲍曼不动杆菌、A族链球菌、金黄色葡萄球菌、流感嗜血杆菌、结核分枝杆菌、脑膜炎双球菌、嗜肺军团菌、肺炎克雷伯菌、大肠埃希菌、铜绿假单胞菌、草绿色链球菌)及人类染色体进行扩增,结果均为阴性,检测特异度为100%。
2.3 TaqMan-MGB real-time PCR体系的扩增效率与检测限评价使用含0.162 fg/μl~1.62 ng/μl的肺炎支原体核酸扩增获得标准曲线及对数曲线,见图1,标准曲线方程Y=-3.371x+36.343,斜率为-3.371,R2=0.999,经公式计算该体系扩增效率E=98.0%。该方法使用3 μl模板进行检测,标准品中的肺炎支原体核酸量实际约为0.5 fg~5 ng,经过换算约为0.2~2×106cfu的肺炎支原体(1 cfu肺炎支原体菌株对应的核酸量约为2.7 fg~5.3 fg核酸)。该方法对肺炎支原体核酸最低检测限为0.2 cfu(对应核酸量为0.5 fg),相应Ct值为38.14,故取整将此方法结果判定cut off值定为Ct值38,检测Ct值>38的标本为阴性,≤38的标本为阳性结果。2.4 TaqMan-MGB real-time PCR与MpP1 real-time PCR两种方法检测限比较 通过肺炎支原体标准浓度核酸扩增,TaqMan-MGB real-time PCR与MpP1 real-time PCR两种方法的Ct值与模板浓度的对数值具有很好的对应关系,但TaqMan-MGB real-time PCR方法对于相同模板的检测Ct值均小于MpP1 real-time PCR方法,且对于0.5 fg核酸仅TaqMan-MGB real-time PCR体系能够检测(图2,表1)。
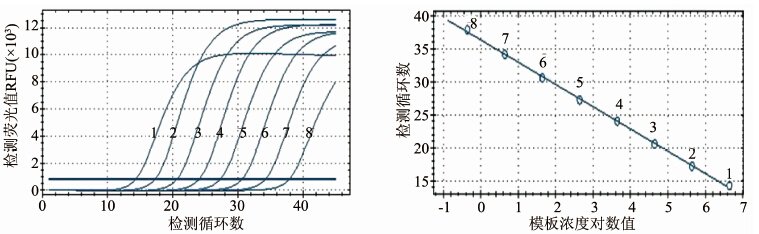
|
| 图1 TaqMan-MGB荧光PCR体系对标准浓度梯度核酸的扩增曲线(左)及模板浓度对数曲线(右) Fig.1 Standard curve (left) and log standard curve (right) of TaqMan-MGB real-time PCR assay for standard concentration M. pneumoniae DNA 注:1:5×106fg; 2:5×105fg; 3:5×104fg; 4:5×103fg; 5:5×102fg; 6:5×10 fg; 7:5 fg; 8:0.5 fg。 |

|
| 图2 TaqMan-MGB荧光PCR(右)与MpP1荧光PCR(左)两种方法检测限比较 Fig.2 Detection Limitation of TaqMan-MGB real-time PCR assay (right) and MpP1 real-time PCR assay (left) for standard concentration M. pneumoniae DNA 注:1:5×106 fg; 2:5×105 fg; 3:5×104 fg; 4:5×103 fg; 5:5×102 fg; 6:5×10 fg; 7:5 fg; 8:0.5 fg。 |
| 标准品浓度(fg) | 标准品检测Ct值 | |
| TaqMan-MGB real-time PCR | MpP1 real-time PCR | |
| 注:(1)Neg为检测阴性。 | ||
| 0.5 | 38.14 | Neg(1) |
| 5 | 34.40 | 38.56 |
| 5×101 | 30.88 | 35.89 |
| 5×102 | 27.49 | 32.16 |
| 5×103 | 24.25 | 29.42 |
| 5×104 | 20.87 | 25.47 |
| 5×105 | 17.46 | 23.00 |
| 5×106 | 14.51 | 18.73 |
目前报道的肺炎支原体TaqMan探针real-time PCR方法的检测限一般为几个cfu至十几个cfu之间[3, 4, 6]。为寻求更加灵敏特异的检测效果,笔者从肺炎支原体菌株基因组中寻找突破。在肺炎支原体基因组中存在着4种重复序列[7, 8]:RepMP1 a-n,RepMP23 a-j,RepMP4 a-h,RepMP5 a-j。以多拷贝重复序列基因作为靶序列检测可将相同条件下的模板数量放大几倍至十几倍,进而提高相同检测技术的检测限和检测灵敏度。通过序列比对发现,肺炎支原体的每种重复序列并非高度一致,只是部分序列一致,且重复序列的稳定性也没有通过大量的测序数据进行验证。因此,对于重复序列中寻找保守且稳定的靶序列,并设计探针引物存在巨大困难。本研究通过前期20株肺炎支原体全基因组测序株数据与NCBI数据库中的RepMP序列比对,寻找到相对保守且在多株菌株中没有突变的92 bp RepMP23区域。由于序列很短,此片段中无法设计出符合扩增要求Tm值的普通TaqMan探针,因此研究采用了TaqMan-MGB探针。通过在探针中添加了MGB分子,可有效提高探针Tm值,使探针的长度缩短,提高结合效率,使无法设计普通TaqMan探针的短序列中获得TaqMan-MGB探针。由于TaqMan-MGB探针在3′端的Quencher基团为不发光荧光基团[9],且与报告基团在空间位置更接近,因此可降低非特异性荧光背景,进一步提高敏感性,同时MGB探针对检出序列要求更保守。
本研究建立的TaqMan-MGB real-time PCR方法标准曲线的R2=0.999,数据线性程度非常好,依此计算的体系扩增效率为98.0%[10],显示出良好的检测能力和稳定性。本研究使用58株肺炎支原体菌株模板和24种非肺炎支原体模板验证了该方法灵敏度和特异度在检测纯菌核酸时均达到100%,且检测限仅为0.2 cfu,比同类real-time PCR检测方法灵敏约一个数量级。这归功于本方法将目的靶基因设计到多拷贝的保守重复序列区域,相同条件下将模板量提高了接近10倍。对于检测相同含量的肺炎支原体核酸,TaqMan-MGB real-time PCR方法的Ct值比MpP1 real-time PCR方法小4个数值左右(表1),有效提高了检测限和检测灵敏度,尤其是对于带菌量较低的标本,将更有利于提高标本的检出率,降低假阴性结果。此外,TaqMan-MGB探针相比普通TaqMan探针对于检测靶基因更加特异,探针区域任何单碱基的不匹配均不会扩增,进一步降低了假阳性扩增结果。因此,此TaqMan-MGB方法相比普通TaqMan real-time PCR方法在灵敏度、特异度和检测限上都有具有明显优势,有望成为临床上肺炎支原体感染快速检测的新技术。
| [1] | Waites KB, Talkington DF. Mycoplasma pneumoniae and its role as a human pathogen[J]. Clin Microbiol Rev,2004,17(4):697-728. |
| [2] | Hammerschlag MR. Mycoplasma pneumoniae infections[J]. Curr Opin Infect Dis,2001,14(2):181-186. |
| [3] | Daxboeck F, Khanakah G, Bauer C, et al. Detection of Mycoplasma pneumoniae in serum specimens from patients with mycoplasma pneumonia by PCR[J]. Int J Med Microbiol,2005,295(4):279-285. |
| [4] | Winchell JM, Thurman KA, Mitchel SL, et al. Evaluation of three real-time PCR assays for detection of Mycoplasma pneumoniae in an outbreak investigation[J]. J Clin Microbiol,2008,46(9):3116-3118. |
| [5] | Himmelreich R, Hilbert H, Plagens H, et al. Complete sequence analysis of the genome of the bacterium Mycoplasma pneumoniae[J]. Nucl Acids Res,1996,24(22):4420-4449. |
| [6] | Zhao F, Cao B, He LH, et al. Evaluation of a new real-time PCR assay for detection of Mycoplasma pneumoniae in clinical specimens[J]. Biomed Environ Sci,2012,25(1):77-81. |
| [7] | Musatovova O, Kannan TR, Baseman JB. Mycoplasma pneumoniae large DNA repetitive elements RepMP1 show type specific organization among strains[J]. PLoS One,2012,7(10):e47625. |
| [8] | Spuesens EBM, van de Kreeke N, Estevão S, et al. Variation in a surface-exposed region of the Mycoplasma pneumoniae P40 protein as a consequence of homologous DNA recombination between RepMP5 elements[J]. Microbiology,2011,157(Pt 2):473-483. |
| [9] | Kutyavin IV, Afonina IA, Mills A, et al. 3'-minor groove binder-DNA probes increase sequence specificity at PCR extension temperatures[J]. Nucleic Acids Res,2000,28(2):655-661. |
| [10] | Peccoud J, Jacob C. Theoretical uncertainty of measurements using quantitative polymerase chain reaction[J]. Biophysical J,1996, 71(1):101-108. |
 2015, Vol. 30
2015, Vol. 30


