扩展功能
文章信息
- 陈玉红, 李美蓉, 陆红云, 张红波, 吴绍武, 刘雪莲, 谢海珍, 钟玲
- CHEN Yu-hong, LI Mei-rong, LU Hong-yun, ZHANG Hong-bo, WU Shao-wu, LIU Xue-lian, XIE Hai-zhen, ZHONG Ling
- 2013年宜春市甲型H1N1流感病毒分离鉴定及HA基因分子进化分析
- HA gene molecular evolution analysis of influenza A (H1N1) pdm09 virus in Yichun, 2013
- 疾病监测, 2016, 31(2): 101-105
- Disease Surveillance, 2016, 31(2): 101-105
- 10.3784/j.issn.1003-9961.2016.02.005
-
文章历史
- 收稿日期: 2015-01-20
流感病毒属正粘病毒科、分节段、负链单股RNA病毒,据核衣壳蛋白(NP)和基质蛋白(MP)的差异性可分为甲、乙、丙或A、B、C三型[1]。而甲型流感病毒根据血凝素(HA)和神经氨酸酶(NA)的抗原差异性,HA分为16个亚型,NA分为10个亚型[2]。甲型H1N1流感始于2009年3月美国和墨西哥暴发的流感大流行[3],该病毒为猪流感、禽流感及人流感三重重组病毒[4]。流感病毒极易发生变异,使病毒能逃逸宿主免疫识别和清除,导致新的流感暴发[5]。HA基因的变异频率最高,是流感病毒抗原性变异的基础。HA序列分析可研究关键抗原位点是否发生变异,评价毒株抗原性变异有无流行病学意义。本研究分析了江西省宜春市2013年甲型H1N1流感病毒HA基因序列分子变异及进化特征,为防控甲型H1N1流感大流行和常规监测提供科学依据。
1 材料与方法 1.1 标本来源采集2013年宜春市监测医院流感咽拭子标本及流感重症咽拭子标本。
1.2 病毒分离和效价测定按照世界卫生组织(WHO)推荐流感病毒分离方法[6],采用犬肾传代细胞(MDCK)培养病毒,病毒收获后采用微量血凝实验方法(HA)进行效价鉴定;将滴度≥1:8的毒株采用血凝抑制方法(HI)进行型别鉴定。所有毒株均经国家流感中心复核鉴定,具有统一编号。
1.3 毒株来源与RNA提取随机挑选11株分离到的甲型H1N1流感病毒毒株,采用美国Promega公司的Maxwell® 16 Instrument核酸自动提取仪及配套Maxwell® 16 Viral Total Nucleic Acid Purification Kit试剂盒提取病毒基因组RNA。
1.4 HA扩增与序列测定HA基因引物序列设计及RT-PCR反应条件参考WHO资料[6],采用QIAGEN One Step RT-PCR Kit试剂盒,以病毒基因组RNA为模板反转录扩增HA基因,收集PCR产物进行双向测序。HA基因序列引物合成及序列测定均委托上海生工生物工程技术服务有限公司完成。
1.5 HA序列分析以WHO北半球甲型H1N1流感疫苗推荐株A/California/07/2009(H1N1)(GenBank:CY121680),并另外选取几株国内外近几年分离到的流感甲型H1N1毒株的HA基因为参考序列,采用DNAStar 7.0和Mega 5.0软件对HA基因核苷酸序列及其编码的氨基酸序列进行同源性比对以及分子进化分析。
2 结果 2.1 病毒分离结果2013年宜春市全年共分离流感毒株82株,其中甲型H1N1分离到51株,占全年毒株分离的62.20%,是2013年流行优势株;其余甲型H2N3、乙型分别分离到23株(28.05%)、 8株(9.75%)。同时流感病毒毒株分离大多数分布在3月(19株)、4月(20株),少数分布在2月(4株)、5月(8株),说明2013年宜春市甲型H1N1流感呈春季集中暴发形势,其他季节未发现。
2.2 HA基因核苷酸和氨基酸同源性及分子进化分析甲型H1N1流感病毒参考株A/California/07/2009(H1N1)(GenBank:CY121680)的HA基因编码区核苷酸长度为1701 bp,编码566个氨基酸;序列分为信号肽(1~51 bp,编码1~17位氨基酸),重链区HA1(52~1032 bp,编码18~344位氨基酸)和轻链区HA2(1033~1701 bp,编码345~566位氨基酸)。
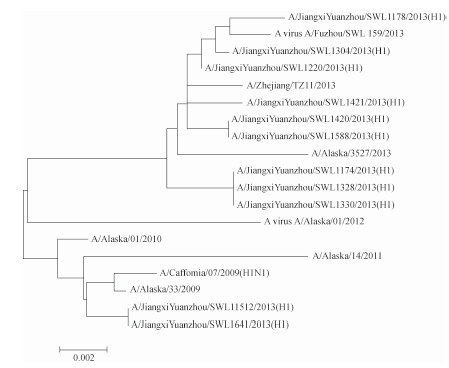
选取的11株甲型H1N1流感毒株HA基因序列经测定均为1632 bp,对应参考序列为69~1701 bp,编码24~566位氨基酸。经DNAStar 7.0、Mega 5.0软件分析,所测的11株毒株与参考株A/California/07/2009(H1N1)进行HA基因比对,其核苷酸序列同源性在98.30%~100.00%之间,平均为99.15%;氨基酸的同源性在97.70%~100.00%之间,平均为98.85%。另外参考几株国内外近几年分离到的流感病毒毒株与所测11株毒株进行HA基因氨基酸序列比对,并绘制分子进化树(图 1),可见除毒株A/Jiangxi Yuanzhou/SWL1641/2013(H1)和A/Jiangxi Yuanzhou/SWL11512/2013(H1)与国外毒株A/California/07/2009(H1N1)、A/Alaska/33/2009、A/Alaska/01/2010、A/Alaska/14/2011在同一分支上,而剩余A/Jiangxi Yuanzhou/SWL1178/2013(H1)等9株毒株与国内A virus A/FuZhou/SWL159/2013、 A/ZheJiang/TZ11/2013和国外毒株A/Alaska/3527/2013、A virus A/Alaska/01/2012在另一分支上。但因为分支单位小,仅为0.002,总体而言所测11株毒株与疫苗株亲缘关系较近,且近几年国内外甲型H1N1流感病毒HA序列没有发生较大变异。
|
| 图 1 2013年宜春市甲型H1N1流感病毒HA基因序列的种系进化树 Figure 1 Phylogenetic tree based on HA amino acid sequences of influenza A (H1N1) pdm09 virus isolated in Yichun, 2013 |
与参考氨基酸序列比对,所测11株HA基因编码24~566位氨基酸,共543个氨基酸,其中24~344位氨基酸为重链区HA1,345~566位氨基酸为轻链区HA2。如图 2所示,所测HA以第24位氨基酸为起始位点,与参考氨基酸序列24~566位氨基酸进行比对,对HA编码的氨基酸序列进行氨基酸变异和蛋白结构变异分析。所测11株HA氨基酸均发生P100S、S220T、I338V替换,9株均发生D114N、S202T、V251I、K300E、E391K、S468N、E516K替换,3株均发生N142D替换,2株均发生K136N、K180N、V289I替换,其余各有1株分别发生D34N、N142S、H143Y、G172E、I284L、V444I替换。

|
| 图 2 2013年宜春市甲型H1N1流感病毒HA氨基酸序列比对分析 Figure 2 comparative analysis based on HA amino acid sequences of influenza A (H1N1) pdm09 virus isolated in Yichun, 2013 |
二硫键通常指在肽和蛋白质分子中的半胱氨酸残基中的化学键。与参考氨基酸序列比对,所测HA编码的氨基酸序列中,第58、72、84、107、153、292、296、320、481、488、555、562、565位的半胱氨酸未出现变异,其他位点也均未出现新的半胱氨酸,HA氨基酸序列二硫键保持稳定。
2.3.2 HA糖基化位点的变异分析氨基酸潜在糖基化位点由糖苷键通过N糖苷键结合在Asn-X-Thr(N-X-T)或Asn-X-Ser(N-X-S)的Asn残基上形成[7]。与参考氨基酸序列比对,所测HA编码的氨基酸序列中,均在第27~29、28~30、40~42、104~106、293~295、304~306、498~500、557~559位的氨基酸发现8个潜在糖基化位点,且均未出现变异,其他位点也均未出现新的也未出现新的糖基化位点,HA氨基酸序列糖基化位点保持稳定。
2.3.3 HA受体结合位点(RBS)的变异分析HA受体结合位点(RBS)包括130环(135~138)、190螺旋(190~198)、220环(221~228)以及这些结构域附近的保守氨基酸[8]。与参考氨基酸序列比对,在所测HA氨基酸中,除有2株毒株在130环发生K136N替换,在130环附近分别有3株发生N142D替换、1株发生N142S替换、1株发生H143Y替换外,其余HA在190螺旋和220环结构域均未出现变异。
2.3.4 HA抗原决定簇的变异分析过去研究认为甲型流感病毒HA蛋白可分为A、B、C、D、E共5个抗原决定簇区,其中A区:133~137、140~146;B区:156~160、187~198;C区:52~54、275、277、278;D区:207、174;E区:63、78、81、83[9]。上述抗原决定簇氨基酸发生替换,一般均会引起HA蛋白抗原性漂移。本研究中通过抗原位点统计分析,与参考氨基酸序列比对,所测11株毒株HA氨基酸序列的HA1区,在A区第133~137位,有2株发生K136N替换;在A区第140~146位,分别有1株发生N142S替换、1株发生H143Y替换、3株发生N142D替换;其他抗原决定簇区未出现变异。
而2010年Xu等[10]分析表明甲型H1N1流感病毒HA蛋白共含有4个抗原决定簇(Sa、Sb、Ca和Cb),其中Sa、Sb与RBS接近,Ca位于亚基交界处,Cb位于痕迹酶作用区域。2014年Matsuzaki等[11]研究使用抗A/Narita/1/2009的16种类型抗HA单克隆抗体来检测599个甲型H1N1流感病毒逃逸突变体,以此来定位HA的抗原表位。结果表明Sa的突变位点为141~143、171~172、177和180;Sb的突变位点为170、173、202、206、210~212;Ca的突变位点为151、154、156~159、200和238;还有一个不确定的突变位点为147。本研究所测11株毒株HA氨基酸序列在Sa区有3株发生N142D替换,1株发生H143Y替换,1株发生G172E替换;在Sb区有9株发生S202T替换;其他抗原决定簇区未出现变异。
3 讨论流感病毒感染初期由表面膜蛋白HA与宿主细胞表面的HA受体结合并附着于细胞表面[12],再由病毒表面膜蛋白NA水解HA受体末端N-乙酰神经氨酸破坏受体,帮助病毒在宿主体内扩散[13]。HA是流感病毒的主要抗原,HA变异是流感变异的分子基础。因而对甲型H1N1病毒HA抗原性进行分析,对于流感疫情的预测和预警具有重要意义[14]。
本研究中,宜春市11株甲型H1N1流感病毒与疫苗推荐株A/California/07/2009(H1N1)相比亲缘性近,HA序列没有发生较大变异;但HA氨基酸序列均发生P100S、S220T、I338V替换,这可能与地域差异性有关; HA氨基酸变异分析表明,HA氨基酸序列二硫键、糖基化位点无变异,与疫苗推荐株一致,这与2010年林毅雄等[15]的研究结果相一致;但有7株毒株在HA抗原决定簇区A区发生氨基酸替换变异,同时这些变异为毒株在130环或其附近发生的氨基酸替换变异。以上表明同地区同期分离的毒株变异率虽低,但仍在一定程度上存在变异。
虽然流感病毒变异不断发生,但并非所有变异毒株均能成为代表性变异株从而具有流行病学意义。一般认为具有流行病学意义的新变种的HA1蛋白分子,至少有4个以上氨基酸发生替换,且替换必须涉及2~3个抗原决定簇位点;同时不是HA蛋白分子上所有的氨基酸替换具有等同的意义,一般RBS周围如226和228位氨基酸发生替换比其他位点发生氨基酸替换要重要的多[9]。本研究中HA变异仅是分布在抗原决定簇的单个位点的变异,RBS周围226和228位点也没发生氨基酸替换,所以选取的11株甲型H1N1流感病毒没有产生具有流行病学意义新的变种。
但一般HA抗原决定簇氨基酸发生替换,会引起蛋白抗原性漂移。例如Klein等[16]的研究参照疫苗株A/California/07/2009(H1N1),发现甲型H1N1流感病毒HA蛋白抗原性漂变加权平均距离从2009年的3.6上升为2013年的11.7,而其相对应的编码区抗原性漂变加权平均距离从2009年的7.4上升为2013年的28.3;且同一时期的甲型H1N1流感病毒与H3N2流感病毒抗原性漂变速率大致相同。另外有资料表明HA受体结合位点的变异会影响病毒HA与其受体的结合,从而导致病毒对NA的依赖性降低,使抗NA流感药物失效[17]。
仅从HA基因特性分析,2013年宜春市甲型H1N1流感病毒不是流行病学新变种,目前的甲型H1N1流感疫苗虽仍对人群有保护作用;然而HA抗原决定簇和受体结合位点的变异表明有可能加速抗原性漂变,仍需要密切关注甲型H1N1流感的再次流行。
| [1] | Neumann G, Noda T, Kawaoka Y. Emergence and pandemic potential of swine-origin H1N1 influenza virus[J]. Nature,2009,459(7249):931-939. |
| [2] | Ron Fouchier AM, Munster V, Wallensten A, et al. Characterization of a novel influenza A virus hemagglutinin subtype (H16) obtained from black-headed gulls[J]. J Virol,2005,79(5):2814-2822. |
| [3] | Smith GJD, Vijaykrishna D, Bahl J, et al. Origins and evolutionary genomics of the 2009 swine-origin H1N1 influenza A epidemic[J]. Nature,2009,459(7250):1122-1125. |
| [4] | Novel Swine-Origin Influenza A (H1N1) Virus Investigation Team, Dawood FS, Jain S, et al. Emergence of a novel swine-origin influenza A (H1N1) virus in humans[J]. N Engl J Med,2009,360(25):2605-2615. |
| [5] | Wang TT, Palese P. Universal epitopes of influenza virus hemagglutinins?[J]. Nat Struct Mol Biol,2009,16(3):233-234. |
| [6] | WHO. WHO information for molecular diagnosis of influenza virus in humans-update[R]. Geneva:WHO,2011. |
| [7] | Basak S, Pritchard DG, Bhown AS, et al. Glycosylation sites of influenza viral glycoproteins:Characterization of tryptic glycopeptides from the A/USSR(H1N1) hemagglutinin glycoprotein[J]. J Virol,1981,37(2):549-558. |
| [8] | Camblin SJ, Haire LF, Russell RJ, et al. The structure and receptor binding properties of the 1918 influenza hemagglutinin[J]. Science,2004,303(5665):1838-1842. |
| [9] | Guo YJ, Cheng XW. Influenza virus and experimental techniques[M]. Beijing:China Three Gorges Publishing Press,1997:16-43. (in Chinese) 郭元吉,程小雯.流行性感冒病毒及其实验技术[M]. 北京:中国三峡出版社, 1997:16-43. |
| [10] | Xu R, Ekiert DC, Krause JC, et al. Structrural basis of preexisting immunity to the 2009 H1N1 pandemic influenza virus[J]. Science,2010,328(5976):357-360. |
| [11] | Matsuzaki Y, Sugawara K, Nakauchi M, et al. Epitope mapping of the hemagglutinin molecule of A/(H1N1)pdm09 influenza virus by using monoclonal antibody escape mutants[J]. J Virol,2014,88(21):12364-12373. |
| [12] | Weis W, Brown JH, Cusack S, et al. Structure of the influenza virus haemagglutinin complexed with its receptor, sialic acid[J]. Nature,1988,333(6172):426-431. |
| [13] | Wagner R, Matrosovich M, Klenk HD. Functional balance between haemagglutinin and neuraminidase in influenza virus infections[J]. Rev Med Virol,2002,12(3):159-166. |
| [14] | Shinde V, Bridges CB, Uyeki TM, et al. Triple-reassortant swine influenza A (H1) in humans in the United States,2005-2009[J]. N Engl J Med,2009,360(25):2616-2625. |
| [15] | Lin YX, Zhang LR, Wei QD, et al. Analysis on genetic characteristics of hemagglutinin of group A influenza H1N1 in Zhuhai in 2008[J]. Practical Preventive Medicine,2010,17(2):230-232. (in Chinese) 林毅雄,张丽荣,魏泉德,等.珠海市2008年H1N1亚型流感病毒血凝素HA1基因特征分析[J]. 实用预防医学,2010,17(2):230-232. |
| [16] | Klein EY, Serohijos AW, Chol JM, et al. Influenza a H1N1 pandemic strain evolution-divergence and the potential for antigenic drift variants[J]. PLoS One,2014,9(4):e93632. |
| [17] | Mishin VP, Novikov D, Hayden FG, et al. Effect of hemagglutinin glycosylation on influenza virus susceptibility to neuraminidase inhibitors[J]. J Virol,2005,79(19):12416-12424. |
 2016, Vol. 31
2016, Vol. 31


