扩展功能
文章信息
- 王斌, 王红, 熊衍文, 许彦梅, 刘祥, 肖波, 孙松松, 周阳, 白锐, 张玲
- WANG Bin, WANG Hong, XIONG Yan-wen, XU Yan-mei, LIU Xiang, XIAO Bo, SUN Song-song, ZHOU Yang, BAI Rui, ZHANG Ling
- 16S rDNA序列在艾伯特埃希菌鉴定中的应用
- Application of 16S rDNA sequence in identification of Escherichia albertii
- 疾病监测, 2016, 31(3): 205-208
- Disease Surveillance, 2016, 31(3): 205-208
- 10.3784/j.issn.1003-9961.2016.03.007
-
文章历史
- 收稿日期: 2015-10-10
2. 中国疾病预防控制中心传染病预防控制所, 传染病预防控制国家重点实验室, 北京 102206
2. State Key Laboratory for Communicable Disease Prevention and Control, Institute for Communicable Disease Prevention and Control, Chinese Center for Disease Control and Prevention, Beijing 102206, China
艾伯特埃希菌(Escherichia albertii)是一种新发现的与人类散发感染和突发食物中毒事件相关的食源性病原菌。艾伯特埃希菌最初分离于孟加拉国腹泻患儿的粪便中,并被鉴定为蜂房哈夫尼菌[1, 2, 3, 4, 5, 6]。后基于表型特征、16S rDNA序列和DNA杂交的分析表明该菌属于埃希菌的一个新种,并命名为艾伯特埃希菌[7, 8]。由于艾伯特埃希菌缺乏明确定义的生化特性,目前尚无该菌种的商业化鉴定系统,很难将其与大肠埃希菌及其他肠杆菌科细菌进行区分[9, 10]。与大肠埃希菌相比,缺乏动力、不发酵木糖、不发酵乳糖,产生β-D葡萄糖醛酸酶为艾伯特埃希菌的重要生化特性。艾伯特埃希菌属于黏附和抹平致病菌,它产生的损伤是通过外膜蛋白紧密黏附素和其他Ⅲ型分泌系统肠上皮细胞抹平LEE毒力岛效应物在肠的上皮细胞表面的共同作用[11]。紧密黏附素是由LEE毒力岛上的eae基因编码,它是一种具有30多种亚型的高度多态性蛋白,而且艾伯特埃希菌包含的紧密黏附素亚型在大肠埃希菌中很少发现[9]。此外,紧密黏附素、细胞膨胀致死毒素已经作为艾伯特埃希菌公认的毒力因子被报道。Hyma等基于艾伯特埃希菌管家基因lysP 和mdh的核酸多态性,设计了特异性的多重聚合酶联反应(PCR)体系,可用以从大肠埃希菌属和志贺菌属中区分艾伯特埃希菌属。通过分析16S rRNA基因和毒力基因核酸序列也支持该种方法[11, 12]。核酸序列多态性不依赖生化和免疫特性,因此提供一种快速准确筛选艾伯特埃希菌有效的方法[13, 14]。
人、动物、鸟类和水存在艾伯特埃希菌已有报道,但艾伯特埃希菌的流行病学特征仍然缺乏,主要原因在于艾伯特埃希菌不易被准确鉴定。本研究对30株疑似艾伯特埃希菌的16S rRNA基因进行扩增和测序,并通过构建进化树法来研究艾伯特埃希菌与E. coli、Shigella spp.、E. hermannii和Shimwellia blattae的亲缘关系,从而通过16S rDNA分析快速鉴定艾伯特埃希菌。
1 材料与方法 1.1 材料菌株来源于2013年3月至2014年7月从自贡市生肉样本中(猪肉、鸡肉、鸭肉、羊肉、鸡肠、鸭肠)分离到的30株疑似艾伯特埃希菌,其中鸡肠16株,鸭肠7株,鸡肉3株,鸭肉2株,羊肉和猪肉各1株。PCR试剂购自TaKaRa公司,引物合成及测序由生工生物工程(上海)有限公司完成,细菌总DNA提取使用Promega公司试剂盒,并按其说明书完成。
1.2 实验方法以30株可疑菌株DNA为模板,使用细菌16S rRNA基因通用引物(表 1)进行扩增,体系中包括25 μl 2×Taq Mastermix,上下游引物(10 μmol/L)各2 μl,模板1 μl,ddH2O 20 μl,总体积50 μl。PCR程序为 95 ℃预变性5 min,94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,30个循环,最后72 ℃延伸10 min。获得特异片段进行测序,并将序列上传至NCBI进行BLAST比对分析(http://blast.ncbi.nlm.nih.gov/Blast.cgi)。将获得的序列与艾伯特埃希菌LMG 20976、KF1、NBRC107761、TW07627以及其他基因组相关的细菌种(大肠埃希菌、志贺菌、费格森埃希菌、赫曼埃希菌、Shimwellia blattae)等的16S rDNA序列剪齐,然后应用ClustalW进行多序列比对。利用Mega 6.0软件中Neighbor-Joining法构建系统进化树以分析疑似艾伯特埃希菌与不同菌种之间的关联性。
| 基因 | 上游引物5′~3′ | 下游引物5′~3′ |
| 16S rRNA | AGA GTT TGA TCM TGG CTC AG | TAC GGY TAC CTT GTT ACG ACT T |
| eaeA | CAG GAT CGC CTT TTT TAT ACG | CTC TGC AGA TTA ACC TCT GC |
以30株可疑菌株DNA为模板使用eaeA基因特异引物(表 1)进行PCR扩增,体系中包括12.5 μl 2×Taq Mastermix,上下游引物(10 μmol/L)各 1 μl,模板1 μl,ddH2O 9.5 μl,总体积25 μl。PCR程序为 95 ℃预变性5 min,94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,30个循环,最后72 ℃延伸10 min。
2 结果提取30株疑似艾伯特埃希菌的核酸,以其为模板使用16S rRNA通用引物进行扩增,结果均获得长度为1500 bp左右的条带,且特异性较高(图 1)。将扩增产物送至生工生物工程股份有限公司进行测序,结果获得1506 bp长度的DNA序列。将该序列进行Blast分析,结果表明与艾伯特埃希菌序列一致性高。30株疑似菌16S rRNA 基因GenBank登录号为KP197062~KP197093。eaeA基因扩增结果均获得长度约500 bp的条带,特异度高(图 2)。

|
| 注:1-8. 样品;-. 阴性对照。 图 1 16S rRNA基因扩增结果 Figure 1 Amplification of 16s rDNA |

|
| 注:1-8. 样品;-. 阴性对照。 图 2 eaeA基因扩增结果 Figure 2 Amplification of eaeA |
将获得的30条16S rDNA序列与艾伯特相近种群16S rRNA核苷酸序列进行比较并生成相似矩阵得分。其中30株艾伯特埃希菌之间的相似性为99.7% ~100%,艾伯特埃希菌菌株LMG 20976相似性为99.4%~ 99.6%,表明具有高度相似性。
系统进化树表明30株疑似艾伯特埃希菌与艾伯特埃希菌KF1菌株相似并且集中(图 3),表明该30株为艾伯特埃希菌,其中SP140701与KF1 16S rDNA序列一致。聚类结果显示30株疑似菌株聚为五类。
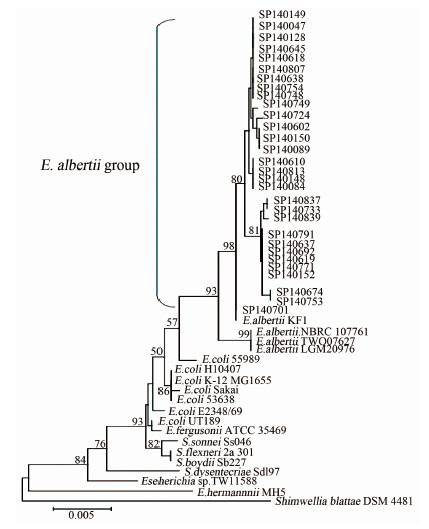
|
| 图 3 30株疑似菌株与标准菌株的进化树 Figure 3 The results of phylogenetic tree |
艾伯特埃希菌为新发潜在致病肠道致病菌,曾在日本一起突发事件中分离到艾伯特埃希菌,也被报道可能为引起美国阿拉斯加野鸟死亡的原因。对于国内对该菌的研究较少,在我国国内并未发现该菌引起的致病患者。
从自贡市市场中收集到的羊肉、猪肉、鸡肉、鸭肉、鸡肠、鸭肠中获得的疑似艾伯特埃希菌经鉴定为艾伯特埃希菌。在肉食品中发现该菌,表明该菌对于公众健康具有潜在的致病危险。进一步的研究需要评估这些菌株的致病性和克隆多样性,以及由该菌引起人类疾病的公共卫生重要性。本实验从自贡市的市场中抽样的肉食品检测结果显示,从多种肉食品中检出(鸡肠分离到16株,鸭肠7株,鸡肉3株,鸭肉2株,羊肉和猪肉各1株),但该菌是否能引起家禽致病还有待于进一步研究。从30株疑似菌株中均扩增出eaeA基因,表明该菌存在致病性的可能,但目前尚未在自贡市发现感染该菌的患者,该菌在人群中的感染筛查工作仍在继续。
在细菌基因组中,编码16S rRNA的rDNA基因具有良好的进化保守性,保守序列区域反映了生物物种间的亲缘关系,而高变序列区域则能体现物种间的差异。本实验通过对疑似菌株16S rDNA序列进行测序,并与艾伯特埃希菌的16S rDNA序列进行比对及聚类分析,能快速准确鉴定该菌是否为艾伯特埃希菌。通过对30株菌株的聚类分析,对于聚类的艾伯特埃希菌之间是否存在差异有待进一步研究。
本次实验发现的艾伯特埃希菌主要分离于鸡肠与鸭肠中,同时在市场的鸡肉、鸭肉、羊肉与牛肉中均发现该菌株,是否为市场中的肉食品污染所致值得进一步探究。今后的工作中,将继续对自贡市的肉类食品进行检测,并加强对腹泻患者的检测。对于鸡肠与鸭肠中存在该菌,是环境因素所致或是由于其本身为艾伯特埃希菌的携带者值得进一步探讨。
| [1] | Albert MJ, Alam K, Islam M, et al. Hafnia alvei, a probable cause of diarrhea in humans[J]. Infect Immu,1991, 59(4):1507-1513. |
| [2] | Konno T, Yatsuyanagi J, Takahashi S, et al. Isolation and identification of Escherichia albertii from a patient in an outbreak of gastroenteritis[J]. Jpn J Infect Dis,2012,65(3):203-207.rh20160306 |
| [3] | Asoshima N, Matsuda M, Shigemura K, et al. Identification of Escherichia albertii as a causative agent of a food-borne outbreak occurred in 2003[J]. Jpn J Infect Dis,2014,67(2):139-140.rh20160306 |
| [4] | Murakami K, Etoh Y, Tanaka E, et al. Shiga toxin 2f-producing Escherichia albertii from a symptomatic human[J]. Jpn J Infect Dis,2014,67(3):204-208.rh20160306 |
| [5] | Oaks JL, Besser TE, Walk ST, et al. Escherichia albertii in wild and domestic birds[J]. Emerg Infect Dis,2010,16(4):638-646.rh20160306 |
| [6] | Ooka T, Tokuoka E, Furukawa M, et al. Human gastroenteritis outbreak associated with Escherichia albertii, Japan[J]. Emerg Infect Dis,2013,19(1):144-146.rh20160306 |
| [7] | Huys G, Cnockaert M, Janda JM, et al.Escherichia albertii sp. nov., a diarrhoeagenic species isolated from stool specimens of Bangladeshi children[J]. Int J Syst Evol Microbiol,2003,53(3):807-810.rh20160306 |
| [8] | Bai XN, Wang H, Zhang ZD, et al. Research Advances on Escherichia albertii[J]. Chinese Journal of Microbiology and Immunology,2014,34(2):154-158. (in Chinese)白向宁,王红,张正东,等. 艾伯特埃希菌研究进展[J]. 中华微生物学和免疫学杂志,2014,34(2):154-158. |
| [9] | Kenny B. Mechanism of action of EPEC type Ⅲ effector molecules[J]. Int J Med Microbiol,2001,291(6/7):469-477. |
| [10] | Abbott SL, O'Connor J, Robin T, et al. Biochemical properties of a newly described Escherichia species, Escherichia albertii[J]. J Clin Microbiol,2003,41(10):4852-4854.rh20160306 |
| [11] | Ooka T, Seto K, Kawano K, et al. Clinical significance of Escherichia albertii[J]. Emerg Infect Dis,2012,18(3):488-492.rh20160306 |
| [12] | Hyma KE, Lacher DW, Nelson AM, et al. Evolutionary genetics of a new pathogenic Escherichia species:Escherichia albertii and related Shigella boydii strains[J]. J Bacteriol,2005,187(2):619-628.rh20160306 |
| [13] | Nimri LF. Escherichia albertii, a newly emerging enteric pathogen with poorly defined properties[J]. Diagn Microbiol Infect Dis,2013,77(2):91-95.rh20160306 |
| [14] | Oh JY, Kang MS, Hwang HT, et al. Epidemiological investigation of eaeA-positive Escherichia coli and Escherichia albertii strains isolated from healthy wild birds[J]. J Microbiol,2011,49(5):747-752.rh20160306 |
 2016, Vol. 31
2016, Vol. 31


