扩展功能
文章信息
- 郭文平, 林献丹, 王文, 李明慧, 王淼若, 邢建光, 郑剑, 孙肖瑜, 倪庆翔, 张永振
- GUO Wen-ping, LIN Xian-dan, WANG Wen, LI Ming-hui, WANG Miao-ruo, XING Jian-guang, ZHENG Jian, SUN Xiao-yu, NI Qing-xiang, ZHANG Yong-zhen
- 浙江省啮齿动物携带心病毒的遗传特征研究
- Analysis on genetic characteristics of cardioviruses carried by rodents in Zhejiang
- 疾病监测, 2016, 31(6): 452-458
- Disease Surveillance, 2016, 31(6): 452-458
- 10.3784/j.issn.1003-9961.2016.06.004
-
文章历史
- 收稿日期:2016-03-22
2. 温州市疾病预防控制中心, 浙江 温州 325001;
3. 龙泉市疾病预防控制中心, 浙江 龙泉 323700;
4. 文成县疾病预防控制中心, 浙江 文成 325300;
5. 瑞安市疾病预防控制中心, 浙江 瑞安 325200
2. Wenzhou Prefecture Center for Disease Control and Prevention, Wenzhou 325001, Zhejiang, China;
3. Longquan County Center for Disease Control and Prevention, Longquan 323700, Zhejiang, China;
4. Wencheng County Center for Disease Control and Prevention, Wencheng 325300, Zhejiang, China;
5. Rui'an County Center for Disease Control and Prevention, Rui'an 325200, Zhejiang, China
心病毒属(Cardiovirus)为小RNA病毒科,其基因组为不分节段的单股正链RNA,长约7.7~8.5 kb。心病毒的基因组包括一个大的开放读码框(open reading frame,ORF),其两侧为5′与3′非编码区(noncoding regions,NCRs),在3′NCR的末端为一个长度可变的多聚腺苷酸尾巴(poly A),部分病毒的5′NCR的末端有一个长度可变的多聚胞嘧啶尾巴(poly C)[1]。心病毒属5′NCR中的内部核糖体进入位点(Internal ribosome entry site,IRES)为Ⅱ型[2]。病毒的mRNA翻译成一个多聚蛋白,然后被水解为功能不同的小蛋白,顺序依次为Leader蛋白(L蛋白)、结构蛋白P1(进一步水解为VP4、VP2、VP3与VP1)、非结构蛋白P2(进一步水解为2A、2B与2C)与P3(进一步水解为3A、3B、3C与3D)[3]。这些蛋白各自行使病毒复制、包装及感染过程中的功能。
根据国际病毒分类委员会的最新分类,心病毒属包括心病毒A、B、C三个种,其宿主主要为啮齿动物、猪、非人灵长类以及人[1, 3]。在心病毒属中,猪脑心肌炎病毒(Encephalomyocarditis virus,EMCV)能引起猪的脑炎、心肌炎等为主要特征的急性传染病[4];Saffold virus(SAFV)可以感染人,引起腹泻、上呼吸道感染,严重的可引起心肌炎[5];Theiler’s murine encephalitis virus(TMEV)可以引起易感宿主的急性脑脊髓炎[6]。目前研究发现,在我国儿童及家猪中均发现心病毒感染[7-8]。啮齿动物是心病毒的重要宿主,国际上对啮齿动物携带该病毒的研究较多[6]。我国的啮齿动物种类众多,但对其携带心病毒情况未进行过系统研究。本研究对我国浙江省心病毒感染情况进行调查,以了解其在啮齿动物中的流行情况及遗传特征。
1 材料与方法 1.1 主要试剂粪便总RNA快速提取试剂盒购自北京百泰克生物技术有限公司;Taq酶购自北京全式金生物技术有限公司;One Step PrimeScript RT-PCR 试剂盒、RACE试剂盒、pMD 19-T载体、DH5α感受态细胞、DNA Marker、M-MLV反转录酶、胶回收试剂盒MiniBEST Agarose Gel DNA Extraction Kit均购自TaKaRa公司。
1.2 标本采集2013年35月在浙江省龙泉市、文成县、龙湾区、瑞安市4个监测点的居民区以及野外采集啮齿类动物。以油条为诱饵用鼠笼捕捉啮齿动物。捕获的啮齿动物经形态学鉴定后,无菌条件下解剖取其直肠,-80 ℃冰箱中保存。
1.3 病毒核酸检测取50 mg粪便标本置于2 ml无RNase的离心管中,参照百泰克生物技术有限公司的RNA提取试剂使用说明书提取粪便RNA。使用引物Oligo dT与M-MLV反转录酶将RNA反转录为cDNA。用半巢式PCR方法扩增心病毒基因组5′NCR的部分区域检测病毒核酸。
1.4 病毒全基因组扩增采用基因组步移法扩增心病毒全基因组,在已知序列中设计两条特异性引物,另一端设计一条简并引物进行半巢式PCR。简并引物根据心病毒已知序列保守区域进行设计。用RACE试剂盒分别扩增基因组的5′与3′末端。引物由生工生物工程(上海)股份有限公司合成。具体引物序列见表 1。
| 引物名称 | 5′~ 3′ | 片段长度 |
| 用于初筛的引物 | ||
| CarF1 | GCC YCT GSG GCC RAA AGC C(+) | 123 bp |
| CarF2 | CTG SGG CCR AAA GCC MCG TG(+) | |
| CarR | GGT ACC TYC HGG RCA TYC YT(-) | |
| 用于扩增全基因组的引物 | ||
| Ruian-Rn93-1 | ||
| 5′RACE-R2 | GGT TCA CCA GAG TAC AAC C(-) | 5′ RACE下游引物 |
| 5′RACE-R1 | CCG CTC ACA CCA GTG GAT(-) | |
| CarF1 | GCC YCT GSG GCC RAA AGC C(+) | 约2100 bp |
| CarF2 | CTG SGG CCR AAA GCC MCG TG(+) | |
| R | GCR TAA GTG GCC TGC AT(-) | |
| 93-1F1 | CTC AAT GCT GGC TGC AGT(+) | 约2500 bp |
| 93-1F2 | CAA CCA GTA CAG AGG ATC T(+) | |
| 93-1R1 | ACY GAT TTY CCT TGT CCT G(-) | |
| 93-1F3 | CAG ATC AAT CAT TGA TTG GT(+) | 约2300 bp |
| 93-1F4 | TGC TCA CAG ACT TCC CAC(+) | |
| 93-1R2 | GAA GGA TTG RTA YAC KAC AT(-) | |
| 93-1F5 | CAC CAA ACT TGG AAC AGA C(+) | 3′ RACE上游引物 |
| 93-1F6 | CAG GCA ATT CTA GGA CTT C(+) | |
| Ruian-Rn93-3 | ||
| 5′RACE-R2 | GAT TCC ACA GAG AAC AAC C(-) | 5′ RACE下游引物 |
| 5′RACE-R1 | ACC ACT CAC AGC AGT GGA T(-) | |
| CarF1 | GCC YCT GSG GCC RAA AGC C(+) | 约2100 bp |
| CarF2 | CTG SGG CCR AAA GCC MCG TG(+) | |
| R | GCR TAA GTG GCC TGC AT(-) | |
| 93-3F1 | CTC TAT TCT TGC AGC TGT(+) | 约2500 bp |
| 93-3F2 | TAA TCA GTA CCG TGG CTC T(+) | |
| 93-3R1 | ACY GAT TTY CCT TGT CCT G(-) | |
| 93-3F3 | CCA AAA GAT CGT TGA GTG G(+) | 约2300 bp |
| 93-3F4 | GGA TCA TTG TGC CTC CAT(+) | |
| 93-3R2 | GAA GGA TTG RTA YAC KAC AT(-) | |
| 93-3F5 | GAG TGT TTA CCA AAC TAG G(+) | 3′ RACE上游引物 |
| 93-3F6 | AGC AGG CCA TCC TTG GAT(+) | |
| Longquan-Aa3 | ||
| 5′RACE-R2 | GGT TCA CCA GAG TAT GAC C(-) | 5′ RACE下游引物 |
| 5′RACE-R1 | CCG CTC ATA GAA GTG GAT(-) | |
| CarF1 | GCC YCT GSG GCC RAA AGC C(+) | 约2100 bp |
| CarF2 | CTG SGG CCR AAA GCC MCG TG(+) | |
| R | GCA TAR GTC GMC TGC AT(-) | |
| 3-3F1 | CTC GAT GCT GGC TGC AGT(+) | 约2500 bp |
| 3-3F2 | CAA CCA GTA TAG AGG ATC T(+) | |
| 3-3R1 | ACY GAT TTY CCT TGT CCT G(-) | |
| 3-3F3 | CAA GGC AAT CAT AGA CTG G(+) | 约2300 bp |
| 3-3F4 | TGC TAA TGG ATT TCC CAG(+) | |
| 3-3R2 | GAA GGA TTG RTA YAC KAC AT(-) | |
| 3-3F5 | TAT TCA CCC GAC TTG GAA C(+) | 3′ RACE上游引物 |
| 3-3F6 | CAA GCA ATT CTC GGA CTA C(+) | |
| Wencheng-Rn416 | ||
| 5′RACE-R2 | TGG TTA CTA AGT CAA ATC C(-) | 5′ RACE下游引物 |
| 5′RACE-R1 | CGC TCT CAA GGG TGC TA(-) | |
| CarF1 | GCC YCT GSG GCC RAA AGC C(+) | 约2100 bp |
| CarF2 | CTG SGG CCR AAA GCC MCG TG(+) | |
| R | GCG TAG GTY CCC TGC AT(-) | |
| 416-3F1 | TCT CTT GGT TCC CTT GCT(+) | 约2500 bp |
| 416-3F2 | CTC ACA GTA TCG TGG TTC T(-) | |
| 416-3R1 | ACY GAT TTY CCT TGT CCT G(-) | |
| 416-3F3 | CAA AAC TCT CTT CAA TTG G(+) | |
| 416-3F4 | AGG CAG ACT CAA CAA GCT(+) | 约2300 bp |
| 416-3R2 | AAC TTT GGT AGA CCA CAT G(-) | |
| 416-3F5 | CAC ACA GCT AGG AAT GGA T(+) | |
| 416-3F6 | GAA GCA GTT GAT GGT GTT G(+) | 3′ RACE上游引物 |
用TaKaRa公司的胶回收试剂盒回收预期目的大小的PCR扩增产物,与T载体连接,转化大肠埃希菌感受态细胞,将大肠埃希菌涂在含有X-gal、IPTG以及氨苄西林的LB固体培养基上进行培养。挑选白色菌落,接种在LB液体培养基中,以菌液为模板进行PCR鉴定。阳性克隆送生工生物工程(上海)股份有限公司测序,每个PCR扩增产物至少送3个阳性克隆。
1.6 系统发生分析用Lasergene 7.1软件中的SeqMan程序拼接测序结果,得到序列的全基因组。采用与相关参考序列的比对以及NetPicoRNA 1.0 server (http://www.cbs.dtu.dk/services/NetPicoRNA/)确定病毒多聚蛋白可能的蛋白酶切位点。用保守序列数据库(Conserved domain database,CDD)分析多聚蛋白中的保守蛋白家族。用Lasergene软件中的MegAlign程序进行氨基酸同源性分析。由于不同心病毒的核苷酸序列差异较大,因此选用蛋白序列来构建系统发育树;同时,由于不同毒株的2A蛋白差异也较大,因此在构建P2蛋白的进化树时未包括2A蛋白。最终,P1、P2(不含2A)与P3蛋白序列被用于系统发生分析。使用Mega 6.06软件将序列保存为FASTA格式,用其中的Muscle程序进行序列比对。同时,用Mega 6.06软件计算氨基酸序列的最佳替代模型,结果显示mtREV+Γ+I为P1、P2(不含2A)与P3蛋白的最佳替代模型。使用Mega 6.06软件将序列转换成PHY格式。然后使用phyML 3.1软件中的最大似然法(Maximum Likelihood,ML)构建系统发育树[9]。分析采用100个多序列组(replicates)。使用mid-point的方法定义系统发育树的根。
2 结果 2.1 心病毒的鉴定本次调查共捕获啮齿动物307只,经形态学鉴定后分属于7个种,分别为黑线姬鼠(Apodemus agrarius)、小家鼠(Mus musculus)、褐家鼠(Rattus norvegicus)、罗赛鼠(R. lossea)、黄胸鼠(R. tanezumi)、东方田鼠(Microtus fortis)与社鼠(Niviventer confucianus)。其中褐家鼠为优势鼠种,其次为黑线姬鼠(表 2)。
| 物种 | 龙泉市 | 文成县 | 龙湾区 | 瑞安市 | 合计 |
| 黑线姬鼠 | 1/36 | 0/21 | 0/0 | 0/4 | 1/61 |
| 小家鼠 | 0/0 | 0/11 | 0/0 | 0/2 | 0/13 |
| 褐家鼠 | 0/37 | 1/29 | 0/5 | 1/33 | 2/104 |
| 罗赛鼠 | 0/27 | 0/2 | 0/2 | 0/10 | 0/41 |
| 黄胸鼠 | 0/0 | 0/27 | 0/2 | 0/9 | 0/38 |
| 东方田鼠 | 0/31 | 0/0 | 0/0 | 0/0 | 0/31 |
| 社鼠 | 0/0 | 0/19 | 0/0 | 0/0 | 0/19 |
| 合计 | 1/131 | 1/109 | 0/9 | 1/58 | 3/307 |
| 注:表内数据为PCR阳性只数/捕获数。 | |||||
从鼠粪便标本中提取总RNA,经半巢式RT-PCR扩增基因组5′NCR的部分区域,在3份样本中获得了与预期大小相符合的目的片段,长度约为150 bp。3份样本分别是龙泉市的黑线姬鼠、文成县及瑞安市的褐家鼠。心病毒在啮齿类动物中的携带率为0.97%,黑线姬鼠的携带率为1.64%,褐家鼠的携带率为1.92%。目的片段经克隆后进行测序,在瑞安市的褐家鼠中扩增出2条大小一致的片段,为共感染。将测序结果进行Blastn比对,发现其中的3条序列与心病毒属中心病毒B的5′NCR的同源性最高,>73.8%;而另1条序列与Boone心病毒的同源性最高,为98.6%。因此,3只鼠携带的4个病毒均属于心病毒属,分别命名为Ruian-Rn93-1、Ruian-Rn93-3、Longquan-Aa3-1与Wencheng-Rn416。
2.2 全基因组特征采用基因组步移法结合RACE方法扩增病毒的全基因组。毒株Ruian-Rn93-1、Ruian-Rn93-3、Longquan-Aa3-1、Wencheng-Rn416扩增到的序列长度分别为7945 bp、7541 bp、8077 bp和8417 bp。根据与已知序列的比对,未扩增到完整的5′NCR,可能是由于病毒RNA的降解引起的。这4株病毒的基因组结构为5′NCR-L+VP4+VP2+VP3+VP1+2A+2B+2C+3A+3B+3Cpro+3Dpol-3′NCR。
根据Kozak的保守序列(RNNAUGG)[10],预测新发现心病毒多聚蛋白翻译的起始密码子。4株病毒的开放读码框分别位于895~7809 bp、490~7404 bp、1037~7948 bp、1339~8250 bp,分别编码2304、2304、2303、2303个氨基酸的多聚蛋白。因毒株Ruian-Rn93-1、Ruian-Rn93-3、Longquan-Aa3-1的5′NCR不完整,因此无法使用软件预测其二级结构,但其与心病毒B的同源性最高,推测拥有Ⅱ型内部核糖体进入位点。毒株Wencheng-Rn416的5′NCR与Boone心病毒有最高98.1%的同源性。目前Boone心病毒的内部核糖体进入位点序列与其他小RNA病毒的差异很大,预测的结构与5个型的内部核糖体进入位点均不一致,不能很好地对其进行分类。
2.3 多聚蛋白的特征根据与相关参考序列的比对以及NetPicoRNA 1.0 server 分析,预测了新发现心病毒多聚蛋白的蛋白酶切位点(表 3、图 1)。本研究中新发现的毒株均具有Rhv-like衣壳结构域(capsid domain) (cd00205),2C RNA 解旋酶(RNA helicase)(pfam00910),3C半胱氨酸蛋白酶的保守结构域(3C cystein protease)(pfam00548)与3D RNA依赖的RNA聚合酶(RdRp)(cd01699)(图 1)。4个毒株的多聚蛋白中均含有L蛋白,分析其蛋白序列,结果显示没有由保守氨基酸残基组成的Cys-His-Asn/Asp催化三联体,因此这些毒株的L蛋白不具有半胱氨酸蛋白酶活性。与Thera virus(TRV)、Theiler’s-like virus一致,毒株Ruian-Rn93-1、Ruian-Rn93-3与Longquan-Aa3-1的基因组还可以编码L*蛋白。对于P1蛋白,所有毒株有保守的十四烷基化的氨基酸残基GXXX(ST)[11],VP1蛋白不包含(PS)ALXAXETG氨基酸残基,也没有整合素结合的RGD基序。对于非结构蛋白P2,除毒株Wencheng-Rn416之外,其他3个毒株的2A与2B间存在NPGP氨基酸残基,所有毒株的2C蛋白包括与NTP结合的GXXGXGKS以及与解旋酶活性相关的DDLXQ氨基酸残基[12-13]。对于非结构蛋白P3,3C蛋白包括酶活性催化三联体H-D-C以及与蛋白酶活性相关的GXCG与GXH氨基酸残基[14];与其他所有的小RNA病毒一致,所有新发现毒株的3D蛋白中包括保守的GLPSG、YGDD与FLKR氨基酸残基[15]。
| 蛋白 | Ruian-Rn93-1 | Ruian-Rn93-3 | Longquan-Aa3-1 | Wencheng-Rn416 |
| L | M1_Q76 | M1_Q76 | M1_Q76 | M1_Q96 |
| VP4 | G77_L147 | G77_M150 | G77_L147 | G97_M168 |
| VP2 | D148_Q414 | D151_Q421 | D148_E414 | D169_Q426 |
| VP3 | S415_Q649 | S422_Q652 | S415_Q648 | A427_Q656 |
| VP1 | G650_E925 | G653_Q925 | G649_E924 | G657_E912 |
| 2A | N926_G1058 | N926_G1058 | N925_G1057 | M913_P1037 |
| 2B | P1059_Q1194 | P1059_Q1194 | P1058_Q1193 | N1038_Q1186 |
| 2C | G1195_Q1520 | S1195_Q1520 | G1194_Q1519 | M1187_Q1513 |
| 3A | S1521_Q1606 | S1521_Q1606 | S1520_Q1605 | S1514_E1610 |
| 3B | A1607_Q1626 | A1607_Q1626 | A1606_Q1625 | A1611_Q1628 |
| 3C | G1627_Q1843 | G1627_Q1843 | G1626_Q1842 | L1629_Q1841 |
| 3D | G1844_R2304 | G1844_R2304 | G1843_R2303 | G1842_Q2303 |
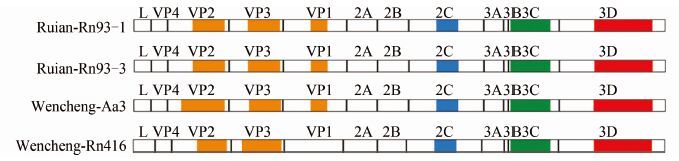
|
| 图 1 本研究中新发现心病毒多聚蛋白的结构图 Figure 1 Polyprotein structures of Cardioviruses found in this study |
| |
根据国际病毒分类委员会的规定,心病毒是根据多聚蛋白、P1以及2C+3CD蛋白的同源性来定义种。在心病毒属中,毒株Ruian-Rn93-1、Longquan-Aa3-1、Ruian-Rn93-3与TMEV、TRV、Vilyuisk human encephalomyelitis virus(VHEV)以及SAFV之间的同源性最高,多聚蛋白的同源性为71.9%~85.4%,P1蛋白的同源性为65.2%~89.8%,2C+3CD蛋白之间的同源性为78.3%~95.4%,这些毒株与心病毒属其他毒株之间的同源性较低。毒株Wencheng-Rn416与Boone心病毒的同源性最高,多聚蛋白、P1蛋白与2C+3CD蛋白之间的同源性分别为92.5%、85.3%与96.9%。毒株Ruian-Rn93-1、Ruian-Rn93-3与Longquan-Aa3-1的L*蛋白与TRV的同源性为44.9%~79.5%。
2.5 系统发育分析用P1、P2(不含2A)与P3蛋白构建的系统发育树见图 2。在P1蛋白构建的系统发育树中,毒株Ruian-Rn93-1、Longquan-Aa3-1与毒株Theiler’s-like virus、TRV聚在一起,然后与Sikhote-Alin virus、Theilovirus、TMEV聚在一起;Ruian-Rn93-3与Human TMEV-like心病毒以及SAFV聚在一起;所有这些毒株构成了心病毒B。Wencheng-Rn416与Boone心病毒聚在一起形成了心病毒C,位于系统发育树最原始的位置。P2(不含2A)、P3蛋白构建的系统发育树的拓扑结构与P1蛋白的类似,只是在P2(不含2A)与P3蛋白构建的系统发育树上,毒株Theiler’s-like virus与TRV的聚类方式发生了变化。除此之外,在P3蛋白构建的系统发育树上,毒株Ruian-Rn93-1、Longquan-Aa3-1的聚类方式也发生了变化。这种拓扑结构不一致表明心病毒属的基因组在进化过程中发生了基因重组。在系统发育树上,毒株RD1338位于心病毒A中较原始的位置。除Sing-M100-02与RD1338之外,该病毒种中的其他毒株的亲缘关系均较近,宿主包括啮齿动物、非人灵长类以及猪。Genet fecal theilovirus在系统发育树上单独形成一支,为一新种。
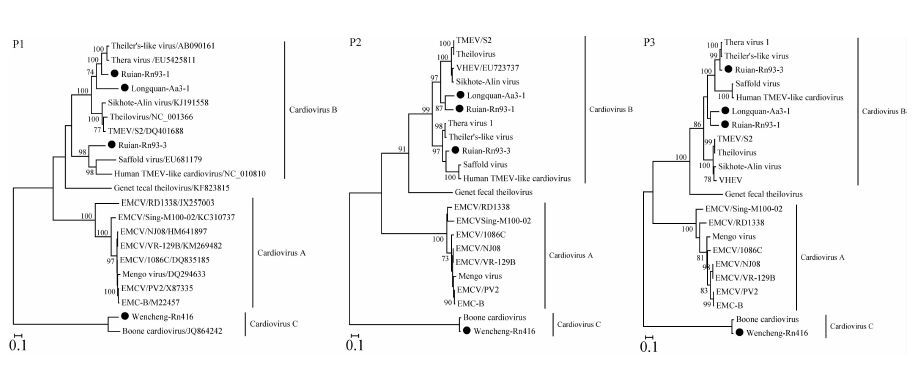
|
| 图 2 基于P1、P2(不含2A)与P3蛋白用最大似然法构建的系统发育树 Figure 2 Phylogenetic trees based on P1,P2(without 2A) and P3 aa sequences by ML methods |
| |
研究发现,啮齿动物可以携带和传播多种病毒[16]。在我国,啮齿动物传播的病毒主要为引起肾综合征出血热的汉坦病毒[17]。然而,Li等[17]的研究发现我国的啮齿动物可以携带沙粒病毒,其致病性未知,应引起关注。本研究采用PCR技术对我国浙江省啮齿动物中心病毒的感染情况进行调查,发现黑线姬鼠与褐家鼠可以携带心病毒,为首次从啮齿动物中检测到该病毒。随后扩增了4株病毒的全基因组,但未获得完整的5′NCR。基因组特征分析表明,毒株拥有典型的心病毒的基因组结构,且ORF编码的多聚蛋白拥有与心病毒属中其他毒株相似的保守结构域以及发挥重要功能的氨基酸残基。同源性比较推测毒株Ruian-Rn93-1、Longquan-Aa3-1,Ruian-Rn93-3可能有Ⅱ型内部核糖体进入位点。
国际病毒分类委员会建议根据多聚蛋白、P1以及2C+3CD蛋白的同源性来定义新发现的病毒是否为新种[1]。因此,笔者扩增这些毒株的全基因组。国际病毒分类委员会对心病毒种的定义为多聚蛋白的同源性>70%,P1蛋白的同源性>60%,2C+3CD蛋白的同源性>70%。本研究毒株Ruian-Rn93-1、Longquan-Aa3-1、Ruian-Rn93-3的多聚蛋白、P1以及2C+3CD蛋白与心病毒B的同源性分别为71.9%~85.4%、65.2%~89.8%与78.3%~95.4%,均大于国际病毒分类委员会对新种的定义,因此这些毒株属于心病毒B。毒株Wencheng-Rn416的多聚蛋白、P1以及2C+3CD蛋白与Boone心病毒的同源性分别高达92.5%、85.3%与96.9%,这两个毒株属于心病毒C。
与氨基酸同源性分析结果类似,毒株Ruian-Rn93-1、Longquan-Aa3-1、Ruian-Rn93-3在系统发育树上与心病毒B聚集在一起,共同形成心病毒B。毒株Wencheng-Rn416与Boone心病毒聚集在一起,共同形成心病毒C。在P1~P3蛋白构建的有根树上,心病毒C位于进化树最原始的位置,迄今为止,该种中发现毒株的宿主为啮齿目动物。在心病毒A中,位于原始位置的毒株RD1338分离自小林姬鼠,毒株C1086的宿主为褐家鼠,毒株PV2的宿主为小家鼠[1, 3]。在心病毒B中,啮齿动物是TMEV与TRV的宿主。综上所述,啮齿动物携带的心病毒具有更大的遗传多样性,并且在进化树中占据着祖先支的位置,数据初步支持啮齿动物在心病毒的起源与进化过程中起着重要作用。但心病毒起源于啮齿动物,这一假设尚需进一步实验验证。
在自然条件下,宿主动物感染病毒后其生长繁殖一般不受影响。然而啮齿动物在自然界中与人随机接触,极有可能导致病毒发生跨物种传播,从而导致相关疾病在人群中暴发与流行[18]。我国啮齿动物的种类众多,分布极为广泛,且与人类活动关系密切。系统发育分析表明,分离自非人灵长类的毒株VR-129B以及Mengo virus与猪以及啮齿动物中毒株的亲缘关系非常近,分离自人的VHEV与啮齿动物中毒株的亲缘关系也非常近。这些结果提示心病毒在不同的物种中可以发生跨种间传播[3]。本研究中发现的Ruian-Rn93-3与感染人的SAFV之间也有较近的亲缘关系,有可能传播到人,应引起重视。
综上所述,我国浙江省的啮齿动物中存在心病毒,系统发育分析发现啮齿动物在心病毒的起源与进化过程中起着重要作用,并且可能发生跨种间传播而感染人。
| [1] | Knowles NJ, Hovi T, Hyypiä T, et al. Picornaviridae[M]//King AMQ, Adams MJ, Carstens EB, et al. Virus Taxonomy:Classification and Nomenclature of Viruses:9th Report of the International Committee on Taxonomy of Viruses. San Diego:Elsevier ,2012:855-880. |
| [2] | Martínez-Salas E, Francisco-Velilla R, Fernandez-Chamorro J, et al. Picornavirus IRES elements:RNA structure and host protein interactions[J]. Virus Res, 2015 : 62–73 . |
| [3] | Carocci M, Bakkali-Kassimi L. The encephalomyocarditis virus[J]. Virulence, 2012, 3 (4) : 351–367 . |
| [4] | Billinis C, Paschaleri-Papadopoulou E, Psychas V, et al. Persistence of encephalomyocarditis virus (EMCV) infection in piglets[J]. Vet Microbiol, 1999, 70 (3/4) : 171–177 . |
| [5] | Nielsen TS, Nielsen AY, Banner J, et al. Saffold virus infection associated with human myocarditis[J]. J Clin Virol, 2016 : 78–81 . |
| [6] | Buckwalter MR, Nga PT, Gouilh MA, et al. Identification of a novel neuropathogenic Theiler's murine encephalomyelitis virus[J]. J Virol, 2011, 85 (14) : 6893–6905 . |
| [7] | Gai XN, Yang HC, Guo X, et al. Isolation and characterization of porcine encephalomyocarditis virus[J]. Acta Veterinariaet Zootechnica Sinica, 2007, 38 (1) : 59–65 .(in Chinese) 盖新娜, 杨汉春, 郭鑫, 等. 猪脑心肌炎病毒的分离与鉴定[J]. 畜牧兽医学报,2007,38 (1) :59–65. |
| [8] | Zhang XA, Lu QB, Wo Y, et al. Prevalence and genetic characteristics of Saffold cardiovirus in China from 2009 to 2012[J]. Sci Rep, 2015 : 7704. |
| [9] | Guindon S, Dufayard JF, Lefort V, et al. New algorithms and methods to estimate maximum-likelihood phylogenies:assessing the performance of PhyML 3[J]. Syst Biol, 2010, 59 (3) : 307–321 . |
| [10] | Kozak M. Point mutations define a sequence flanking the AUG initiator codon that modulates translation by eukaryotic ribosomes[J]. Cell, 1986, 44 (2) : 283–292 . |
| [11] | Chow M, Newman JFE, Filman D, et al. Myristylation of picornavirus capsid protein VP4 and its structural significance[J]. Nature, 1987, 327 (6122) : 482–486 . |
| [12] | Gorbalenya AE, Koonin EV, Wolf YI. A new superfamily of putative NTP-binding domains encoded by genomes of small DNA and RNA viruses[J]. FEBS Lett, 1990, 262 (1) : 145–148 . |
| [13] | Gorbalenya AE, Koonin EV, Donchenko AP, et al. Two related superfamilies of putative helicases involved in replication, recombination, repair and expression of DNA and RNA genomes[J]. Nucleic Acids Res, 1989, 17 (12) : 4713–4730 . |
| [14] | Gorbalenya AE, Donchenko AP, Blinov VM, et al. Cysteine proteases of positive strand RNA viruses and chymotrypsin-like serine proteases[J]. FEBS Lett, 1989, 243 (2) : 103–114 . |
| [15] | Kamer G, Argos P. Primary structural comparison of RNA-dependent polymerases from plant, animal and bacterial viruses[J]. Nucleic Acids Res, 1984, 12 (18) : 7269–7282 . |
| [16] | Meerburg BG, Singleton GR, Kijlstra A. Rodent-borne diseases and their risks for publichealth[J]. Crit Rev Microbiol, 2009, 35 (3) : 221–270 . |
| [17] | Li K, Lin XD, Wang W, et al. Isolation and characterization of a novel arenavirus harbored by Rodents and Shrews in Zhejiang province, China[J]. Virology, 2015 : 37–42 . |
| [18] | Zhang YZ, Zou Y, Fu ZF, et al. Hantavirus infections in humans and animals, China[J]. Emerg Infect Dis, 2010, 16 (8) : 1195–1203 . |
 2016, Vol. 31
2016, Vol. 31


