扩展功能
文章信息
- 张慧芳, 陶晓霞, 何利华, 闫笑梅, 王忱诚, 张建中, 肖迪
- ZHANG Hui-fang, TAO Xiao-xia, HE Li-hua, YAN Xiao-mei, WANG Chen-cheng, ZHANG Jian-zhong, XIAO Di
- 传代培养对基于肽质量指纹谱的细菌识别稳定性影响分析
- Effect of passage on the stability of bacterial identification based on peptide mass fingerprinting
- 疾病监测, 2016, 31(9): 734-739
- Disease Surveillance, 2016, 31(9): 734-739
- 10.3784/j.issn.1003-9961.2016.09.007
-
文章历史
- 收稿日期:2016-02-01
2. 浙江大学医学院附属第一医院传染病诊治国家重点实验室, 感染性疾病诊治协同创新中心, 浙江 杭州 310003
2. State Key Laboratory for Diagnosis and Treatment of Infectious Diseases, Collaborative Innovation Center for Diagnosis and Treatment of Infectious Diseases, The First Affiliated Hospital, College of Medicine, Zhejiang University, Hangzhou 310003, Zhejiang, China
病原体的快速、准确鉴定是传染病预防控制和临床病原诊断等公共卫生问题的关键。病原微生物感染的诊断方法应满足快速、准确、高效等多方面因素。传统方法主要有血清学鉴定、生化鉴定和分子鉴定等。血清学鉴定和生化鉴定操作复杂、步骤繁琐、检测时间长,并且带有一定的主观性。分子鉴定是识别病原菌最有效的方法,但需传代增菌提核酸,检测时间长、价格昂贵。传统的诊断方法不能满足病原诊断的理想化需求,新方法的探索一直是研究热点。
随着软电离技术的发展,基于基质辅助激光解析飞行时间质谱(matrix-assisted laser desorption/ionization time-of-flight mass spectrometry,MALDI-TOF MS)的肽质量指纹谱(peptide mass fingerprinting,PMF)]微生物识别技术也随之问世。与传统的血清学方法、分子诊断方法相比,PMF对病原菌的诊断更高效、准确、价廉[1]。PMF通过样品与酸性基质混合形成结晶,基质辅助样品裂解,激光激发细胞裂解物,形成肽质量指纹图谱,并与数据库中已知微生物的标准指纹图谱进行比对,从而实现对微生物的快速正确鉴定。目前真菌、厌氧菌、杆菌属、克罗诺杆菌属、军团菌、β溶血链球菌、幽门螺杆菌、小肠耶尔森菌、艰难梭菌、葡萄球菌、根瘤菌、非发酵菌、大规模的临床分离株都进行了PMF识别能力评价分析[2-18];显示了良好的识别准确性。PMF对微生物识别能力评价、分离来源、培养时间、生长条件、培养基的选择等的影响已有报道,菌株传代会导致菌株基因突变、启动子变化、毒力基因变化,而是否会导致分子水平的PMF发生变化、是否因其变化而导致对微生物识别能力下降尚待研究。
PMF进行微生物识别是基于一簇多肽(20~70个)对细菌种水平进行识别,同一菌种不同菌株间的肽质量谱簇没有显著差异,都可以识别为同一菌种。幽门螺杆菌是革兰阴性菌,其基因呈多态性,具有高度自然变异性;大肠埃希菌为革兰阴性菌、常见病原,亦为微生物鉴定用质谱仪校正用菌株;金黄色葡萄球菌是革兰阳性菌的代表。因此本研究拟对幽门螺杆菌、大肠埃希菌、金黄色葡萄球菌进行多次传代培养,并研究其PMF的变化及对菌株识别能力的影响。
1 材料与方法 1.1 仪器与试剂采用德国布鲁克公司的Microflex LT质谱仪和普通营养琼脂平板。细菌培养基为哥伦比亚血平板(CM0331,OXOID)。其他试剂:超纯水(18.2兆欧),α-氢基-4-羟基肉桂酸(α-cyano-4-hydroxycinnamic acid,CHCA,Sigma),色谱级甲酸(Sigma)、乙醇(Sigma)、乙腈(Sigma)、三氟乙酸(TFA,Sigma)。
1.2 实验菌株革兰阳性菌:金黄色葡萄球菌BJ0030〔分离于北京疾病预防控制中心(CDC)健康人鼻拭子〕;革兰阴性菌:幽门螺杆菌CQDPHP719(分离于重庆大坪医院胃黏膜标本),大肠埃希菌ATCC8739,由中国CDC传染病预防控制所诊断室保存。
1.3 实验方法 1.3.1 细菌传代幽门螺杆菌和金黄色葡萄球菌采用哥伦比亚血平板培养,大肠埃希菌采用普通营养琼脂平板培养。从-80 ℃冰箱中取出原代菌株进行复苏,挑取单菌落接种于新鲜的培养基上。金黄色葡萄球菌BJ0030、大肠埃希菌 ATCC8739于37 ℃、需氧培养12~24 h,金黄色葡萄球菌采用革兰染色、镜检、触媒及凝固酶实验对其进行表型及生化确认;幽门螺杆菌CQDPHP719 于37 ℃、微需氧培养24~48 h,并采用革兰染色、镜检、尿素酶、氧化酶及过氧化氢酶实验对其进行表型及生化确认。将通过表型和生化确认的金黄色葡萄球菌和幽门螺杆菌进行传代培养,金黄色葡萄球菌、大肠埃希菌培养24 h为一代,金黄色葡萄球菌传代至100代、大肠埃希菌传代至50代;幽门螺杆菌培养48 h为一代,传代至57代。
1.3.2 蛋白样品制备采用乙醇/甲酸法制备样本:取适量(5~10 mg)细菌,加入300 μl超纯水,混匀,再加入900 μl无水乙醇,混匀。高速离心(12 000 r/min) 2 min,弃去上清(必要时再离心一次,尽量去除乙醇和水)。加入50 μl 70%甲酸,混匀,再加入50 μl乙腈,混匀,高速离心(12 000 r/min)2 min,吸出上清置干净的EP管中备用。
1.3.3 质谱分析条件和数据采集质谱仪为德国布鲁克公司的Microflex LT。数据采集软件为德国布鲁克公司的FlexControlTM 3.0。微生物识别软件为德国布鲁克公司的Biotyper 2.0软件包;谱图分析软件为德国布鲁克公司的FlexAnalysis 3.0。样品承载靶为德国布鲁克公司的MSP 96 target ground steelTM 600-μm。
大肠埃希菌ATCC8739为标准品校正仪器,校正后分子量平均偏差小于200 ppm。取1 μl样品点于样品靶上,室温自然干燥后再点上1 μl基质溶解液(50%超纯水、50%乙腈和2.5%的三氟乙酸制备的CHCA饱和溶液),自然干燥后进行质谱检测。每张谱图 50 shots,500 shots叠加为一张总谱图。
1.3.4 细菌识别判读标准质谱仪Microflex LT所产生的谱图经系统标准方法处理后,采用Biotyper软件搜索包含有3995张参考谱的数据库。使用软件标准参数,判读标准:分值≥2.300表示种水平高可信度识别,分值≥2.000表示种水平识别,分值1.700~1.999表示属水平识别,分值<1.700表示未识别。
1.3.5 NanoLC-MS/MS蛋白扫描分析将幽门螺杆菌用于质谱鉴定的2000~20 000 Da的预提取蛋白进行酶解。采用纳升级高效液相色谱进行分离,与装配有纳米电喷雾离子源的LTQ Orbitrap XL质谱相连。采用LTQ Orbitrap XL数据依赖模式在MS与MS/MS之间转换。在m/z 350~1600范围内、m/z 400的分辨率为100 000时全扫描一级谱图,而后选择10个最强母离子进行二级数据采集。采用35%的归一化碰撞能量,默认0.25激活值,激活时间30 ms。Xcalibur 2.1.0软件记录谱图。
Mascot Daemon(Version 2.3.0,MatrixScience,London,UK)软件用于搜索UniProtKB数据库(Swiss-Prot和TrEMBL)。串联谱图采用extract_msn.exe(Thermo Fisher Science Inc.)提取。两个漏切位点,半胱氨酸carbamidomethy1修饰选为固定修饰,蛋氨酸oxidation为可变修饰,肽质量偏差为20 ppm,离子片段偏差为0.8 Da。采用反转数据库去除假阳性鉴定。
2 结果大肠埃希菌传代50代、幽门螺杆菌共传57代,搜索Biotyper肽质量参考谱库,每代都正确识别到种水平。菌株各代PMF谱图与各代叠加的PMF谱图没有显著差异,见图 1、2。

|
| 图 1 大肠埃希菌50代肽质量指纹谱 Figure 1 PMF of 50 generations of E. coli |
| |

|
| 图 2 幽门螺杆菌57代肽质量指纹谱 Figure 2 PMF of 57 generations of H. pylori |
| |
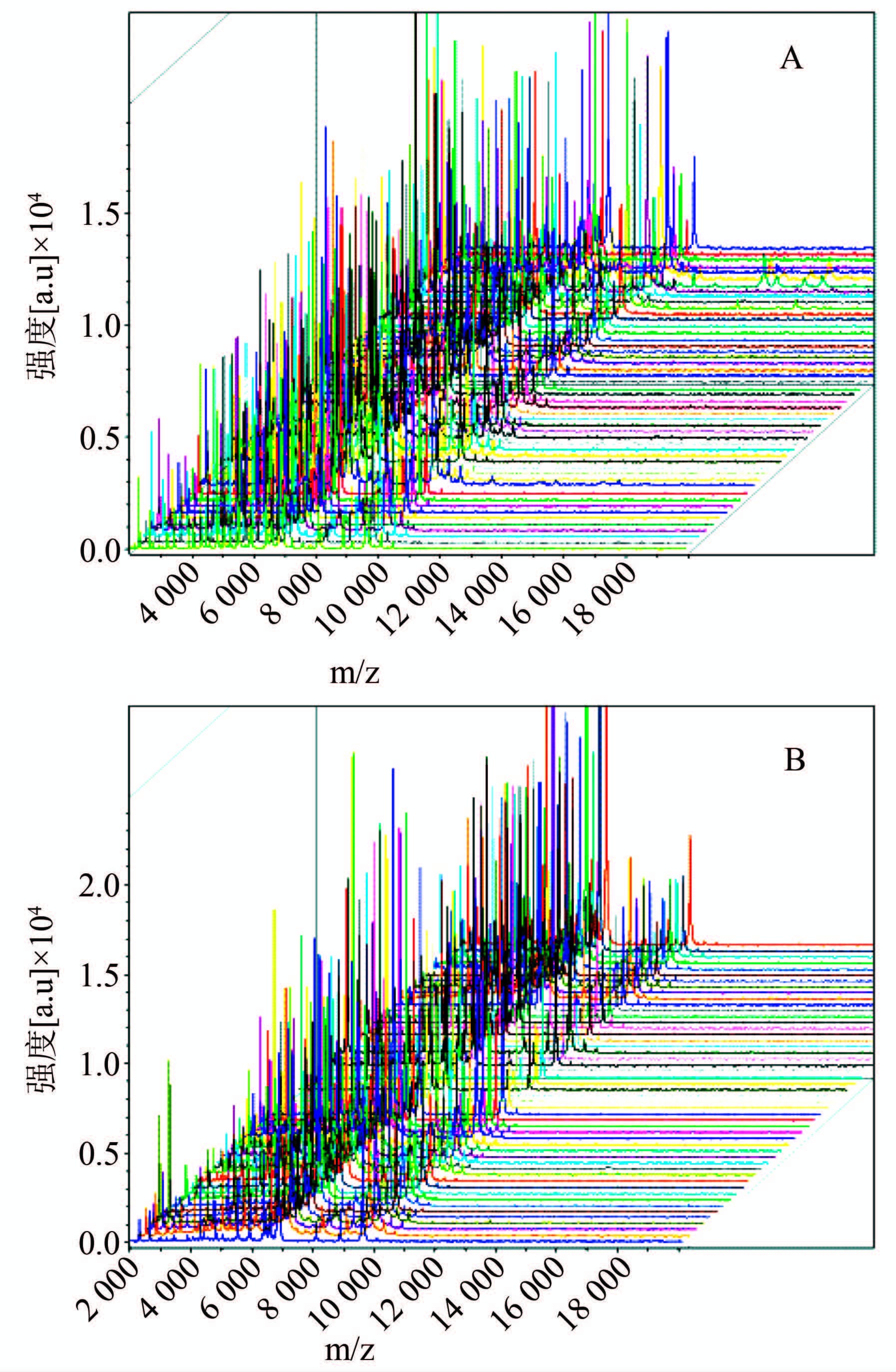
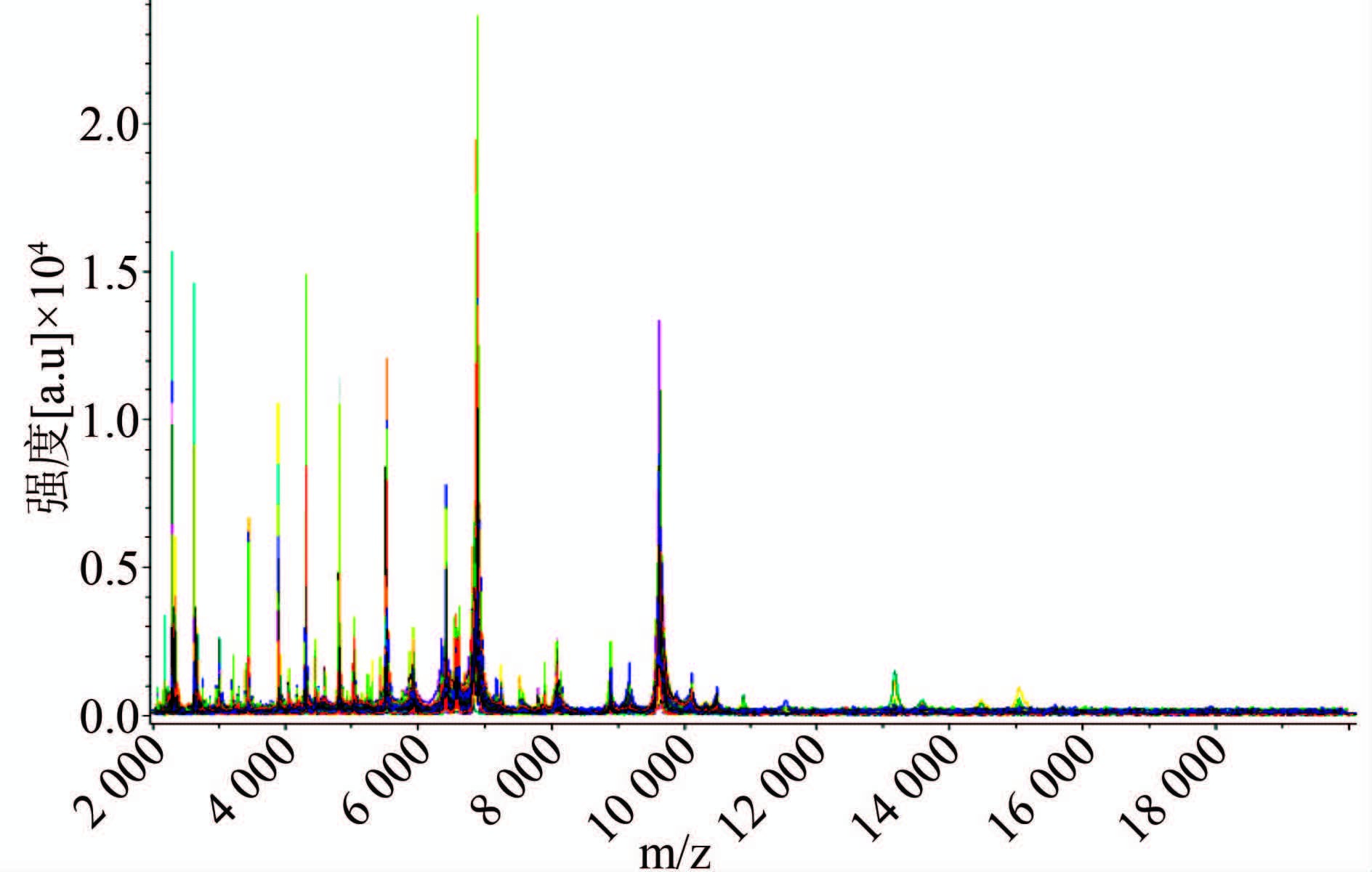
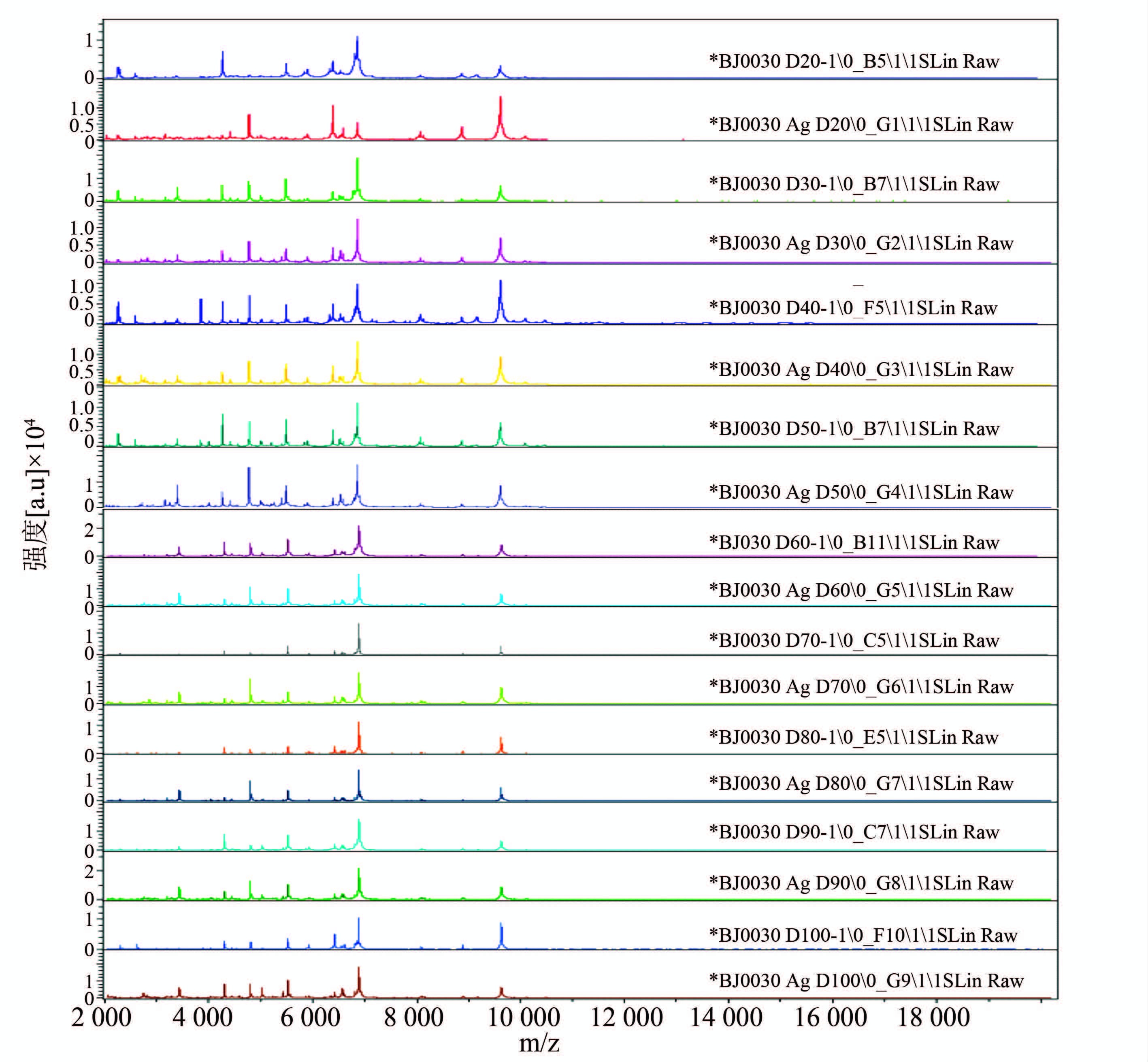
金黄色葡萄球菌共传100代,每代在强度一致的情况下谱图没有显著差异,见图 3。100代的叠加谱图也显示出很好的一致性,见图 4。搜索Biotyper肽质量参考谱库,每代均能正确识别到种水平。各代冻存抗原的PMF和新鲜菌株的PMF一致性很好,所有抗原均同时采集的谱图,第一代与最后一代相隔6个月,谱图间没有显著差异,见图 5,并且都被正确识别到种水平。
|
| 图 3 金黄色葡萄球菌1~100代肽质量指纹谱 Figure 3 PMF of 1 - 100 generations of S. aureus |
| |
|
| 图 4 金黄色葡萄球菌1~100代肽质量指纹谱叠加图 Figure 4 Superposition of PMF of 1-100 generations of S. aureus |
| |
|
| 图 5 金黄色葡萄球菌新鲜菌及抗原100代肽质量指纹谱对比图 Figure 5 Comparison of 100 generations PMF of fresh bacteria and frozen antigen of S. aureus |
| |
扫描幽门螺杆菌用于微生物鉴定的2000~20 000 Da的预提取蛋白共鉴定出206个蛋白,包括酶类(29.6%)、核糖体蛋白(15.5%)、外膜蛋白(10.7%)、假想蛋白(19.0%)、转运相关蛋白(2.0%),其余的22.0%包括毒力相关蛋白VacA、CagA、CagI、CagF、甲基趋化因子蛋白、反应调节蛋白、铁蛋白、DNA结合蛋白、鞭毛蛋白、黏附蛋白、肿瘤坏死因子和延伸因子。
3 讨论细菌在体外人工培养基上连续多次传代后,基因组会发生突变,毒力基因发生变化,导致菌株发生变异,随着传代次数增多,细菌的变异概率增大。付乔芳等[19]研究显示嗜水气单胞菌在培养基上连续多次传代后,菌株毒力逐渐减弱甚至消失;嗜水气单胞菌毒力基因的分布亦受传代影响,细菌5种毒力基因经多次传代后,基因型发生了显著变化。金黄色葡萄球菌5种毒力因子α溶血素(ALF)、β溶血素B(BET)、血浆凝固酶(Xjngm)、黏附素(ZFB)、耐热核酸酶(Nuc)在传代过程中Xjngm、ALF、BET 基因携带率发生较大变化,Xjngm在第20代和第25代中该毒力基因都未被检测到;Nuc在原代、第5代、第10代、第15代、第20代携带率基本没有变化,在第25代携带率变化较大;ZFB的携带率基本未变[20]。红色毛癣菌在传代过程中容易发生变异,失去原来的生理和生化特性[21]。国外的一些研究也显示细菌和病毒在体外连续传代后其毒力明显减弱[22-24]。
本研究显示,多次传代不会影响幽门螺杆菌、大肠埃希菌、金黄色葡萄球菌基于PMF的正确识别。即使金黄色葡萄球菌抗原被冻存6个月后,其PMF与新鲜培养菌没有显著差异,各代均能正确识别。新鲜培养的菌株室温存储时间过长、4 ℃条件存放超过8 d将严重影响鉴定结果[8]。对于菌株鉴定前的保存,本研究提示,通过冷冻保存菌株抗原,至少6个月内均可正常使用。
相对于传统的微生物鉴定方法,PMF技术是一种操作简单、快速、高通量的微生物鉴定方法。其主要针对菌体所含小分子量蛋白或多肽进行检测,即细菌菌体表面蛋白和细菌内高丰度和持续表达的蛋白。虽然多次传代会导致幽门螺杆菌和金黄色葡萄球菌的基因突变,启动子发生变化,某些毒力基因会发生变化,其他微生物也会有类似的情况,但用于微生物识别的PMF是基于2000~20 000 Da的多肽或小蛋白,这部分蛋白或多肽的重要组成成分是核糖体蛋白[25-26]。本研究扫描用于幽门螺杆菌鉴定的蛋白成分,共发现206个蛋白,其中包括15.5%的核糖体蛋白,核糖体蛋白虽然不是含量最多的蛋白,但推测它是丰度较高的、在MALDI-TOF质谱线性模式下有一定峰强的、保守的用于PMF识别的主要蛋白。真正用于识别的具有属和种共性的肽质量谱是不多于70个肽的肽系列簇。这部分肽在细菌传代过程中相对保守,即使有少许的变化,也不会影响整个系统对细菌的正确识别。PMF使用特异的谱图模式对微生物进行快速的识别,具有很好的重复性,细菌的代数不会影响PMF对细菌的正确识别。
| [1] | Saffert RT, Cunningham SA, Ihde SM, et al. Comparison of Bruker Biotyper matrix-assisted laser desorption ionization-time of flight mass spectrometer to BD Phoenix automated microbiology system for identification of gram-negative bacilli[J]. J Clin Microbiol , 2011, 49 (3) : 887–892. DOI:10.1128/JCM.01890-10 |
| [2] | Putignani L, Del Chierico F, Onori M, et al. MALDI-TOF mass spectrometry proteomic phenotyping of clinically relevant fungi[J]. Mol Bio Syst , 2011, 7 (3) : 620–629. |
| [3] | Theel ES, Hall L, Mandrekar J, et al. Dermatophyte identification using matrix-assisted laser desorption ionization-time of flight mass spectrometry[J]. J Clin Microbiol , 2011, 49 (12) : 4067–4071. DOI:10.1128/JCM.01280-11 |
| [4] | Erhard M, Hipler UC, Burmester A, et al. Identification of dermatophyte species causing onychomycosis and tinea pedis by MALDI-TOF mass spectrometry[J]. Exp Dermatol , 2008, 17 (4) : 356–361. DOI:10.1111/exd.2008.17.issue-4 |
| [5] | Yan Y, He Y, Maier T, et al. Improved identification of yeast species directly from positive blood culture media by combining sepsityper specimen processing and microflex analysis with the matrix-assisted laser desorption ionization biotyper system[J]. J Clin Microbiol , 2011, 49 (7) : 2528–2532. DOI:10.1128/JCM.00339-11 |
| [6] | Stevenson LG, Drake SK, Shea YR, et al. Evaluation of matrix-assisted laser desorption ionization-time of flight mass spectrometry for identification of clinically important yeast species[J]. J Clin Microbiol , 2010, 48 (10) : 3482–3486. DOI:10.1128/JCM.00687-09 |
| [7] | Justesen US, Holm A, Knudsen E, et al. Species identification of clinical isolates of anaerobic bacteria: a comparison of two matrix-assisted laser desorption ionization-time of flight mass spectrometry systems[J]. J Clin Microbiol , 2011, 49 (12) : 4314–4318. DOI:10.1128/JCM.05788-11 |
| [8] | Alispahic M, Christensen H, Hess C, et al. Identification of Gallibacterium species by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry evaluated by multilocus sequence analysis[J]. Int J Med Microbiol , 2011, 301 (6) : 513–522. DOI:10.1016/j.ijmm.2011.03.001 |
| [9] | Stephan R, Ziegler D, Pflüger V, et al. Rapid genus-and species-specific identification of Cronobacter spp. by matrix-assisted laser desorption ionization-time of flight mass spectrometry[J]. J Clin Microbiol , 2010, 48 (8) : 2846–2851. DOI:10.1128/JCM.00156-10 |
| [10] | He Y, Chang TC, Li HJ, et al. Matrix-assisted laser desorption ionization time-of-flight mass spectrometry and database for identification of Legionella species[J]. Can J Microbiol , 2011, 57 (7) : 533–538. DOI:10.1139/w11-039 |
| [11] | Cherkaoui A, Emonet S, Fernandez J, et al. Evaluation of matrix-assisted laser desorption ionization-time of flight mass spectrometry for rapid identification of Beta-hemolytic streptococci[J]. J Clin Microbiol , 2011, 49 (8) : 3004–3005. DOI:10.1128/JCM.00240-11 |
| [12] | Ilina EN, Borovskaya AD, Serebryakova MV, et al. Application of matrix-assisted laser desorption/ionization time-of-flight mass spectrometry for the study of Helicobacter pylori[J]. Rapid Commun Mass Spectrom , 2010, 24 (3) : 328–334. DOI:10.1002/rcm.v24:3 |
| [13] | Stephan R, Cernela N, Ziegler D, et al. Rapid species specific identification and subtyping of Yersinia enterocolitica by MALDI-TOF mass spectrometry[J]. J Microbiol Methods , 2011, 87 (2) : 150–153. DOI:10.1016/j.mimet.2011.08.016 |
| [14] | Reil M, Erhard M, Kuijper EJ, et al. Recognition of Clostridium difficile PCR-ribotypes 001,027 and 126/078 using an extended MALDI-TOF MS system[J]. Eur J Clin Microbiol Infect Dis , 2011, 30 (11) : 1431–1436. DOI:10.1007/s10096-011-1238-6 |
| [15] | Bergeron M, Dauwalder O, Gouy M, et al. Species identification of staphylococci by amplification and sequencing of the tuf gene compared to the gap gene and by matrix-assisted laser desorption ionization time-of-flight mass spectrometry[J]. Eur J Clin Microbiol Infect Dis , 2011, 30 (3) : 343–354. DOI:10.1007/s10096-010-1091-z |
| [16] | Ferreira L, Sánchez-Juanes F, García-Fraile P, et al. MALDI-TOF mass spectrometry is a fast and reliable platform for identification and ecological studies of species from family Rhizobiaceae[J]. PLoS One , 2011, 6 (5) : e20223. DOI:10.1371/journal.pone.0020223 |
| [17] | Mellmann A, Cloud J, Maier T, et al. Evaluation of matrix-assisted laser desorption ionization-time-of-flight mass spectrometry in comparison to 16S rRNA gene sequencing for species identification of nonfermenting bacteria[J]. J Clin Microbiol , 2008, 46 (6) : 1946–1954. DOI:10.1128/JCM.00157-08 |
| [18] | Seng P, Drancourt M, Gouriet F, et al. Ongoing revolution in bacteriology: routine identification of bacteria by matrix-assisted laser desorption ionization time-of-flight mass spectrometry[J]. Clin Infect Dis , 2009, 49 (4) : 543–551. DOI:10.1086/599188 |
| [19] | Fu QF, Qiu JQ, Hu K, et al. Stability of virulence genes of Aeromonas hydrophila strains during subculture[J]. Biotechnology Bulletin , 2011 (9) : 130–135. (in Chinese) 付乔芳, 邱军强, 胡鲲, 等. 嗜水气单胞菌毒力基因在传代过程中的稳定性研究[J]. 生物技术通报 , 2011 (9) : 130–135. |
| [20] | Sun MY, He JB. Stability of virulence genes in staphylococcus aureus strains during subculture[J]. Modern Journal of Animal Husbandry and Veterinary Medicine , 2014 (2) : 13–16. (in Chinese) 孙美英, 何剑斌. 金黄色葡萄菌球菌毒力基因在传代过程中的稳定性[J]. 现代畜牧兽医 , 2014 (2) : 13–16. |
| [21] | Yang GL, Du YC, Zhang ML, et al. Influence on phenotype and ribosomal DNA sequence of Trichophytin rubrum resulting from subculture[J]. The Chinese Journal of Dermatovenereology , 2007, 21 (5) : 263–266. (in Chinese) 杨国玲, 杜迎春, 张明莉, 等. 传代对红色毛癣菌表型和核糖体基因影响的研究[J]. 中国皮肤性病学杂志 , 2007, 21 (5) : 263–266. |
| [22] | Songe MM, Thoen E, Evensen Ø, et al. In vitro passages impact on virulence of Saprolegniaparasitica to Atlantic salmon, Salmo salar L. parr[J]. J Fish Dis , 2014, 37 (9) : 825–834. DOI:10.1111/jfd.2014.37.issue-9 |
| [23] | Almaguer-Chávez JA, Welsh O, Lozano-Garza HG, et al. Decrease of virulence for BALB/c mice producedby continuous subculturing of Nocardiabrasiliensis[J]. BMC Infect Dis , 2011, 11 : 290. DOI:10.1186/1471-2334-11-290 |
| [24] | Druelle J, Sellin CI, Waku-Kouomou D, et al. Wild type measles virus attenuation independent of type Ⅰ IFN[J]. Virol J (1) : 1–12. |
| [25] | Suh MJ, Hamburg DM, Gregory ST, et al. Extending ribosomal protein identifications to unsequenced bacterial strains using matrix-assisted laser desorption/ionization mass spectrometry[J]. Proteomics , 2005, 5 (18) : 4818–4831. DOI:10.1002/(ISSN)1615-9861 |
| [26] | Christensen JJ, Dargis R, Hammer M, et al. Matrix-assisted laser desorption ionization-time of flight mass spectrometry analysis of Gram-positive, catalase-negative cocci not belonging to the Streptococcus or Enterococcus genus and benefits of database extension[J]. J Clin Microbiol , 2012, 50 (5) : 1787–1791. DOI:10.1128/JCM.06339-11 |
 2016, Vol. 31
2016, Vol. 31


