扩展功能
文章信息
- 韩艳青, 侯凤伶, 张淑红, 申玉学, 史红, 关文英, 申志新
- HAN Yan-qing, HOU Feng-ling, ZHANG Shu-hong, SHEN Yu-xue, SHI Hong, GUAN Wen-ying, SHEN Zhi-xin
- 基于多重PCR的副溶血弧菌大流行菌群及其毒力基因检测方法的建立与评价
- Establishment and evaluation of a novel multiplex PCR assay in detection of pandemic group of Vibrio parahaemolyticus and toxic genes
- 疾病监测, 2016, 31(9): 750-754
- Disease Surveillance, 2016, 31(9): 750-754
- 10.3784/j.issn.1003-9961.2016.09.010
-
文章历史
- 收稿日期:2015-10-30
副溶血弧菌(Vibrio parahaemolyticus,Vp)是一种嗜盐的革兰阴性菌,广泛分布于水生环境及无脊椎动物中,也是常见的食源性致病菌。与引起人类胃肠炎的临床分离株相比,环境分离株普遍缺乏耐热直接溶血素(thermostable direct hemolysin,TDH)、TDH相关溶血素(TDH-related hemolysin,TRH)等致病性蛋白的编码基因tdh和trh等[1-3],因此tdh和trh成为鉴别副溶血弧菌致病性的重要标志物。
副溶血弧菌最早发现于1953年,1963年由Sakazaki等[4]正式命名,此后一直在世界范围内呈现由不同血清型引起的散发特征。1996年Okuda等[5]首次报道了O3∶K6血清型副溶血弧菌引起的胃肠炎的流行,此后其发病率在世界范围内显著增高,此后又陆续发现了O4∶K68、O1∶K25、O1∶K26、O1∶KUT(O1∶K untypeable)等共14种高致病性的血清型,共同命名为“大流行菌群”(pandemic group)[6-7]。这些大流行菌株的共同特点除了致病性基因trh阴性和tdh阳性等特征之外,还有特征性的独有序列ToxR/new,位于基因toxRS操作子。因此基于此序列的群特异性PCR即GS-PCR是大流行菌群的主要鉴定依据[8-9]。
近年来副溶血弧菌,特别是大流行菌群严重威胁食品安全和人民健康[10-12]。为了快速准确的鉴定副溶血弧菌大流行菌群并了解其致病性,本研究结合了编码致病因子TDH和TRH的基因tdh和trh序列以及GS-PCR所常用的toxRS基因序列,建立了一种快速方便地对副溶血弧菌大流行菌群以及毒力基因准确鉴定的多重PCR方法。
1 材料与方法 1.1 实验菌株及核酸提取本实验所用副溶血弧菌临床分离株UP8(O1∶K6血清型,tdh阳性,trh阳性)及大流行株FR2(O3∶K6血清型,tdh阳性)由国家食品风险评估中心提供。副溶血弧菌临床分离株为本实验保存。大肠埃希菌、阪崎肠杆菌、金黄色葡萄球菌、沙门菌、志贺菌、单核细胞增生性李斯特菌、霍乱弧菌、蜡样芽孢杆菌、空肠弯曲菌、结肠弯曲菌、阪崎肠杆菌、小肠结肠炎耶尔森菌、副溶血弧菌(非大流行株)等对照菌株来源于中国医学细菌保藏管理中心。
选取对数生长期的平板菌落,用细菌DNA提取试剂盒(北京卓诚惠生生物科技有限公司)提取细菌基因组DNA。操作步骤参照试剂盒说明书进行。
1.2 引物设计及多重PCR体系构建参照NCBI中GS-PCR特征基因toxRS,以及致病因子编码基因tdh和trh序列,用Primer 5.0软件设计特异性引物。引物序列用BLAST在线比对GenBank数据库,未发现与其他物种有非特异结合。tdh、toxRS和trh 目的条带大小分别为:269 bp、509 bp和653 bp,GS-PCR上下游引物位于参考序列(根据文献报道选用GenBank accession no.L11929)576位和1196位的两个大流行群固有的变异位点,经过primerBLAST验证,引物仅识别大流行菌群,而不识别非大流行的副溶血弧菌toxRS序列。为了提高大流行菌群鉴定的特异性,将大流行菌群特异碱基设于引物3′端第一位。引物由Introvigen合成。引物序列见表 1。
| 引物名称 | 引物序列(5′~3′) |
| tdh-F | GTA TTC ACA ACG TCA GGT AC |
| tdh-R | TTA CCA CTA CCA CTC TCA TA |
| trh-F | TAT TCG ACT MTT TCT GCT TTR A |
| trh-R | AGR TCA ATC GCG AAT GAT TTA G |
| toxRS-F(1) | GCT AAT GAG GTA GAA ACA |
| toxRS-R(1) | ACG TAA CGG GCC TAC A |
| 注:(1)带下划线碱基为大流行菌群特异性位点识别碱基。 | |
用TaKaRa Taq HS(TaKaRa)按试剂盒说明书建立每反应25 μl的多重PCR反应体系。每条引物工作浓度均为200 nmol/L,每反应加入2 μl 所提取的细菌DNA,反应条件:预变性95 ℃ 5 min;循环温度为94 ℃ 30 s;57 ℃ 30 s;72 ℃ 60 s;重复30个循环;最后72 ℃延伸10 min。
1.3 多重PCR产物分析PCR产物使用QIAxcel(QIAGEN)系统进行全自动电泳分析。具体参数:电泳卡夹选用QIAxcel DNA Screening Kit,分析方法选择AM420,用Size Marker 100 bp~1.5 kb和Alignment Marker 15 bp/2 kb作为分子质量参比。
1.4 多重PCR特异性验证为了验证多重PCR体系的特异性和准确性,从非大流行的临床分离株UP8和大流行菌株FR2中分别提取核酸,同时也从其他食源性致病菌,包括大肠埃希菌、阪崎肠杆菌、金黄色葡萄球菌、沙门菌、志贺菌、单核细胞增生性李斯特菌、霍乱弧菌、蜡样芽孢杆菌、空肠弯曲菌、结肠弯曲菌、阪崎肠杆菌和小肠结肠炎耶尔森菌中以同样的方法提取核酸作为阴性对照。所有的样本核酸在相同的条件下构建多重PCR反应体系比较PCR产物。
1.5 多重PCR灵敏度验证为了验证多重PCR体系的灵敏度,对2个阳性菌株,非大流行临床分离株UP8和大流行株FR2,分别用PBS建立10倍梯度稀释的菌悬液(108CFU/ml~104 CFU/ml)离心收集菌体并提取核酸,用多重PCR体系扩增并电泳检测条带。
1.6 血清型鉴定待测菌株分别接种于两管3%氯化钠胰蛋白胨大豆琼脂斜面培养。分别用日本生研株式会社的血清鉴定11种O抗原和74种K抗原。
2 结果 2.1 多重PCR反应的特异性用参考菌株所提取的DNA对引物特异性进行评价的结果:副溶血弧菌临床分离株UP8的基因组中可特异性扩增出269 bp的tdh条带和509 bp的trh条带。副溶血弧菌大流行株FR2可特异扩增出653 bp的GS-PCR条带的同时,也可扩增出269 bp的tdh条带,但未扩出trh条带。非大流行株的副溶血弧菌无653 bp的GS-PCR条带。其他食源性致病菌,如大肠埃希菌、阪崎肠杆菌、金黄色葡萄球菌、沙门菌、志贺菌、单核细胞增生性李斯特菌、霍乱弧菌、蜡样芽孢杆菌、空肠弯曲菌、结肠弯曲菌、溶藻孤菌、小肠结肠炎耶尔森菌等,也无相应扩增产物,见图 1。因此该组引物特异性强,可用于PCR检测副溶血弧菌大流行株及其毒力基因。
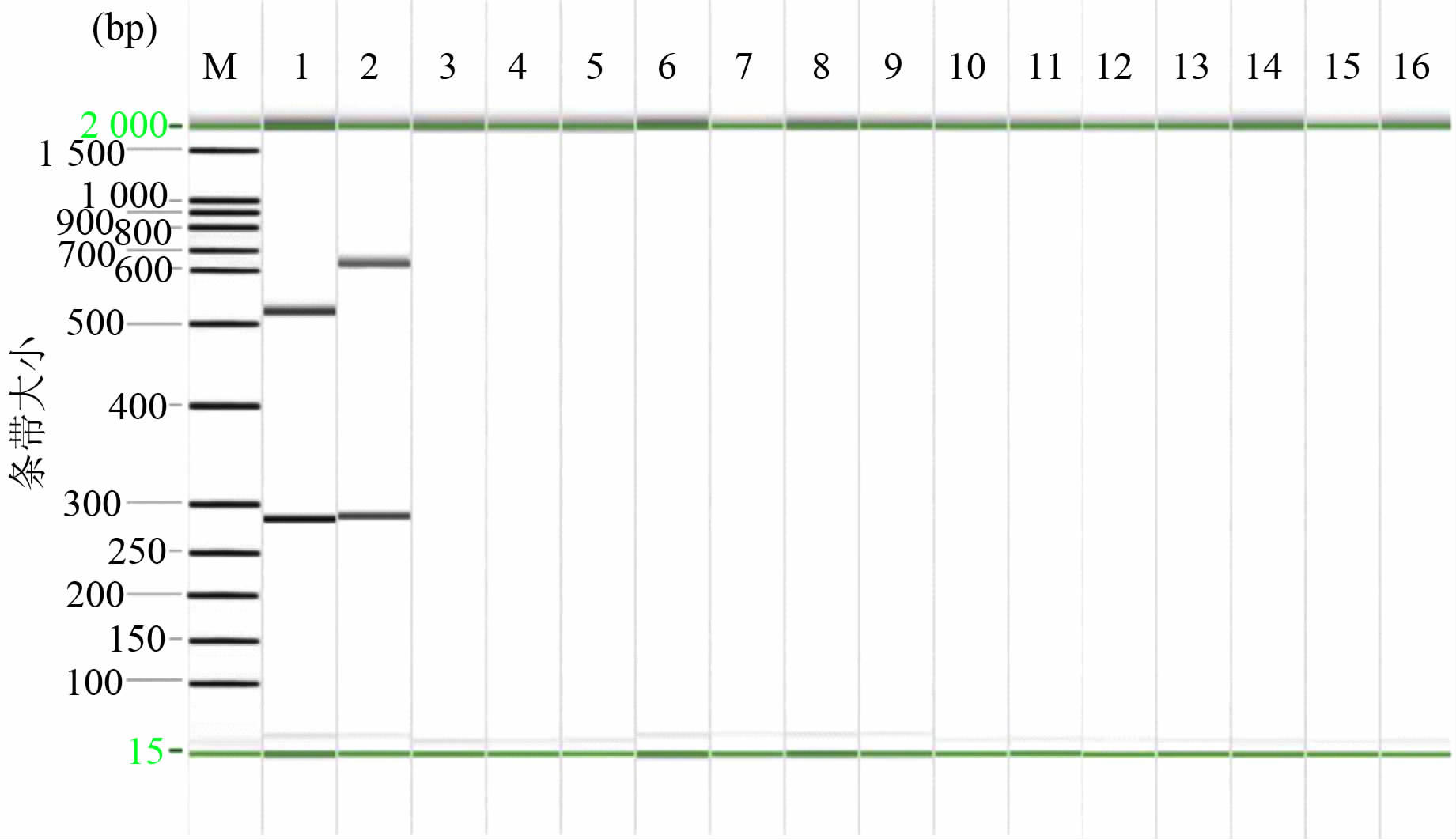
|
| 图 1 引物特异性验证的PCR产物全自动电泳结果 Figure 1 Automated electrophoresis results of PCR products for primer specificity |
| |
本实验对副溶血弧菌临床分离株UP8(tdh与trh阳性)的鉴定灵敏度可达105 CFU/ml。对大流行株的鉴定灵敏度也能达到105 CFU/ml,见图 2。

|
| 图 2 灵敏度验证的PCR产物全自动电泳结果 Figure 2 Automated electrophoresis results of PCR products for specificity |
| |
用建立的多重PCR方法对实验室保存的224株食品分离株和3株临床分离株副溶血弧菌进行鉴定。从5株食品分离株中鉴定到大流行菌群标志toxRS。其中3株为tdh阳性、trh阴性,2株为tdh阴性、trh阴性。血清学鉴定发现除1株tdh阳性株为O2∶KUT型以外,其余4株均为大流行菌群最常见的O3∶K6型。而本研究的临床分离株中未发现tdh、trh和大流行菌群的分子标记物,见表 2和图 3。
| 菌株名称 | toxRS | tdh | trh | 血清分型 | 来源 |
| HBV332 | + | + | - | O2∶KUT | 毛蚶 |
| 13TS027 | + | + | - | O3∶K6 | 生食基围虾 |
| 12BD021 | + | + | - | O3∶K6 | 虹鳟鱼刺身 |
| 10QH024-VP02 | + | - | - | O3∶K6 | 鱼类 |
| FR-14SJ054 | + | - | - | O3∶K6 | 青蛤 |

|
| 图 3 副溶血弧菌分离株鉴定结果 Figure 3 Electrophoresis results in identification of V. parahaemolyticus isolates |
| |
副溶血弧菌广泛存在于水体中和无脊椎动物体内,在食物中具有极高的检出率。其中大流行菌群更是近年来危害食品安全和公共卫生的严重问题。因此高效灵敏地检测副溶血弧菌,并对其致病性进行鉴别,具有重要的现实意义。
多重PCR方法可以通过简单的操作,一次性检出多个检测靶标,因此常用于多病原检测或者单病原多致病基因检测[13-14]。本实验针对副溶血弧菌大流行株的标识基因toxRS及与副溶血弧菌致病的毒力基因trh和tdh设计引物,并优化退火温度等反应条件,实现了副溶血弧菌大流行株及其毒力基因的同步检测。方便操作,快速准确且灵敏度较高,可以满足食品安全和公共卫生工作的大量样本检测需求。
携带tdh或trh的菌株被认为是致病性菌株,目前认定tdh和trh为副溶血弧菌的主要致病因子。通常认为这2个基因在环境分离株中很少出现,但在临床分离株的携带率较高,是鉴别副溶血弧菌致病性最常用的检测标志物。1996年以来高致病性血清型在世界范围内的大流行,使副溶血弧菌的“大流行菌群”引起了更加广泛的关注,这些血清型经过验证,发现是由原始 O3∶K6 菌株通过基因重组或水平转移等方式演变而来的新的变种[15-16]。这些大流行菌株共同的特点是致病性较强、trh阳性、trh阴性。本实验室不仅分别分离到trh阳性、trh阴性的副溶血弧菌大流行株,而且发现了tdh阴性、trh阴性的大流行菌株。此外,血清学鉴定还发现了大流行株罕有报道的O2∶KUT血清型,该型虽然有报道发现于海产品和海洋环境中,但多为tdh阴性和trh阳性[17],在食品卫生监测中该型也通常不引起重视[18],而该分离株的致病基因也符合大流行菌群常见特征(trh阳性、trh阴性,见表 2)。此外,在本研究的临床分离株中,并未检测到大流行菌群标志基因toxRS以及致病基因trh和tdh。
通过本研究可以发现日常食品特别是水产品中存在威胁人群健康的副溶血弧菌大流行菌群。大流行菌群的致病基因分布会发生变化,GS-PCR阳性菌群中也会出现不常见的trh和tdh双阴性菌株。而血清型分布则更为复杂,一些未报道或极少报道与大流行菌群相关的血清型也可能会出现。因此血清型鉴定并不能直观而准确的反应副溶血弧菌的致病性和分类学特征,如本研究中出现的O2∶KUT血清型等。而非大流行菌群的副溶血弧菌也有造成散发的胃肠道疾病的可能。
本报道建立的副溶血弧菌大流行株及毒力基因多重PCR检测方法,实现多个基因的一次性同时扩增,敏感特异,简便有效。可以用于大量样本的快速检测。简便直观的从基因水平反映出副溶血弧菌的致病性等关键特征,满足食品卫生监测的需求。
| [1] | Gutierrez West CK, Klein SL, Lovell CR. High frequency of virulence factor genestdh,trh, andtlh inVibrio parahaemolyticus strains isolated from a pristine estuary[J]. Appl Environ Microbiol , 2013, 79 (7) : 2247–2252. DOI:10.1128/AEM.03792-12 |
| [2] | Shinoda S. Sixty years from the discovery ofVibrio parahaemolyticus and some recollections[J]. Biocontrol Sci , 2011, 16 (4) : 129–137. DOI:10.4265/bio.16.129 |
| [3] | Li L, Wong HC, Nong WY, et al. Comparative genomic analysis of clinical and environmental strains provides insight into the pathogenicity and evolution ofVibrio parahaemolyticus[J]. BMC Genomics , 2014, 15 : 1135. DOI:10.1186/1471-2164-15-1135 |
| [4] | Sakazaki R, Iwanami S, Fukumi H. Studies on the enteropathogenic, facultatively halophilic bacteria,Vibrio parahaemolyticus. Ⅰ. Morphological, cultural and biochemical properties and its taxonomical position[J]. Jpn J Med Sci Biol , 1963, 16 (4) : 161–188. DOI:10.7883/yoken1952.16.161 |
| [5] | Okuda J, Ishibashi M, Hayakawa E, et al. Emergence of a unique O3 : K6 clone ofVibrio parahaemolyticus in Calcutta, India, and isolation of strains from the same clonal group from Southeast Asian travelers arriving in Japan[J]. J Clin Microbiol , 1997, 35 (12) : 3150–3155. |
| [6] | Ansaruzzaman M, Lucas M, Deen JL, et al. Pandemic serovars (O3 : K6 and O4 : K68) ofVibrio parahaemolyticus associated with diarrhea in Mozambique: spread of the pandemic into the African continent[J]. J Clin Microbiol , 2005, 43 (6) : 2559–2562. DOI:10.1128/JCM.43.6.2559-2562.2005 |
| [7] | Bhuiyan NA, Ansaruzzaman M, Kamruzzaman M, et al. Prevalence of the pandemic genotype ofVibrio parahaemolyticus in Dhaka, Bangladesh, and significance of its distribution across different serotypes[J]. J Clin Microbiol , 2002, 40 (1) : 284–286. DOI:10.1128/JCM.40.1.284-286.2002 |
| [8] | Matsumoto C, Okuda J, Ishibashi M, et al. Pandemic spread of an O3 : K6 clone ofVibrio parahaemolyticus and emergence of related strains evidenced by arbitrarily primed PCR andtoxRS sequence analyses[J]. J Clin Microbiol , 2000, 38 (2) : 578–585. |
| [9] | Okura M, Osawa R, Iguchi A, et al. Genotypic analyses ofVibrio parahaemolyticus and development of a pandemic group-specific multiplex PCR assay[J]. J Clin Microbiol , 2003, 41 (10) : 4676–4682. DOI:10.1128/JCM.41.10.4676-4682.2003 |
| [10] | Liu XM. Studies on the techniques for the monitoring and controlling foodborne illness[J]. Chinese Journal of Food Hygiene , 2004, 16 (1) : 3–9. (in Chinese) 刘秀梅. 食源性疾病监控技术的研究[J]. 中国食品卫生杂志 , 2004, 16 (1) : 3–9. |
| [11] | Ning XB, Liu DX, Zhang JL. Pathogenesis and rapid detection ofVibrio parahaemolyticus[J]. Journal of Microbes and Infections , 2008, 3 (1) : 53–56. (in Chinese) 宁喜斌, 刘代新, 张继伦. 副溶血弧菌的致病性及其快速检测[J]. 微生物与感染 , 2008, 3 (1) : 53–56. |
| [12] | Liu XM, Cheng SY, Chen Y, et al. Active surveillance onVibrio parahaemolyticus in retail seafoods from coastal areas of China in 2003[J]. Chinese Journal of Food Hygiene , 2005, 17 (2) : 97–99. (in Chinese) 刘秀梅, 程苏云, 陈艳, 等. 2003年中国部分沿海地区零售海产品中副溶血弧菌污染状况的主动监测[J]. 中国食品卫生杂志 , 2005, 17 (2) : 97–99. |
| [13] | Mahony J, Chong S, Merante F, et al. Development of a respiratory virus panel test for detection of twenty human respiratory viruses by use of multiplex PCR and a fluid microbead-based assay[J]. J Clin Microbiol , 2007, 45 (9) : 2965–2970. DOI:10.1128/JCM.02436-06 |
| [14] | Oliveira DC, de Lencastre H. Multiplex PCR strategy for rapid identification of structural types and variants of themec element in methicillin-resistantStaphylococcusaureus[J]. Antimicrob Agents Chemother , 2002, 46 (7) : 2155–2161. DOI:10.1128/AAC.46.7.2155-2161.2002 |
| [15] | Chen YS, Dai JL, Morris Jr JG, et al. Genetic analysis of the capsule polysaccharide (K antigen) and exopolysaccharide genes in pandemicVibrio parahaemolyticus O3 : K6[J]. BMC Microbiol , 2010, 10 : 274. DOI:10.1186/1471-2180-10-274 |
| [16] | Chao GX, Wang F, Zhou XH, et al. Origin ofVibrio parahaemolyticus O3 : K6 pandemic clone[J]. Int J Food Microbiol , 2011, 145 (2/3) : 459–463. |
| [17] | Caburlotto G, Gennari M, Ghidini V, et al. Serological and molecular characterization ofVibrio parahaemolyticus marine strains carrying pandemic genetic markers[J]. ISME J , 2010, 4 (8) : 1071–1074. DOI:10.1038/ismej.2010.34 |
| [18] | Ju CY, Yu MH, Huang RM, et al. Genetic characterization ofVibrio parahaemolyticus O3 : K6 serovariant isolated in Shenzhen[J]. Chinese Journal of Preventive Medicine , 2015, 49 (1) : 21–25. (in Chinese) 鞠长燕, 俞慕华, 黄锐敏, 等. 深圳市副溶血弧菌O3 : K6血清变异型病原学特征研究[J]. 中华预防医学杂志 , 2015, 49 (1) : 21–25. |
 2016, Vol. 31
2016, Vol. 31


