扩展功能
文章信息
- 张桂, 杨晶, 金东, 卢珊, 徐建国
- ZHANG Gui, YANG Jing, JIN Dong, LU Shan, XU Jian-guo
- 使用两种培养基一种气体条件分离高原鼠兔肠道菌
- Enteric bacteria isolation from plateau pika with two media at the gas condition of 5% CO2
- 疾病监测, 2017, 32(3): 241-246
- Disease Surveillance, 2017, 32(3): 241-246
- 10.3784/j.issn.1003-9961.2017.03.017
-
文章历史
- 收稿日期:2016-11-10
青藏高原是地球上海拔最高、面积最大、形成最晚的高原[1],具有独特的气候和生态环境,如在地质发育和自然演替过程中形成了迥异的高寒草原和草甸生态系统。特殊的环境中生活着多种特有的野生动物,如喜马拉雅旱獭、高原鼠兔等。高原鼠兔是草食性小动物[2],以双子叶植物蒲公英、马先蒿、凤毛菊的花、棘豆的嫩叶以及单子叶禾本科、莎草科的嫩叶为食,属兔形目,栖息于海拔3 200~5 200 m植物疏稀的悬岩乱石堆和农田附近的土坡上,营洞生活,不冬眠,藏医称为“阿鸟那”。鉴定工作高原鼠兔是皮、肉、药、观赏兼备的小动物,成鼠体重200 g,身长12 cm左右,面似兔。
已知许多传染性疾病的病原体来源于动物,如鼠疫耶尔森菌、HIV、埃博拉病毒、马尔堡病毒、布鲁氏菌、包虫等[3-5]。而我国对青藏高原野生动物体内的微生物种群构成及多样性、病原体携带状况的了解很少。已知的仅为旱獭体内携带的鼠疫耶尔森菌可以引起鼠疫,该病为烈性传染病,又称“一号病”、“黑死病”,是我国法定报告的甲类传染病。近几年国内关于高原鼠兔的研究集中在两个方面:高海拔多年生存的低氧适应机制研究[6-8],与青藏高原草原生态系统的相互影响[9-11];而关于高原鼠兔体内微生物组成以及可能携带的病原体所知甚少,这对人兽共患病、自然疫源性疾病以及新发传染病的防治具有一定的隐患。
近年来国内外学者用培养组学的方法对一些动物特定组织微生物菌群进行了大批量的分离。Lagier等[12]尝试用212种培养方式 (不同的培养基组成和培养条件) 对3份人粪便进行分离,最终鉴定117个菌属的340个细菌菌种,其中174个细菌菌种是首次在人粪便中分离获得;Pfleiderer等[13]对神经性厌食症患者的粪便进行了分析,成功分离12 700个菌株,最终鉴定了133个细菌菌种; Bittar等[3]对非洲大猩猩肠道菌群进行研究时共计分离12 800个菌株,最终鉴定了147个细菌菌种,并首次提出微生物培养组学 (microbial culturomics) 的概念,即利用大规模多组合的培养方式从样本中分离获得大量单菌落,通过质谱和/或16S rDNA测序来对菌株进行鉴定。本研究受培养组学的启示:营养丰富的培养基 (brain heart infusion) 适于绝大多数细菌的生长。我们使用脑心浸液羊血培养基对高原鼠兔肠内容物菌属进行分离,了解脑心浸液羊血培养基上所分离的高原鼠兔肠内容物细菌菌属的种类和丰度,为日后开展以培养组学为基础的高原鼠兔肠内容物微生物组学研究提供依据;同时用KF链球菌羊血培养基对高原鼠兔肠道菌进行选择性分离以了解高原鼠兔肠道中是否存在新种链球菌。本研究有助于我们初步了解高原鼠兔肠内容物微生物菌属构成以及可能携带的病原体,同时对人兽共患病、自然疫源性疾病以及新发传染病的防治具有一定的意义。
1 材料与方法 1.1 材料 1.1.1 样品从青海省玉树州国家鼠疫监测点果青沟捕获的青藏高原鼠兔20只。用乙醚麻醉后迅速无菌解剖,取肠内容物,约5~10 g,置15 ml的甘油脑心保存液中,-80 ℃低温冰箱保存,待检。
1.1.2 仪器设备CO2孵箱:Thermo Forma Steri-Cycle CO2incubator; PCR扩增仪:Ferotec Thermal Cycler TC-48/T/H (a); 水平电泳装置:Junyi+600恒压恒流电泳仪; 凝胶成像系统:美国Bio-Rad公司GEL Doc2000;台式离心机:Epeendrof Centrifuge 5415D;恒温金属浴:杭州博日科技有限公司。
1.1.3 培养基及试剂脑心浸液羊血培养基 (BHI)(CM 917,北京陆桥公司):牛心浸粉4.0 g/L,蛋白胨5.0 g/L,酪蛋白胨16.0 g/L,氯化钠5.0 g/L,葡萄糖2.0 g/L,磷酸氢二钠2.5 g/L,琼脂13.5 g/L,添加5%脱纤维羊血;KF链球菌琼脂 (CM 165,北京陆桥公司):麦芽糖0.02 g/L,溴甲酚紫5.0 g/L,氯化钠10.0 g/L,甘油磷酸钠13.0 g/L,琼脂0.1 g/L,TTC 0.4 g/L,叠氮钠,添加5%脱纤维羊血。灭菌的去离子水。脱纤维羊血由北京莱贝斯生物有限公司提供,2×PCR Mix由北京擎科生物有限公司提供;16S测序通用引物27F (5′-AGA GTT TGA TCC TGG CAC AG-3′),1492R (5′-ACG GCT ACC TTG TTA CTC AG-3′)[14]; 20%甘油脑心:100 ml脑心浸液中含20 ml甘油混合均匀,121 ℃,20 min高压灭菌,冷却分装,4 ℃冷藏保存。
1.2 方法 1.2.1 细菌分离培养与鉴定将20份鼠兔肠内容物标本从-80 ℃低温冰箱取出后,于4 ℃环境下缓慢完全解冻后,用灭菌处理的1 ml枪吹吸均匀,吸取20 μl均匀涂布于脑心浸液羊血培养基,在5% CO2气体条件下培养48~72 h后按菌落挑选的原则 (大小、形状、颜色、透明度、边缘整齐度、湿润度、表面光滑与否)[15],每份标本挑取20个左右单菌落,转接于相同的培养基于相同的条件下培养24~48 h。
分离菌株的核酸提取及扩增:在高压灭菌的1.5 ml EP管中加入100 μl灭菌的纯水,用灭菌的接种环挑取菌落纯培养物于EP管中,使液体浸没菌体,然后将其置于98 ℃金属浴中加热10~15 min,加热期间将EP管上下混匀几次,加热完成后离心 (16 000 r/min,2 min),取上清于新的灭菌的EP管中,并做好相应的标记,进行PCR鉴定 (27F/1492R)。PCR反应体系:2×PCR Mix 15 μl;d H2O 13 μl;27F 1 μl;1492R 1 μl。PCR三步法反应条件:95 ℃ 5 min;95 ℃ 45 s,54 ℃ 45 s,72 ℃ 1.5 min,32个循环;72 ℃10 min;4 min停止反应。用1.5%琼脂糖凝胶电泳分离PCR产物。委托北京天一辉远生物科技有限公司完成测序。
1.2.2 新种链球菌的选择性分离根据2.1的实验结果,使用KF链球菌羊血培养基对已分离出链球菌的样本进行选择性分离,后续实验步骤如1.2.1。
1.2.3 序列分析使用DNASTAR软件 (DNASTAR公司,美国) 检查测序质量,将获取的多条序列输出为fas格式文件。通过http://rdp.cme.msu.edu/seqmatch/seqmatch_intro.jsp在线检测序列质量[16]。使用GenBank公共数据库进行序列比对。比对过程中对于命中序列检测其质量,舍弃质量不高的序列。对于比对结果有疑问的序列重新检索以排除GenBank公共数据库中低质量序列对比对结果的影响。结果判定:序列比对的一致性 (即identity) 大于98.7%为已知菌属,即菌株尚可鉴定;取一致性最高者[17];若一致性最高者为Uncultured bacteria而比其一致性低者有已知菌属时,选择已知菌属作为鉴定结果,对于不能鉴定到属的序列则为Uncultured bacteria;一致性大于98.7%且数值相等,而鉴定结果含两个及两个以上种名时,若有模式菌株则定为模式菌株的种名,若无模式菌株则为待鉴定菌株。一致性低于98.7%则为疑似新种。
2 结果 2.1 鼠兔肠内容物细菌的分离鉴定本次试验从高原鼠兔肠道中共分离鉴定了19个属的607株细菌。在属水平上,节杆菌属、肠球菌属、芽孢杆菌属为优势菌属,分别占31.47%、16.14%和15.98%,结果见图 1。有441株细菌可鉴定为31个种,其中Arthrobacter luteolus、Enterococcus faecium为优势菌种,结果见表 1。有166株细菌使用现有的方法无法鉴定到种。其中,有107株细菌的全长16S rDNA序列 (1 500 bp) 和数据库中多条16S rDNA序列的一致性>98.7%,且没有模式菌株序列可供分析,称之为难鉴定菌株。还有59株菌的全长16S rDNA序列和数据库中16S rDNA序列一致性 < 98.7%。可能是新的种,这59株菌可鉴定到8个属,包括节杆菌属、芽孢杆菌属、鲍特菌属、微杆菌属、奈瑟菌属、类芽孢杆菌属、红球菌属。
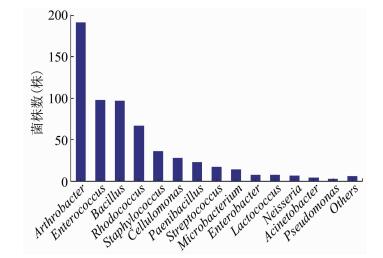
|
| 图 1 从高原鼠兔肠道内容物分离的607株细菌的属水平丰度分析 Figure 1 Abundance at genus level of 607 strains isolated from intestinal contents of plateau pika 注:Others包括丰度低于0.33%的6个属,如Yersinia,Areomicrobium,Bordetella,Corynebacterium,Escherichia,Sphingomonas。 |
| |
| 门 (phylum) | 种 (species) | 百分比 (%) |
| Firmicutes | Enterococcus faecium | 16.78 |
| Bacillus mycoides | 4.08 | |
| Bacillus simplex | 3.85 | |
| Staphylococcus epidermidis | 2.49 | |
| Paenibacillus amylolyticus | 2.27 | |
| Streptococcus ferus | 1.81 | |
| Lactococcus garvieas | 1.81 | |
| Staphylococcus succinus | 1.35 | |
| Enterococcus mundtii | 1.35 | |
| Staphylococcus haemolyticus | 1.13 | |
| Bacillus pumilus | 1.13 | |
| Streptococcus lutetiensis | 0.90 | |
| Streptococcus equinus | 0.67 | |
| Staphylococcus sciuri | 0.67 | |
| Bacillus licheniformis | 0.67 | |
| Staphylococcus equorum | 0.40 | |
| Staphylococcus warneri | 0.22 | |
| Staphylococcus pasteuri | 0.22 | |
| Enterococcus durans | 0.08 | |
| Actinomycetes | Arthrobacter luteolus | 17.67 |
| Rhodococcus coprophilus | 13.61 | |
| Arthrobacter citreus | 13.38 | |
| Arthrobacter gandavensis | 6.70 | |
| Cellulomonas pakistanensis | 2.27 | |
| Cellulomonas flavigena | 0.40 | |
| Rhodococcus zopfii | 0.22 | |
| Proteobacteria | Enterobacter kobei | 2.04 |
| Acinetobacter lwoffi | 0.90 | |
| Yersinia enterocolitica | 0.40 | |
| Enterobacter cloacae | 0.40 | |
| Escherichia coli | 0.22 | |
| 注:黑色加粗菌属为已知致病菌、条件致病菌或具有医学意义的细菌[18]。 | ||
分离的607株细菌属于厚壁菌门、放线菌门和变形菌门,其中厚壁菌门与放线菌门的丰度较高,为优势菌群。用高通量测序的方法对鼠兔盲肠内微生物菌群进行分析,也发现厚壁菌门、拟杆菌门、变形菌门、螺旋体 (>1%);疣微菌门、放线菌门、软皮菌类、蓝藻、酸杆菌 (≤1%)[19-20]。
在分离的31个已知细菌种中,有19种为具有医学意义的细菌,包括致病菌、条件致病菌等[18],占61.29%。如洛菲不动杆菌为条件致病菌,可引起手术后感染,是常见的院感菌之一,曾有儿童败血症的报道[21]。屎肠球菌是医院感染的重要致病菌,可引起尿路感染、腹盆腔感染、败血症和心内膜炎。其他致病菌包括小肠结肠炎耶尔森菌[22]、表皮葡萄球菌、溶血性葡萄球菌等。
2.2 不同培养基菌株分离情况本研究首先利用脑心浸液羊血培养基对20份鼠兔肠内容物标本进行分离,共分离鉴定495株菌,优势菌属是节杆菌属、芽孢杆菌属、红球菌属,分别占38.66%、18.42%和13.56%,见图 2。根据脑心浸液羊血培养基的菌株分离结果,使用KF链球菌羊血选择性培养基对已分离出链球菌的鼠兔肠内容物标本进行选择性分离,最终分离出3个菌属:肠球菌属、链球菌属、葡萄球菌属,在2种培养基上均可生长,见表 2。
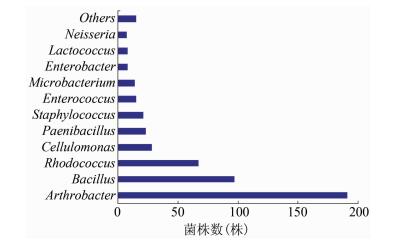
|
| 图 2 脑心羊血培养基分离的495株菌属水平丰度分析 Figure 2 Abundance at genus level of 495 strains isolated with BHI with 5% defibrinated sheep blood 注:Others包括丰度低于0.81%的8个属:Acinetobacter、Pseudomonas、Streptococcus、Yersinia、Areomicrobium、Bordetella、Escherichia、Sphingomonas。 |
| |
| 培养基种类 | 分离的菌属 | ||
| 脑心羊血培养基 | Acinetobacter | Bordetella | Rhodococcus |
| Sphingomonas | Lactococcus | Escherichia | |
| Arthrobacter | Areomicrobium | Staphylococcus | |
| Bacillus | Microbacterium | Streptococcus | |
| Cellulomonas | Neisseria | Pseudomonas | |
| Enterobacter | Paenibacillus | Yersinia | |
| Enterococcus | |||
| KF羊血培养基 | Enterococcus | Streptococcus | Staphylococcus |
| 注:黑色加粗的菌属在2种培养基上均可分离到。 | |||
本研究使用脑心浸液羊血培养基和KF链球菌羊血培养基对高原鼠兔肠道菌进行分离。根据相关文献报道[3],首先使用营养丰富的脑心浸液羊血培养基对20份鼠兔肠内容物标本进行分离,共计分离19个属的495株细菌。同时根据本课题组其他研究人员的结果:已从某些高原野生动物体内分离出多个链球菌新种[23-26],故在脑心浸液羊血培养基菌株分离结果的基础上,我们用KF链球菌羊血选择性培养基对已分离出链球菌属的标本进行选择性分离,共分离鉴定112株细菌,分属于链球菌属、葡萄球菌属、肠球菌属。
本研究以鼠兔肠道菌的16S rDNA全长 (1 500 bp) 分析结果作为鉴定标准,共计分离鉴定19个属的607株细菌,在属水平上,优势菌属是节杆菌、芽孢杆菌属、肠球菌属。Li等[19-20]使用高通量测序分析发现,鼠兔盲肠内微生物菌群的优势菌属是普雷沃菌属、颤螺菌属、瘤胃球菌属。在属水平上,我们的培养结果和高通量分析有差异。
从高原鼠兔肠道内容物分离到19个属的细菌,有441株可鉴定到种水平,分为31个种。其中一些菌种具有医学意义,如Enterobacter kobei、Acinetobacter lwoffi、Yersinia enterocolitica、Staphylococcus sciuri、Staphylococcus equorum、Staphylococcus warneri、Staphylococcus pasteuri、Staphylococcus succinus、Enterococcus mundtii、Staphylococcus haemolyticus、Bacillus pumilus、Enterococcus faecium、Bacillus mycoides、Staphylococcus epidermidis等。
分离的菌株中有166株细菌无法鉴定到种:(1) 16S rDNA全长序列和数据库中多条16S rDNA序列的一致性>98.7%,且没有模式菌株序列可供分析;(2) 序列的一致性<98.7%,可能是新的种。一般情况下,根据16S rDNA序列一致性确定分离的菌株是否为新种的原则为:94.5%≤一致性≤98.7%为一个新种,一致性≤94.5%为新的属。但是在新种鉴定过程中,不同种属之间的一致性值的范围不同,如埃希菌属内种与种亲缘关系很近。Liu等[27]分离命名的旱獭埃希菌 (Escherichia marmotae),其16S rDNA序列分析显示,其与Escherichia fergusonii的一致性最高值为99.3%,但是经过基因组杂交、生化等分析后,所有数据支持其仍为埃希菌属内的一个新种。Hu等[28]分离命名的喜马拉雅螺杆菌 (Helicobacter himalayensis)16S rDNA序列分析的一致性最高值为96.7%也是一个新种。Bai等[23]分离鉴定的藏羚羊链球菌 (Streptococcus pantholopis) 其16S rRNA序列identity最高值为93.4%,链球菌属种与种之间亲缘关系相差较大。
我们分离的16S rDNA序列一致性<98.7%的59株细菌中已有4个属的11株菌可鉴定为5个新种,已得到德国微生物菌种保藏中心 (DSMZ) 和中国普通微生物菌种保藏管理中心 (CGMCC) 的鉴定并已获得菌株号,提示鼠兔肠内容物中存在一定数量的微生物新种。
本研究利用脑心浸液羊血培养基从20份高原鼠兔肠内容物标本中分离鉴定了比较丰富的细菌菌属,为开展以培养组学为基础的高原鼠兔肠内容物菌群研究提供了一定的依据。
作者贡献:
张桂 ORCID:0000-0003-0810-9345
张桂:撰写文章和实验研究
金东、杨晶:给予实验技术指导
卢珊:负责整个样本的采集工作
徐建国:修改文章、最终定稿
| [1] | Xie GD, Lu CX, Leng YF, et al. Ecological assets valuation of the Tibet Plateau[J]. Journal of Natural Resources, 2013, 18(2): 189–196. (in Chinese) 谢高地, 鲁春霞, 冷允法, 等. 青藏高原生态资产的价值评估[J]. 自然资源学报, 2013, 18(2): 189–196. |
| [2] | Chen J, Gao YQ, Huang C, et al. Comparative proteomic analysis of plateau Pika and Wistar rat gastrocnemius mitochondria[J]. Acta Academiae Medicinae Militaris Tertiae, 2007, 29(2): 97–100. (in Chinese) 陈建, 高钰琪, 黄城, 等. 高原鼠兔和Wistar大鼠骨骼肌线粒体的比较蛋白质组学初步研究[J]. 第三军医大学学报, 2007, 29(2): 97–100. |
| [3] | Bittar F, Keita MB, Lagier GC, et al. Gorilla gorilla gorilla gut: a potential reservoir of pathogenic bacteria as revealed using culturomics and molecular tools[J]. Sci Rep, 2014. DOI:10.1038/strep07174 |
| [4] | Hua WH, Wan G, Xu XM, et al. Analysis on epidemic trend of 228 inpatients with brucellosis during 2008-2014[J]. International Journal of Laboratory Medicine, 2016, 37(1): 45–46. (in Chinese) 华文浩, 万钢, 徐新民, 等. 2008-2014年228例布鲁氏菌感染患者流行趋势分析[J]. 国际检验医学杂志, 2016, 37(1): 45–46. |
| [5] | Ding Y, Duan XY, Wang L, et al. Study on the economic burden of disease in patients with hydatid hydatid disease in Xinjiang hospitals[J]. Chinese Journal of Health Statistics, 2013, 30(2): 240–241. (in Chinese) 丁艳, 段新宇, 王乐, 等. 新疆包虫病定点医院包虫病患者经济负担研究[J]. 中国卫生统计, 2013, 30(2): 240–241. |
| [6] | Wang XJ, Wei DB, Wei L, et al. Physiological character of erythrocyte adapting to hypoxia in plateau zokor and plateau pika[J]. Sichuan Journal of Zoology, 2008, 27(6): 1100–1103. (in Chinese) 王晓君, 魏登邦, 魏莲, 等. 高原鼢鼠和高原鼠兔红细胞低氧适应特征[J]. 四川动物, 2008, 27(6): 1100–1103. |
| [7] | Chen QH, Liu FY. Adaptation mechanism of pika (Ochotona curzoniae) to hypoxia[J]. Chinese Journal of Zoology, 2003, 38(5): 109–113. (in Chinese) 陈秋红, 刘凤云. 高原鼠兔低氧适应机制的研究概况[J]. 动物学杂志, 2003, 38(5): 109–113. |
| [8] | Qi XZ, Wang XJ, Zhu SH, et al. Hypoxic adaptation of the hearts of plateau zokor (Myospalax baileyi) and plateau pika (Ochotona curzoniae)[J]. Acta Physiologica Sinica, 2008, 60(3): 348–354. (in Chinese) 齐新章, 王晓君, 朱世海, 等. 高原鼢鼠和高原鼠兔心脏对低氧环境的适应[J]. 生理学报, 2008, 60(3): 348–354. |
| [9] | Zhang XL, Li G. Effects of rodents activities on grazing land and ecosystem in alpine meadow[J]. Pratacultural Science, 2015, 32(5): 816–822. (in Chinese) 张兴禄, 李广. 高原鼠兔和高原鼢鼠在高寒草甸生态系统的作用[J]. 草业科学, 2015, 32(5): 816–822. |
| [10] | Wang FN, Tang MX, Chen Z, et al. Determination of hemogram indicators in plateau pika in different altitude[J]. Chinese Qinghai Journal of Animal and Veterinary Sciences, 2015, 45(2): 15–16. (in Chinese) 王福宁, 唐美霞, 陈志, 等. 不同海拔高原鼠兔的血象指标测定[J]. 青海畜牧兽医杂志, 2015, 45(2): 15–16. |
| [11] | Li NN, Li H, Lei GC. Ecological function of plateau pika (Ochotona curzoniae)[J]. Chinese Journal of Wildlife, 2013, 34(4): 238–242. (in Chinese) 李娜娜, 李华, 雷光春. 高原鼠兔 (Ochotona curzoniae) 的生态功能[J]. 野生动物, 2013, 34(4): 238–242. |
| [12] | Lagier JC, Armougom F, Million M, et al. Microbial culturomics: paradigm shift in the human gut microbiome study[J]. Clin Microbiol Infect, 2012, 18(12): 1185–1193. DOI:10.1111/1469-0691.12023 |
| [13] | Pfleiderer A, Lagier JC, Armougom F, et al. Culturomics identified 11 new bacterial species from a single anorexia nervosa stool sample[J]. Eur J Clin Microbiol Infect Dis, 2013, 32(11): 1471–1481. DOI:10.1007/s10096-013-1900-2 |
| [14] | Neilan BA, Wilton AN, Jacobs D. A universal procedure for primer labelling of amplicons[J]. Nucleic Acids Res, 1997, 25(14): 2938–2939. DOI:10.1093/nar/25.14.2938 |
| [15] | Li GN, Xia XJ, Zhao HH, et al. On intestinal bacteria colony morphology of silkworm and enzyme-producing correlation[J]. Journal of Southwest China Normal University:Natural Science Edition, 2014, 39(12): 53–57. (in Chinese) 李冠楠, 夏雪娟, 赵欢欢, 等. 家蚕肠道细菌菌落形态与产消化酶相关性研究[J]. 西南师范大学学报:自然科学版, 2014, 39(12): 53–57. |
| [16] | Hu SK. The found of Helicobacter himalayensis sp. nov in the gastric mucosa of Marmota himalayana [D]. Beijing: Infectious Disease Prevention and Control Center of China Disease Prevention and Control Center, 2014. (in Chinese) 胡守奎. 喜马拉雅旱獭胃壁新种螺杆菌的发现[D]. 北京: 中国疾病预防控制中心传染病预防控制所, 2014. |
| [17] | Hiergeist A, Gläsner J, Reischl U, et al. Analyses of intestinal microbiota: culture versus sequencing[J]. ILAR J, 2015, 56(2): 228–240. DOI:10.1093/ilar/ilv017 |
| [18] | Woo PCK, Teng JLL, Yeung JMY, et al. Automated identification of medically important bacteria by 16S rRNA gene sequencing using a novel comprehensive database, 16S pathDB[J]. J Clin Microbiol, 2011, 49(5): 1799–1809. DOI:10.1128/JCM.02350-10 |
| [19] | Li H, Li TT, Beasley DE, et al. Diet diversity is associated with beta but not alpha diversity of pika gut microbiota[J]. Front Microbiol, 2016, 7: 1169. |
| [20] | Li H, Li TT, Yao MJ, et al. Pika gut may select for rare but diverse environmental bacteria[J]. Front Microbiol, 2016, 7: 1269. |
| [21] | Yang XS, Sun LX. One case about Acinetobacter sepsis of children[J]. Medical Information, 2011, 24(11): 332. (in Chinese) 杨晓松, 孙凌新. 儿童洛菲不动杆菌败血症1例[J]. 医学信息, 2011, 24(11): 332. |
| [22] | Xiao YC, Liang JR, Gu WP, et al. Investigation on distribution of Yersinia enterocolitica in different animalhosts[J]. Chinese Journal of Zoonoses, 2012, 28(5): 418–420. (in Chinese) 肖玉春, 梁俊容, 古文鹏, 等. 不同动物宿主小肠结肠炎耶尔森氏菌分离及结果分析[J]. 中国人兽共患病学报, 2012, 28(5): 418–420. |
| [23] | Bai XN, Xiong YW, Lu S, et al. Streptococcus pantholopis sp. nov., isolated from faeces of the Tibetan antelope (Pantholopshodgsonii)[J]. Int J Syst Evol Microbiol, 2016, 66(9): 3281–3286. DOI:10.1099/ijsem.0.001189 |
| [24] | Niu LN, Lu S, Hu SK, et al. Streptococcus marmotae sp. nov., isolated from the respiratory tract of Marmota himalayana[J]. Int J Syst Evol Microbiol, 2016, 66(11): 4315–4322. DOI:10.1099/ijsem.0.001350 |
| [25] | Niu LN, Lu S, Lai LH, et al. Streptococcus himalayensis sp. nov., a new streptococcal species isolated from the respiratory tract of Marmota himalayana in Qinghai-Tibet Plateau of China[J]. Int J Syst Evol Microbiol, 2016. DOI:10.1099/ijsem.0.001609 |
| [26] | Niu LN, Lu S, Hu SK, et al. Streptococcus halotolerans sp. nov.isolated from the respiratory tract of Marmota himalayana in Qinghai-Tibet Plateau of China[J]. Int J Syst Evol Microbiol, 2016, 66(10): 4211–4217. DOI:10.1099/ijsem.0.001337 |
| [27] | Liu S, Jin D, Lan RT, et al. Escherichia marmotae sp. nov., isolated from faeces of Marmota himalayana[J]. Int J Syst Evol Microbiol, 2015, 65(7): 2130–2134. DOI:10.1099/ijs.0.000228 |
| [28] | Hu SK, Jin D, Lu S, et al. Helicobacter himalayensis sp. nov.isolated from gastric mucosa of Marmota himalayana[J]. Int J Syst Evol Microbiol, 2015, 65(6): 1719–1725. DOI:10.1099/ijs.0.000163 |
 2017, Vol. 32
2017, Vol. 32


