扩展功能
文章信息
- 储琼芳, 李先平, 华玉婷, 宋丽琼, 肖玉春, 黄元铭, 朱思逸, 任志鸿
- Chu Qiongfang, Li Xianping, Hua Yuting, Song Liqiong, Xiao Yuchun, Huang Yuanming, Zhu Siyi, Ren Zhihong
- TaqMan实时荧光定量PCR检测艰难梭菌
- Detection of Clostridium difficile with TaqMan-based quantitative RT-PCR
- 疾病监测, 2018, 33(5): 417-422
- Disease Surveillance, 2018, 33(5): 417-422
- 10.3784/j.issn.1003-9961.2018.05.015
-
文章历史
- 收稿日期:2017-10-19
艰难梭菌感染(Clostridium difficile infection,CDI)是指由艰难梭菌所致感染,主要通过产生TcdA、TcdB两种毒素而致病。CDI是医院抗生素相关性腹泻的最常见原因之一,其临床表现轻重不一,包括无症状感染、症状较轻的自限性腹泻、症状严重的假膜性结肠炎症、血液感染和组织损伤、甚至中毒巨结肠[1]。据报道无症状的老年人粪便内可检测到产毒艰难梭菌,无症状的带菌者可成为CDI的重要传染源[2]。因此对患者粪便中产毒艰难梭菌的检测,无论是对临床诊断还是对病原监测的传染病防控工作都至关重要。
艰难梭菌(Clostridium difficile)是严格厌氧菌,传统的体外分离培养菌落计数的方法操作繁琐费时,灵敏性和特异性有限,对粪便进行定量检测的准确性欠佳。近年发展的荧光定量PCR方法检测艰难梭菌已在欧美发达国家和中国的大型医院被广泛应用[3]。多数的荧光定量PCR方法是基于已知菌量的纯培养细菌DNA进行梯度稀释后制备标准曲线[2]。由于实际需求是对含菌粪便样本进行定量检测,从含菌粪便标本中提取的细菌DNA存在PCR抑制物,抑制物含量的高低会不同程度地影响PCR的扩增效率。由于不确定用纯菌的DNA做标准曲线定量检测粪便标本的细菌含量,其结果是否可能和粪便样本中的真实含菌量有偏差,因此本研究拟制备2种标准曲线,一是提取定量纯菌DNA后做系列稀释后制备标准曲线,另一个是在等量粪便中分别添加梯度浓度的艰难梭菌后分别提取DNA做标准曲线,应用荧光定量PCR方法评价2种标准曲线哪个更适用于实际工作中粪便中艰难梭菌的准确定量。
艰难梭菌的致病机制目前尚不清楚,人们常常借助建立动物模型来达到研究艰难梭菌发病机理的目的,本研究通过建立CDI模型,验证建立的实时荧光定量PCR方法与传统的艰难梭菌选择性培养计数方法间的差异。
1 材料与方法 1.1 实验材料 1.1.1 实验动物和菌株本实验采用6~8周龄C57BL/6雌鼠,体质量18~20 g,共18只,购自维通利华实验动物有限公司。实验已通过中国疾病预防控制中心(CDC)的实验动物福利和伦理委员会审查。艰难梭菌NAP1/027(ATCC1803)来自中国CDC传染病预防控制所医院感染室的惠赠。动物实验操作严格按照审批的实验动物伦理审查描述的实验方案执行。动物于中国CDC实验动物中心生物安全二级负压环境下饲养,室温20~26 ℃,光照12 h轮换。
1.1.2 主要试剂和仪器艰难梭菌琼脂基础培养基(C. difficile Agar Base)、艰难梭菌选择性添加剂(C. difficile Selective Supplement)、2.5 L厌氧产气袋、DNA粪便提取试剂盒(QIAamp Fast DNA Stool Mini Kit)均购于英国OXOID公司,脱纤维马血购自北京宝特医疗器械有限公司,细菌基因组DNA提取试剂盒(BacteriaGen DNA Kit)购于康为世纪公司,PCR Premix[Premix Ex Taq(Probe qPCR)]购于日本TaKaRa公司,卡那霉素(kanamycin)、庆大霉素(gentamicin)、粘菌素(colistin)、甲硝唑(metronidazole)、克林霉素(clindamycin)均购于中国药品检定所,万古霉素(vancomycin)购于美国INALCOL公司。PCR引物(tcdA - f:TCT ACC ACT GAA GCA TTA C;tcdA - r:TAG GTA CTG TAG GTT TAT TG;tcdA - Probe:GGA TTT TGA ATC TCT TCC TCT AGT AGC)[5],由北京生工生物工程股份有限公司合成,ABI7500Fast实时荧光定量PCR仪购自美国ABI公司。
1.2 实验方法 1.2.1 艰难梭菌的培养将冻存的艰难梭菌NAP1/ 027复苏后,挑选若干菌落接种于5 ml BHI液体培养基,37 ℃厌氧环境下,180 r/min振荡培养24 h,振荡混匀,按照20%的接种量转接于含有5 ml BHI液体培养基的养菌管中。37 ℃厌氧环境下,180 r/ min振荡培养20 h。收集艰难梭菌培养菌液,4 000 r/ min离心10 min,弃上清后用PBS重悬。取1 ml重悬后的菌液进行倍比稀释,涂板厌氧培养对纯培养艰难梭菌进行活菌计数。
1.2.2 艰难梭菌TaqMan实时荧光定量PCR检测体系建立总反应体系为20 μl,包括正、反向引物(250 nmol/L)、探针(500 nmol/L)、TaqMan mix 10 μl、细菌DNA模板2 μl、DyeⅡ 0.4 μl。荧光定量PCR扩增条件:95 ℃ 30 s,及40个循环的95 ℃ 10 s和55 ℃ 30 s。以Ct值< 35的扩增作为细菌检测的阳性结果。
1.2.3 艰难梭菌TaqMan实时荧光定量PCR方法的敏感性将上述培养、计数的艰难梭菌4 000 r/min离心10 min,弃上清,根据细菌基因组DNA提取试剂盒说明书,提取细菌DNA,将提取的细菌DNA进行10倍梯度稀释,以检测建立的实时荧光定量PCR方法能够检出的最小细菌量,分析该方法的灵敏度。
1.2.4 艰难梭菌TaqMan实时荧光定量PCR方法的特异性以艰难梭菌、干酪乳杆菌、植物乳杆菌、发酵乳杆菌、德氏乳杆菌、嗜酸乳杆菌、鼠李糖杆菌(LGG)、Domla乳杆菌(Domla)的DNA为模板,同上条件进行实时荧光定量PCR反应,分析该方法的特异性。
1.2.5 检测艰难梭菌的2种标准曲线的制备与评价(1)纯菌DNA系列稀释制备标准曲线取1 ml菌液含108 CFU提取DNA,对提取的DNA进行梯度浓度1:10的倍比稀释,共稀释5个梯度。(2)基于粪便混合艰难梭菌的DNA制备的标准曲线收集小鼠粪便200 mg加入到1 ml的PBS中制备粪便悬液,取100 μl该粪便悬液分装到5个EP管中。每管分别加入倍比稀释的109~105 CFU菌液100 μl。按照DNA粪便提取试剂盒说明书提取粪便DNA。应用荧光定量PCR定量检测上述各管中的艰难梭菌含量。以细菌菌落形成单位(CFU)的对数值为横坐标,荧光定量PCR的Ct值为纵坐标制作标准曲线。
1.2.6 艰难梭菌感染小鼠模型的建立C57BL/6小鼠随机分为对照组和CDI模型组。对照组小鼠饮用正常水。如图 1所示,从-7 d开始,CDI组小鼠连续5 d饮用混合抗生素水溶液,混合抗生素水溶液含卡那霉素(0.4 mg/ml),庆大霉素(0.035 mg/ml),粘菌素(850 U / ml),甲硝唑(0.215 mg / ml),万古霉素(0.045 mg/ml)。从-2 d起停用抗生素水溶液改换普通饮用水直至实验结束,-1 d腹腔注射克林霉素(20 mg/kg)[6],0 d对实验组小鼠灌胃处理,每只小鼠灌胃量为200 μl PBS(含有106 CFU艰难梭菌)。感染后的5 d内,每天观察小鼠神态,如有无弓背、腹泻(湿尾)或死亡情况,每天收取小鼠的粪便,进行活菌计数和荧光定量PCR定量检测,并对2种不同计数方法进行统计分析(用SPSS 17.0软件进行t检验),P < 0.05为差异有统计学意义。

|
| 图 1 难艰梭菌感染模型建立流程图 Figure 1 Flowchart of CDI mouse model establishment 注:以感染小鼠当天为0 d,感染之前用“-”表示“,-7”表示感染之前的7 d。 |
| |
结果显示,TaqMan实时荧光定量PCR检测方法具有较佳的线性范围,当细菌浓度为1.05×103时,平均 Ct值为34.17,当浓度降到1.05×102时,平均Ct值达到37.59,一般Ct值>35认为检测结果阴性,表明该检测方法的检测限可达到103 CFU。由于提取细菌基因组DNA后的溶解体积为100 μl,取2 μl进行定量PCR反应,因此本方法对艰难梭菌的检测下限为10 CFU/PCR反应。当细菌浓度为10 CFU时,所建立的实时荧光定量PCR方法仍能够产生良好的特异性荧光扩增曲线(图 2),进一步说明该检测方法具有非常高的灵敏性。

|
| 图 2 艰难梭菌TaqMan实时荧光定量PCR敏感性检测结果 Figure 2 Sensitivity of TaqMan probe based TR-PCR for C. difficile |
| |
TaqMan实时荧光定量PCR检测方法对艰难梭菌的检测结果为阳性,Ct值为25.17,出现了特异性的荧光扩增曲线;而对干酪乳杆菌、植物乳杆菌、发酵乳杆菌、德氏乳杆菌、嗜酸乳杆菌、LGG、Domla的检测结果均为典型的阴性反应,均未见特异性的荧光扩增曲线。结果表明,该方法检测艰难梭菌有较强的特异性,见图 3。

|
| 图 3 艰难梭菌TaqMan实时荧光定量PCR特异性检测结果 Figure 3 Specificity of TaqMan probe based TR-PCR for C. difficile |
| |
在纯菌浓度为1.05×102/ml~ 1.05×108/ml时,对应的Ct值显示检测下限为1.05× 103/ml(表 1)。由于提取细菌基因组DNA后的溶解体积为100 μl,取2 μl进行定量PCR反应,因此本方法对艰难梭菌的检测下限为10 CFU/PCR反应。通过10×倍比稀释的标准品3次平行结果,由细菌对数值及其对应Ct值绘制的标准曲线的r绝对值为0.999 8(y=-3.400 4x+38.602 0),见图 4。
| 项目 | 1.05×108 | 1.05×107 | 1.05×106 | 1.05×105 | 1.05×104 | 1.05×103 | 1.05×102 |
| 样本 | 17.08 | 20.64 | 23.68 | 27.25 | 30.42 | 33.95 | 38.87 |
| 复孔1 | 17.09 | 20.67 | 23.90 | 27.25 | 30.58 | 33.96 | 36.93 |
| 复孔2 | 17.16 | 20.72 | 23.79 | 27.16 | 30.73 | 34.60 | 36.96 |
| 均值 | 17.11 | 20.68 | 23.79 | 27.22 | 30.58 | 34.17 | 37.59 |

|
| 图 4 艰难梭菌浓度对数值及其对应Ct值的标准曲线 Figure 4 Log koc of C. difficile concentration and standard curve of corresponding Ct value |
| |
厌氧培养活菌并计数,确认5份等量粪便匀浆中混合已知的艰难梭菌量依次为104~108 CFU。提取粪便中的DNA后,通过荧光定量PCR检测每份含菌粪便中的艰难梭菌数量,并绘制出细菌含量(CFU)对数值与Ct值的标准曲线,与纯菌DNA倍比稀释(对应的细菌浓度范围分别是104~108)绘制的标准曲线对比。如图 5所示,两者的标准曲线几乎重合,无明显差异。根据上述艰难梭菌标准曲线可知,实时荧光定量PCR检测艰难梭菌的下限为10 CFU/PCR反应,而混有艰难梭菌的粪便质量为20 mg(200 mg / ml × 100 μl),因此,该标准曲线检测粪便中艰难梭菌的检测下限为103 CFU/g粪便。
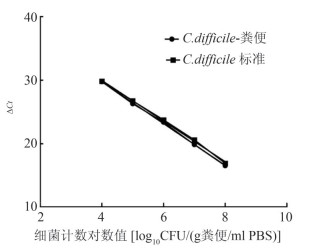
|
| 图 5 纯菌培养标准曲线和粪便中相同数量细菌标准曲线的比较 Figure 5 Analytical curve of the C. difficile-spiked stool(●) compared with the analytical curve of C. difficile pure culture(■)at same serial dilutions |
| |
分别应用传统的活菌分离培养计数的方法和实时荧光定量PCR,对小鼠感染后第1~5天收集的粪便进行艰难梭菌定量检测。结果表明小鼠感染后的第1天细菌即成功定植,定植菌量的对数值为(6.17±0.25)。尽管第2和第3天感染小鼠体重呈现一过性显著降低,但粪便中艰难梭菌数量却在感染后的5 d内都没有显著变化。且2种不同的方法对小鼠粪便中艰难梭菌的计数结果无显著差异,见图 6。该结果表明本研究给予的感染剂量可成功建立CDI小鼠模型,整个急性感染期细菌稳定定植没有显著波动,且腹泻症状轻重和体重减轻与粪便中细菌数量没有相关性,表明宿主症状改善体重恢复也不能彻底清除艰难梭菌定植,也揭示了无症状携带艰难梭菌宿主作为潜在传染源的危害。

|
| 图 6 2种方法检测艰难梭菌感染小鼠粪便中艰难梭菌的含量比较 Figure 6 Comparison of two different method detecting C. difficile counts in feces of CDI mice |
| |
艰难梭菌是严格厌氧菌,在进行艰难梭菌活菌计数的实验过程中,要求对粪便样本及时进行涂板计数厌氧培养,要求及时将样本放入厌氧设备,样本暴露在空气中时间的长短往往会影响计数结果。在选择性培养基上进行活菌计数时,由于平板上杂菌很多,并不能将艰难梭菌菌落一一特异识别,这 可能会造成读数不够精确。一般情况下,定量PCR法检测粪便样本中的细菌含量,是根据已知浓度纯培养艰难梭菌的标准曲线进行Ct值和细菌计数的换算得来的。有文献表明粪便标本中提取的DNA存在抑制物[2],导致定量PCR得出的Ct值比实际值要高,可能会导致检测粪便样本中的目标菌株的数量比实际的偏低。为了明确用纯菌做标准曲线是否是最佳方法,本实验通过比较相同数量的细菌混在粪便里和纯培养的情况下,分别提取DNA,采用定量PCR做出细菌计数的对数值与Ct值的对应曲线,结果发现两者并无差异。证实了此次实验粪便标本中提取的DNA不存在对艰难梭菌的扩增产生抑制的物质,可以运用纯菌梯度浓度曲线,直接准确的计算出小鼠粪便中的艰难梭菌含量。本实验证明用纯菌培养和粪便中的艰难梭菌两种方法做标准曲线,检测模拟带菌粪便和CDI模型中的艰难梭菌计数没有显著差异。证明了用纯菌培养的艰难梭菌做标准曲线结果可靠,能够简化实验过程,减少工作量,提高工作效率。
由于抗生素破坏了肠道菌群平衡,致使宿主失去了抵御致病菌定植的能力,因此抗生素的使用被认为是CDI的首要危险因素[7-9]。本研究选用的CDI模型,利用抗生素混合物破坏了C57BL/6小鼠肠道菌群,为艰难梭菌的感染定植创造了条件。该模型中的小鼠感染后保持持续稳定的带菌状态,该CDI小鼠模型中的症状特征表现为腹泻、体重减轻以及肠道的组织损伤,这与人的感染症状类似[10],因此该模型可应用于CDI机制和干预的研究。针对人感染艰难梭菌的不同程度的感染症状,可以在小鼠模型中通过控制感染小鼠的菌量来达到相似的感染效果,并且可以进一步发展艰难梭菌的复发感染模型[9]。有文献报道运用该小鼠模型探究CDI发展过程中肠道菌群的变化,结果通过比较表现有严重临床症状的小鼠与对CDI不敏感的小鼠,肠道菌群存在明显的差异[11]。而这些实验都离不开对粪便中菌量的定时监测,以便及时评估肠道内致病菌菌量和疾病程度之间的相关性。
本研究首先证实了利用纯菌制备RT-PCR标准曲线能准确定量地检测小鼠粪便样本中的艰难梭菌含量,继而建立小鼠感染模型,同时采用活菌计数以及RT⁃PCR两种方法检测粪便中的艰难梭菌含量。此次实验中的活菌计数,采用的是每个实验组依次分别计数,增加了实验时间成本和试剂耗材成本。而菌量计数结果表明两种方法检测结果并无显著的差异,说明进一步实验考虑到实验的经济性和准确性,可以单独选择RT⁃PCR法,这对提高实验效率具有重要的实际意义。本研究建立的荧光定量PCR方法和CDI小鼠模型将用于进一步研究评价更安全有效的新组合微生态制剂,以达到类似粪便移植有效清除CDI的效果。
作者贡献:
储琼芳 ORCID:0000-0003-0943-0624
储琼芳:实验设计和实验操作,论文撰写
任志鸿、李先平、宋丽琼:实验设计,技术指导以及论文修改
华玉婷、肖玉春、黄元铭、朱思逸:协助完成实验操作
| [1] |
Sun XM, Hirota SA. The roles of host and pathogen factors and the innate immune response in the pathogenesis of Clostridium difficile infection[J]. Mol Immunol, 2015, 63(2): 193-202. DOI:10.1016/j.molimm.2014.09.005 |
| [2] |
Kubota H, Sakai T, Gawad A, et al. Development of TaqMan-based quantitative PCR for sensitive and selective detection of toxigenic Clostridium difficile in human stools[J]. PLoS One, 2014, 9(10): e111684. DOI:10.1371/journal.pone.0111684 |
| [3] |
Sloan LM, Duresko BJ, Gustafson DR, et al. Comparison of real-time PCR for detection of thet cdC gene with four toxin immunoassays and culture in diagnosis of Clostridium difficile infection[J]. J Clin Microbiol, 2008, 46(6): 1996-2001. DOI:10.1128/JCM.00032-08 |
| [4] |
Best EL, Freeman J, Wilcox MH. Models for the study of Clostridium difficile infection[J]. Gut Microbes, 2012, 3(2): 145-167. DOI:10.4161/gmic.19526 |
| [5] |
Bélanger SD, Boissinot M, Clairoux N, et al. Rapid detection of Clostridium difficile in feces by real-time PCR[J]. J Clin Microbiol, 2003, 41(2): 730-734. DOI:10.1128/JCM.41.2.730-734.2003 |
| [6] |
Chen XH, Katchar K, Goldsmith JD, et al. A mouse model of Clostridium difficile-associated disease[J]. Gastroenterology, 2008, 135(6): 1984-1992. DOI:10.1053/j.gastro.2008.09.002 |
| [7] |
Britton RA, Young VB. Interaction between the intestinal microbiota and host in Clostridium difficile colonization resistance[J]. Trends Microbiol, 2012, 20(7): 313-319. DOI:10.1016/j.tim.2012.04.001 |
| [8] |
Buffie CG, Pamer EG. Microbiota-mediated colonization resistance against intestinal pathogens[J]. Nat Rev Immunol, 2013, 13(11): 790-801. DOI:10.1038/nri3535 |
| [9] |
Yurist-Doutsch S, Arrieta MC, Vogt SL, et al. Gastrointestinal microbiota-mediated control of enteric pathogens[J]. Annu Rev Genet, 2014, 48: 361-382. DOI:10.1146/annurev-genet-120213-092421 |
| [10] |
Hutton ML, Mackin KE, Chakravorty A, et al. Small animal models for the study of Clostridium difficile disease pathogenesis[J]. FEMS Microbiol Lett, 2014, 352(2): 140-149. DOI:10.1111/1574-6968.12367 |
| [11] |
Reeves AE, Theriot CM, Bergin IL, et al. The interplay between microbiome dynamics and pathogen dynamics in a murine model of Clostridium difficile Infection[J]. Gut Microbes, 2011, 2(3): 145-158. DOI:10.4161/gmic.2.3.16333 |
 2018, Vol. 33
2018, Vol. 33


