Distribution of IS CR1in clinical strains and its relationship with blaPER-1gene
CHEN Xia1, LI Gui-xi2, YU Hui-lan1, YUAN Min1, HOU Xiao-ping2, LI Juan1
State Key Laboratory for Communicable Disease Control and Prevention, Institute for Communicable Disease Control and Prevention, Chinese Centre for Disease Control and Prevention, Beijing 102206, China
Abstract
Objective To understand the distribution of blaPER-1 gene and IS CR1in clinical isolates of different species and the relationships between IS CR1and multidrug resistance, and between IS CR1and blaPER-1 gene. Methods Clinical isolates were collected from a hospital in Hebei province during 2011-2012. The species of the isolates were identified by VITEK® 2 COMPACT automatic microbial identification system and 16S rDNA sequence analysis. The distributions of IS CR1, blaPER-1 gene and IS CR1-blaPER-1 were detected by PCR. Results A total of 314 clinical isolates of different species were collected. IS CR1and blaPER-1 gene were detected in 28.7% and 12.4% of the isolates, respectively. IS CR1-blaPER-1 sequence structure was detected in 1 Proteus vuigaris strain and 21 Acinetobacter spp. strains. Up to 96.7% of IS CR1contained strains were multidrug resistant, and 93.3% of them were resistant to all 9 antibiotics. Conclusion IS CR1was distributed in various species of clinical isolates widely, which was related with multidrug resistance in clinical bacteria. blaPER-1 gene was distributed in various species of clinical isolates. IS CR1might facilitate the spread of blaPER-1 gene among Acinetobacter spp. and other bacteria species.
插入序列共同区在多种临床菌株间的分布及其与超广谱β-内酰胺酶基因blaPER-1的关系研究
陈霞1, 李桂喜2, 禹惠兰1, 袁敏1, 侯小平2, 李娟1
1. 中国疾病预防控制中心传染病预防控制所, 细菌耐药室, 传染病预防控制国家重点实验室, 北京 102206;
2. 中国人民解放军第二五一医院检验科
2. 中国人民解放军第二五一医院检验科
摘要
目的 研究临床来源的多种类细菌中插入序列共同区(insertion sequence common regions,ISCRs)及超广谱β-内酰胺酶基因blaPER-1的分布情况,分析可移动元件IS CR1 与细菌多药耐药表型及blaPER-1基因散播的关系。 方法 收集2011-2012年河北省某医院临床独立菌株,应用梅里埃VITEK® 2 COMPACT全自动微生物生化鉴定系统结合16S rDNA序列分析进行种属鉴定,利用普通聚合酶链反应(polymerase chain reaction,PCR)方法进行IS CR1、blaPER-1和IS CR1-blaPER-1间区的扩增;分析IS CR1、blaPER-1种属分布状况及IS CR1与菌株耐药性的关系。 结果 共得到17种(属)314株临床独立菌株,其中IS CR1携带率为28.7%(90/314),在不动杆菌和肠杆菌科的多种细菌中携带较多;携带IS CR1的菌株中,96.7%(87/90)为多药耐药菌株,93.3%(84/90)耐受9类及以上抗菌药物;blaPER-1基因携带率为12.4%(39/314),在7种细菌检出,不动杆菌属细菌中检出最多(31株);21株不动杆菌属细菌和1株普通变形杆菌存在IS CR1-blaPER-1结构。 结论 IS CR1在多种细菌中广泛分布,细菌多药耐药表型和其携带有一定关系;blaPER-1基因在多种细菌中散播,IS CR1的基因捕获和移动机制可能造成了blaPER-1基因在不动杆菌属细菌中及在不同种细菌间的散播。
内容大纲
-
1 材料与方法
- 1.1 实验材料
- 1.2 菌株的分离及鉴定
- 1.3 菌株药物敏感性试验结果判定
- 1.4 耐药基因扩增用细菌基因组的提取
- 1.5 相关基因的检测
- 1.5.1 blaPER-1基因的检测
- 1.5.2 可移动元件IS CR1 的检测
- 1.5.3 blaPER-1-IS CR1 间区检测
- 2.1 收集细菌的种属分布情况
- 2.2 IS CR1 分布情况
- 2.3 IS CR1 携带菌株的多药耐药情况
- 2.4 耐药基因blaPER-1分布情况
- 2.5 blaPER-1基因与IS CR1 间区扩增情况
1 材料和方法
1.1 实验材料 脑心浸液肉汤培养基及脑心浸液琼脂培养基购于北京陆桥生物技术有限公司;细菌DNA提取使用QIANamp细菌基因组DNA提取试剂盒和普通聚合酶链反应(PCR)扩增酶(Taq PCR MasterMix),购于天根生化科技(北京)有限公司;长片段扩增PCR保真扩增酶(2×Lamp MasterMix)均购于北京康为世纪生物科技有限公司产物胶回收试剂盒(Qiaquick Gel Extraction Kit)购于德国QIAGEN公司;产物测序产物连接试剂盒(pEASYTM-T1克隆试剂盒)购于北京全式金生物技术有限公司;DNA分子质量(Marker BM5000)购于北京博迈德科技发展有限公司。
1.2 菌株的分离及鉴定
314株独立菌株分离自河北省某医院2011 - 2012年不同门诊及住院患者的送检标本,标本类型包括血液、尿液、痰液、分泌物、粪便、穿刺液、脑脊液、胆汁、咽拭子、胸水等。不同样本经过临床检验科微生物接种培养程序进行接种后,对培养呈阳性的样本菌株应用梅里埃VITEK 2 COMPACT全自动微生物生化鉴定系统结合16S rDNA序列分析进行种属鉴定。16S rDNA序列利用细菌16S rDNA通用引物27F(5′-AGA GTT TGA TCA TGG CTC AG-3′)和1492R(5′-TAC GGT TAC CTT GTT ACG ACT T-3′)进行扩增。扩增反应条件如下:94 ℃预变性10 min,94 ℃变性30 s,55.5 ℃退火30 s,72 ℃延伸 1 min,30个循环,72 ℃终延伸4 min。扩增得到的产物和载体连接后送北京擎科新业生物技术有限公司进行测序,并和GenBank数据库进行Blast比对。
确定种属的样本菌株挑选单克隆,接种脑心浸液琼脂培养基进行纯培养,37 ℃培养18~24 h后,生长的菌苔分别收集到1 ml含有灭菌20%甘油-肉汤培养基的冻存管中,-80 ℃长期保存。
1.3 菌株药物敏感性试验结果判定
应用梅里埃VITEK 2 COMPACT全自动微生物生化鉴定系统进行临床常见13种抗菌药物敏感性测试。13种抗菌药物按照美国临床与实验室标准委员会(CLSI)推荐的2011年版进行药物分类,共分为9类。不同种类细菌耐药、中介敏感和敏感的结果判定标准参考CLSI 2011年版判定标准[14],见表1。由于CLSI 2011中缺乏部分细菌对药物的结果判定参考折点,故选择相关细菌的相关折点进行参考。如缺乏铜绿假单胞菌、不动杆菌和其他非肠杆菌科细菌头孢唑林和氨苄西林参考折点,则采用肠杆菌科细菌相应折点参考;缺乏不动杆菌属细菌氨曲南参考折点,采用其他非肠杆菌科细菌相应折点参考;缺乏铜绿假单胞菌和其他非肠杆菌科细菌氨苄西林/舒巴坦参考折点,采用肠杆菌科细菌相应折点参考;缺乏铜绿假单胞菌复方新诺明参考折点,采用其他非肠杆菌科细菌相应折点参考;缺乏铜绿假单胞菌四环素参考折点,采用其他非肠杆菌科细菌相应折点参考。当一类药物中包含多种受试药物时,细菌对该类药物中任何一种药物耐药均判定为对该类药物耐药。细菌对3类及以上种类药物耐药判定为多药耐药细菌。
1.4 耐药基因扩增用细菌基因组的提取
314株分离株细菌冻存株全部分别接种脑心浸液琼脂培养基,37 ℃复苏24 h后,挑选单克隆再接种脑心浸液琼脂培养基进行传代纯培养,37 ℃纯培养18~24 h。收集适量菌体,按照商品化细菌基因组提取试剂盒使用说明进行操作,得到的基因组DNA样本于-40 ℃冻存备用。
1.5 相关基因的检测
1.5.1 blaPER-1基因的检测
超广谱β-内酰胺酶编码基因blaPER-1用引物blaPER-1-F(5′-GTA GTT ACT GCC TCG ACG CT-3′)和引物blaPER-1-R (5′-TCA TTG GTT CGG CTT GA-3′)进行普通PCR扩增。扩增反应条件如下:94 ℃预变性2 min,94 ℃变性30 s,54.6 ℃退火30 s,72 ℃延伸45 s,30个循环,72 ℃终延伸4 min。获得预期阳性产物为830 bp。部分阳性扩增产物胶回收后经和载体连接后送北京擎科新业生物技术有限公司进行测序,并和GenBank数据库进行Blast比对。
表1 药物敏感性试验受试药物选择及不同细菌结果判定折点(1)
Table 1 Antimicrobial agents breakpoints cited from CLSI,2011 μg/ml
| 药物类别 | 药物名称 | 肠杆菌科 | 铜绿假单胞菌 | |||||
| S | I | R | S | I | R | |||
| 氨基糖苷类 | 庆大霉素 | ≤4 | 8 | ≥16 | ≤4 | 8 | ≥16 | |
| 碳青霉烯类 | 亚胺培南 | ≤1 | 2 | ≥4 | ≤4 | 8 | ≥16 | |
| 头孢烯类 | 头孢唑林 | ≤2 | 4 | ≥8 | ≤2 | 4 | ≥8 | |
| 头孢他啶 | ≤4 | 8 | ≥16 | ≤8 | 16 | ≥32 | ||
| 头孢吡肟 | ≤8 | 16 | ≥32 | ≤8 | 16 | ≥32 | ||
| 单环内酰胺类 | 氨曲南 | ≤4 | 8 | ≥16 | ≤8 | 16 | ≥32 | |
| 青霉素类 | 氨苄西林 | ≤8 | 16 | ≥32 | ≤8 | 16 | ≥32 | |
| 哌拉西林 | ≤16 | 32~64 | ≥128 | ≤64 | - | ≥128 | ||
| β-内酰胺/β-内酰胺抑制剂类 | 氨苄西林/舒巴坦 | ≤8/4 | 16/8 | ≥32/16 | ≤8/4 | 16/8 | ≥32/16 | |
| 哌拉西林/他唑巴坦 | ≤16/4 | 32/4~64/4 | ≥128/4 | ≤64/2 | - | ≥128/4 | ||
| 磺胺类 | 复方新诺明 | ≤2/38 | - | ≥4/76 | ≤2/38 | - | ≥4/76 | |
| 氟喹诺酮类 | 环丙沙星 | ≤1 | 2 | ≥4 | ≤1 | 2 | ≥4 | |
| 四环素类 | 四环素 | ≤4 | 8 | ≥16 | ≤4 | 8 | ≥16 | |
| 药物类别 | 药物名称 | 不动杆菌属 | 其他非肠杆菌科 | |||||
| S | I | R | S | I | R | |||
| 氨基糖苷类 | 庆大霉素 | ≤4 | 8 | ≥16 | ≤4 | 8 | ≥16 | |
| 碳青霉烯类 | 亚胺培南 | ≤4 | 8 | ≥16 | ≤4 | 8 | ≥16 | |
| 头孢烯类 | 头孢唑林 | ≤2 | 4 | ≥8 | ≤2 | 4 | ≥8 | |
| 头孢他啶 | ≤8 | 16 | ≥32 | ≤8 | 16 | ≥32 | ||
| 头孢吡肟 | ≤8 | 16 | ≥32 | ≤8 | 16 | ≥32 | ||
| 单环内酰胺类 | 氨曲南 | ≤8 | 16 | ≥32 | ≤8 | 16 | ≥32 | |
| 青霉素类 | 氨苄西林 | ≤8 | 16 | ≥32 | ≤8 | 16 | ≥32 | |
| 哌拉西林 | ≤16 | 32~64 | ≥128 | ≤16 | 32~64 | ≥128 | ||
| β-内酰胺/β-内酰胺抑制剂类 | 氨苄西林/舒巴坦 | ≤8/4 | 16/8 | ≥32/16 | ≤8/4 | 16/8 | ≥32/16 | |
| 哌拉西林/他唑巴坦 | ≤16/4 | 32/4~64/4 | ≥128/4 | ≤16/4 | 32/4~64/4 | ≥128/4 | ||
| 磺胺类 | 复方新诺明 | ≤2/38 | - | ≥4/76 | ≤2/38 | - | ≥4/76 | |
| 氟喹诺酮类 | 环丙沙星 | ≤1 | 2 | ≥4 | ≤1 | 2 | ≥4 | |
1.5.2 可移动元件IS CR1 的检测
与多种耐药基因获得和移动相关的元件IS CR1 利用引物IS CR1 -F(5′-ATG TCG CTG GCA AGG AAC GC-3′)和引物IS CR1 -R(5′-AGA CGA CTC TGT GAT GG ATC-3′)进行筛查。扩增反应条件如下:94 ℃预变性2 min,94 ℃变性30 s,56.5 ℃退火30 s,72 ℃延伸1 min,30个循环,72 ℃终延伸4 min。获得的预期阳性产物约为 1450 bp。挑选部分阳性产物经和载体连接后送北京擎科新业生物技术有限公司进行测序,并和GenBank数据库进行Blast比对。
1.5.3 blaPER-1-IS CR1 间区检测
blaPER-1基因筛查呈阳性的菌株,利用引物IS CR1 -F和blaPER-1-R进行长片段PCR扩增。扩增反应条件如下:94 ℃预变性2 min,94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸3 min,30个循环,72 ℃终延伸4 min。获得预期阳性产物约为2800 bp。所有阳性扩增产物胶回收后经和载体连接后送北京擎科新业生物技术有限公司进行测序,并和GenBank数据库进行Blast比对。
2 结果
2.1 收集细菌的种属分布情况 收集的314株临床分离细菌分布在17个种(属),包括革兰阴性菌309株,革兰阳性菌5株,具体的菌株分布情况见表2。其中分离得到最多的是不动杆菌属细菌(62株)和大肠埃希菌(57株),肺炎克雷伯菌、铜绿假单胞菌、产酸克雷伯菌和阴沟肠杆菌分离也较多,其他11种细菌检出量较少。
2.2 IS CR1 分布情况
314株多种类临床分离株中共检出IS CR1 携带菌株90株,携带率为28.7%,分布在包括不动杆菌属、大肠埃希菌、肺炎克雷伯菌、粘质沙雷菌、阴沟肠杆菌、产酸克雷伯菌、弗氏枸橼酸杆菌、奇异变形杆菌、普通变形杆菌、产气肠杆菌、摩根氏摩根氏菌、液化沙雷菌、布氏枸橼酸杆菌、温和气单胞菌在内的14个不同种属的细菌中。其中检出最多的是不动杆菌属细菌(37/62,59.7%),肺炎克雷伯菌、粘质沙雷菌和阴沟肠杆菌检出也较多(表2)。
表2 314株不同种类临床分离细菌IS CR1 和blaPER-1检出情况
Table 2 Detection results of IS CR1 and blaPER-1 gene in 314 clinical isolates of different species
| 细菌名称 | 细菌数 | IS CR1 阳性 | blaPER-1阳性 | |||
| 株数 | 阳性率(%) | 株数 | 阳性率(%) | |||
| 不动杆菌属 | 62 | 37 | 59.7 | 31 | 50.0 | |
| 大肠埃希菌 | 57 | 3 | 5.3 | 2 | 3.5 | |
| 肺炎克雷伯菌 | 48 | 12 | 25.0 | 1 | 2.1 | |
| 铜绿假单胞菌 | 46 | 0 | 0.0 | 0 | 0.0 | |
| 产酸克雷伯菌 | 24 | 6 | 25.0 | 2 | 8.3 | |
| 阴沟肠杆菌 | 21 | 10 | 47.6 | 0 | 0.0 | |
| 粘质沙雷菌 | 13 | 10 | 76.9 | 0 | 0.0 | |
| 弗氏枸橼酸杆菌 | 8 | 2 | 25.0 | 0 | 0.0 | |
| 奇异变形杆菌 | 6 | 1 | 16.7 | 0 | 0.0 | |
| 产气肠杆菌 | 6 | 3 | 50.0 | 0 | 0.0 | |
| 摩根氏摩根氏菌 | 5 | 1 | 20.0 | 0 | 0.0 | |
| 液化沙雷菌 | 4 | 2 | 50.0 | 0 | 0.0 | |
| 表皮葡萄球菌 | 5 | 0 | 0.0 | 1 | 20.0 | |
| 嗜麦芽寡养单胞菌 | 3 | 0 | 0.0 | 0 | 0.0 | |
| 普通变形杆菌 | 3 | 1 | 33.3 | 1 | 33.3 | |
| 布氏枸橼酸杆菌 | 2 | 1 | 50.0 | 0 | 0.0 | |
| 温和气单胞菌 | 1 | 1 | 100.0 | 1 | 100.0 | |
| 合计 | 314 | 90 | 28.7 | 39 | 12.4 | |
2.3 IS CR1 携带菌株的多药耐药情况
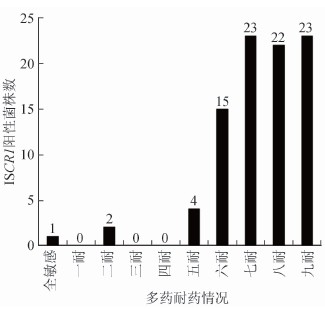
在携带IS CR1 基因的90株细菌分离株中,2株对受试的9类13种抗菌药物表现出全部敏感或中介敏感,1株为两重耐药菌株,96.7%(87/90)为多药耐药菌株。超过90%的菌株(84/90)耐受6类及以上抗菌药物 (简称六耐,以下类推),其中有15株细菌同时耐受6 类抗菌药物,23株细菌同时耐受7类抗菌药物,22株细菌同时耐受8类抗菌药物,23株细菌对受试的9类抗菌药物全部耐药,见图1。
图1 携带IS CR1 菌株与多药耐药表型对应关系
Figure 1 Relationship between IS CR1 positive isolates and multidrug resistance phenotypes
2.4 耐药基因blaPER-1分布情况
在314株多种属临床菌株中,携带有blaPER-1基因的菌株有39株,占总分离菌株的12.4%,包括不动杆菌属细菌31株,大肠埃希菌2株,产酸克雷伯菌2株,表皮葡萄球菌1株,肺炎克雷伯菌1株,温和气单胞菌1株,普通变形杆菌1株,不动杆菌属细菌占总检出菌株的79.5%(31/39),见表2、图2。
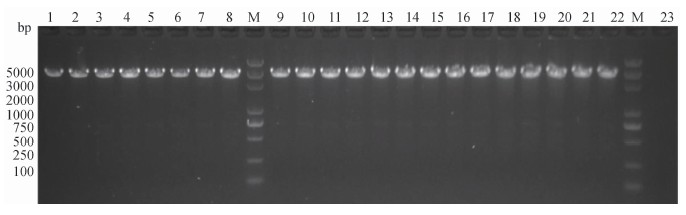
图2 IS CR1 -blaPER-1间区结构的PCR产物的扩增电泳图
Figure 2 Agarose electrophoresis results of PCR amplification products of IS CR1 -blaPER-1 in 22 bacteria isolates 注:电泳条件:150 V,25 min,1.5%TAE琼脂糖凝胶GelRed染色,上样量3 μl; 泳道1~8,10~22为鲍曼不动杆菌IS CR1 -blaPER-1间区结构扩增电泳结果; 泳道9为普通变形杆菌IS CR1 -blaPER-1间区结构扩增电泳结果; 泳道23为不含IS CR1 -blaPER-1间区结构的阴性对照电泳结果;M为DNA分子质量Marker BM5000。
2.5 blaPER-1基因与IS CR1 间区扩增情况
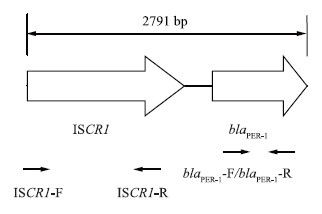
在39株携带有blaPER-1的细菌中,22株细菌用IS CR1 的上游引物IS CR1 -F和blaPER-1下游引物blaPER-1-R扩增出片段大小约2800 bp的产物片段(图2),其中包括21株不动杆菌属细菌和1株普通变形杆菌。序列拼接及分析显示,其与GenBank中blaPER-1-IS CR1 间区结构完全一致,见图3。
3 讨论
3.1 IS CR1 在不同菌种间的散布及其与多药耐药表型的关系 IS CR1 (或orf513)是最早发现的ISCR元件,通常与耐药基因相连,位于两段1类整合子的
图3 IS CR1 -blaPER-1间区结构及PCR引物策略示意图
Figure 3 Diagram of IS CR1 -blaPER-1 sequence structure and PCR primer strategy
3′保守区中间,且紧接在缩短的3′保守区的下游,介导对包括氯霉素类、甲氧苄啶类、喹诺酮类、氨基糖苷类耐药基因以及A 类和C 类β-内酰胺类等多类抗菌药物的耐受,分布在大肠埃希菌、克雷伯菌、枸橼酸杆菌等多种革兰阴性菌中[1]。最近报道的质粒介导的喹诺酮类耐药基因qnrA及氨基糖苷类16S rRNA甲基化酶基因armA均与IS CR1 有关[1]。IS CR1 基因是ISCR其中一型,是目前发现分布广泛,和耐药因子转移密切相关,研究较为深入的可移动元件之一 。本研究从314株多种(属)临床细菌分离株中检测到90株IS CR1 携带株,它们分布于14种(属)细菌中,该结果佐证了IS CR1 拥有复杂的菌种分布的特征,也丰富了IS CR1 在我国多种(属)细菌中广泛散布的数据。在90株IS CR1 携带株中,89株为肠杆菌科细菌(克雷伯菌属、肠杆菌属、沙雷菌属、枸橼酸杆菌属、变形杆菌属等)或不动杆菌属细菌,提示IS CR1 可能与临床肠杆菌科和不动杆菌的耐药相关,应广泛开展其IS CR1 流行分布的调查研究,深入研究相关耐药机制,以降低IS CR1 相关的多种类耐药基因散播的风险。
多药耐药细菌、泛耐药细菌甚至全耐药细菌越来越困扰临床对抗细菌感染治疗,不同类型的耐药基因借由各种基因捕获或移动系统在同种或者不同种细菌间广泛播散,提高了多药耐药细菌产生和传播的潜在风险。本研究将携带IS CR1 的菌株与其多药耐药表型进行比对分析发现,IS CR1 的携带情况与细菌多药耐药表型间存在一定联系:携带IS CR1 几乎全部为多药耐药菌株,90%的菌株耐受超过8类抗菌药物,25.5%的菌株(23/90)对9类受试的抗菌药物全部耐药,极少数菌株为两重耐药(1/90)。只有2株敏感/中介敏感菌耐药菌也携带有IS CR1 ,可能由于细菌本身与IS CR1 相关的某些耐药基因不足以提高细菌MIC值表现为耐药表型,之前有关文献也有报道[15]。提示作为和耐药基因捕获、转移密切相关的IS CR1 携带情况和细菌多药耐药表型之间关系密切,应将IS CR1 作为今后进行多药耐药细菌防控研究的重要因子,以期为降低多药耐药细菌的产生和散播风险提供基础数据。
3.2 blaPER-1基因在不同菌种间的分布
blaPER-1基因是编码ESBLs基因的一种,编码的PER-1介导对第三代头孢菌素,如头孢他啶、头孢噻肟及头孢哌酮等药物的耐药性[16]。blaPER-1基因1993年在法国首先报道[5],在我国北京、江西、广东、山东均有零星报道,在韩国、希腊、法国、土耳其、意大利等国家也有少量报道 。目前blaPER-1基因在鲍曼不动杆菌、铜绿假单胞菌、粪产碱菌中有较多报道,在其他种细菌中发现和报道非常少 。本研究对来自同一地区多种类细菌进行检测发现,blaPER-1基因在7种(属)39株细菌中散播,主要在不动杆菌属细菌(31/39)中存在,在大肠埃希菌、表皮葡萄球菌、温和气单胞菌、克雷伯菌属细菌、普通变形杆菌中也有零星检出,这显示该基因在临床细菌主要分布菌种上的相对单一性,也表明该基因能够在多种(属)细菌中存在,提示应加强对其流行分布研究的重视。另外在受试的不动杆菌属细菌中50%的菌株(31/62)携带有blaPER-1基因,有报道指出PER-1是介导鲍曼不动杆菌对第三代头孢菌素产生耐药的重要酶类[14],本研究结果佐证了已有研究的结论。国外有报道曾在铜绿假单胞菌中发现存在blaPER-1基因,但本研究中分离的46株铜绿假单胞菌中并未发现该基因的存在。
3.3 IS CR1 -blaPER-1间区结构在不同菌种间的散布
报道指出IS CR1 与多种耐药基因的获得和散播有关,曾有国外研究显示在鲍曼不动杆菌和铜绿假单胞菌blaPER-1基因的上游存在有IS CR1 样结构,推测blaPER-1基因的获得和移动与IS CR1 及其构成的相关结构有关 。本研究发现21株不动杆菌属细菌和1株普通变形杆菌中含有IS CR1 -blaPER-1间区结构,这提示IS CR1 不但和blaPER-1基因在不动杆菌属细菌中的流行相关,而且很可能与blaPER-1基因的跨种属散播有关。不动杆菌属细菌和普通变形杆菌是临床常分离到的病原微生物,IS CR1 的参与会造成blaPER-1基因散播范围扩大,给应用第三代头孢菌素类药物进行抗感染治疗带来困扰。
综上所述,IS CR1 作为一种新被发现和逐渐认识的耐药基因捕获和移动元件,其流行分布研究在我国尚在起步,本研究为其在临床多种类细菌中的流行情况提供了可靠的流行病学数据。本研究结果提示,与多种耐药基因捕获和移动有关的IS CR1 可能与我国临床多药耐药细菌、泛耐药细菌的产生和散播有一定关系,应加强对IS CR1 流行病学研究和介导耐药的分子机制研究,为临床防控多药耐药细菌的散播提供基础分析数据。本研究结果还提示blaPER-1基因可借由IS CR1 在不动杆菌属细菌中传播,也可跨种属散播,为临床应用第三代头孢菌素类药物进行抗感染治疗和深入研究IS CR1 侧翼序列基因环境提供基础数据。
参考文献
[1] Toleman MA,Bennett PM,Walsh TR. ISCR elements:novel gene-capturing systems of the 21st century[J].Microbiol Mol Biol Rev,2006,70(2):296-316.
[2] Xia R,GuoX,Zhang Y,et al. qurVC-like gene located in a novel complex class 1 integron harboring the IS CR1 element in an Aeromonas punctata strain from an aquatic environment in Shandong province,China[J].Antimicrob Agents Chemother,2010,54(8):3471-3474.
[3] Lee CH, Liu JW, Li CC, et al. Spread of ISCR1 elements containing blaDHA-1 and multiple antimicrobial resistance genes leading to increase of flomoxef resistance in extended-spectrum-β-lactamase-producing Klebsiella pneumoniae[J]. Antimicrob Agents Chenmother,2011,55(9):4058-4063.
[4] Zhang L, Huang LY. Progress in research of extended-spectrum β-lactamases in Gram-negative bacillus[J].Chinese Journal of Nosocomiology,2008,18(6):897-900. (in Chinese) 张玲,黄留玉.革兰氏阴性杆菌产超广谱β-内酰胺酶研究进展[J].中华医院感染学杂志,2008,18(6):897-900.
[5] Nordmann P,Ronco E,Naas T,et al. Characterization of a novel extended-spectrum β-lactamase from Pseudomonas aeruginosa[J].Antimicrob Agents Chemother,1993,37:962-969.
[6] Vahaboglu H,Oztürk R,Aygün G,et al. Widespread detection of PER-1-type extended-spectrumβ-lactamases among nosocomial Acinetobacter and Pseudomonas aeruginosa isolates in Turkey:a nationwide multicenter study[J].Antimicrob Agents Chemother,1997,41:2265-2269.
[7] Corvec S,Caroff N,Espaze E,et al. AmpC cephalosporinase hyperproduction in Acinetobacter baumannii clinical strains[J].J Antimicrob Chemother,2003,52:629-635.
[8] Yong D,Shin JH,Kim S,et al. High prevalence of PER-1 extended-spectrum β-lactamase-producing Acinetobacter spp. in Korea[J].Antimicrob Agents Chemother,2003,47:1749-1751.
[9] Pagani L,Mantegoli E,Migliavacca R,et al. Multifocal detection of multidrug-resistant Pseudomonas aeruginosa producing the PER-1 extended-spectrum β-lactamase in Northern Italy[J].J Clin Microbiol,2004,42:2523-2529.
[10] Naas T,Bogaerts P,Bauraing C,et al. Emergence of PER and VEB extended-spectrum β-lactamases in Acinetobacter baumannii in Belgium[J]. J Antimicrob Chemother,2006,58:178-182.
[11] Naas T, Kernbaum S, Allali S, et al. Multidrug-resistant Acinetobacter baumannii, Russia[J]. Emerg Infect Dis,2007,13:669-671.
[12] Yamano Y, Nishikawa T, Fudimura T,et al. Occurrence of PER-1-producing clinical isolates of Pseudomonas aeruginosa in Japan and their susceptibility to doripenem[J]. J Antibiot,2006,59:791-796.
[13] Wang H,Guo P,Sun H,et al. Molecular epidemiology of clinical isolates of carbapenem-resistant Acinetobacter spp. from Chinese hospitals[J]. Antimicrob Agents Chemother,2007,51:4022-4028.
[14] Wayne PA. Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing; Twenty-first informational supplement [S]. Pennsylvania,USA:Clinical and Laboratory Standards Institute,2011.
[15] Park CH,Robicsek A,Jacoby GA,et al. Prevalence in the United States of aac(6')-Ib-cr encoding a ciprofloxacin-modifying enzyme. Antimicrob Agents Chemother,2006,50(11):3953-3955.
[16] Lee Y,Bae IK,Kim J,et al. Dissemination of ceftazidime-resistant Acinetobacter baumannii clonal complex 92 in Korea[J]. J Appl Microbiol,2012,112:1207-1211.
[17] Nordmann P,Naas T. Sequence analysis of PER-1 extended-spectrum beta-lactamase from Pseudomonas aeruginosa and comparison with class A beta-lactamases[J]. Antimicrob Agents Chemother,1994,38(1):104-114.
[18] Xia R,GuoX,Zhang Y,et al. qurVC-like gene located in a novel complex class 1 integron harboring the ISCR1 element in an Aeromonas punctata strain from an aquatic environment in Shandong province,China[J]. Antimicrob Agents Chemother,2010,54(8):3471-3474.
[19] Poiel L,Cabanne H,Vahaboglu,et al. Genetic environment and expression of the extended-spectrum beta-lactamase blaPER-1 gene in gram-negative bacteria[J]. Antimicrob Agents Chemother,2005,49(5):1708-1713.
[2] Xia R,GuoX,Zhang Y,et al. qurVC-like gene located in a novel complex class 1 integron harboring the IS CR1 element in an Aeromonas punctata strain from an aquatic environment in Shandong province,China[J].Antimicrob Agents Chemother,2010,54(8):3471-3474.
[3] Lee CH, Liu JW, Li CC, et al. Spread of ISCR1 elements containing blaDHA-1 and multiple antimicrobial resistance genes leading to increase of flomoxef resistance in extended-spectrum-β-lactamase-producing Klebsiella pneumoniae[J]. Antimicrob Agents Chenmother,2011,55(9):4058-4063.
[4] Zhang L, Huang LY. Progress in research of extended-spectrum β-lactamases in Gram-negative bacillus[J].Chinese Journal of Nosocomiology,2008,18(6):897-900. (in Chinese) 张玲,黄留玉.革兰氏阴性杆菌产超广谱β-内酰胺酶研究进展[J].中华医院感染学杂志,2008,18(6):897-900.
[5] Nordmann P,Ronco E,Naas T,et al. Characterization of a novel extended-spectrum β-lactamase from Pseudomonas aeruginosa[J].Antimicrob Agents Chemother,1993,37:962-969.
[6] Vahaboglu H,Oztürk R,Aygün G,et al. Widespread detection of PER-1-type extended-spectrumβ-lactamases among nosocomial Acinetobacter and Pseudomonas aeruginosa isolates in Turkey:a nationwide multicenter study[J].Antimicrob Agents Chemother,1997,41:2265-2269.
[7] Corvec S,Caroff N,Espaze E,et al. AmpC cephalosporinase hyperproduction in Acinetobacter baumannii clinical strains[J].J Antimicrob Chemother,2003,52:629-635.
[8] Yong D,Shin JH,Kim S,et al. High prevalence of PER-1 extended-spectrum β-lactamase-producing Acinetobacter spp. in Korea[J].Antimicrob Agents Chemother,2003,47:1749-1751.
[9] Pagani L,Mantegoli E,Migliavacca R,et al. Multifocal detection of multidrug-resistant Pseudomonas aeruginosa producing the PER-1 extended-spectrum β-lactamase in Northern Italy[J].J Clin Microbiol,2004,42:2523-2529.
[10] Naas T,Bogaerts P,Bauraing C,et al. Emergence of PER and VEB extended-spectrum β-lactamases in Acinetobacter baumannii in Belgium[J]. J Antimicrob Chemother,2006,58:178-182.
[11] Naas T, Kernbaum S, Allali S, et al. Multidrug-resistant Acinetobacter baumannii, Russia[J]. Emerg Infect Dis,2007,13:669-671.
[12] Yamano Y, Nishikawa T, Fudimura T,et al. Occurrence of PER-1-producing clinical isolates of Pseudomonas aeruginosa in Japan and their susceptibility to doripenem[J]. J Antibiot,2006,59:791-796.
[13] Wang H,Guo P,Sun H,et al. Molecular epidemiology of clinical isolates of carbapenem-resistant Acinetobacter spp. from Chinese hospitals[J]. Antimicrob Agents Chemother,2007,51:4022-4028.
[14] Wayne PA. Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing; Twenty-first informational supplement [S]. Pennsylvania,USA:Clinical and Laboratory Standards Institute,2011.
[15] Park CH,Robicsek A,Jacoby GA,et al. Prevalence in the United States of aac(6')-Ib-cr encoding a ciprofloxacin-modifying enzyme. Antimicrob Agents Chemother,2006,50(11):3953-3955.
[16] Lee Y,Bae IK,Kim J,et al. Dissemination of ceftazidime-resistant Acinetobacter baumannii clonal complex 92 in Korea[J]. J Appl Microbiol,2012,112:1207-1211.
[17] Nordmann P,Naas T. Sequence analysis of PER-1 extended-spectrum beta-lactamase from Pseudomonas aeruginosa and comparison with class A beta-lactamases[J]. Antimicrob Agents Chemother,1994,38(1):104-114.
[18] Xia R,GuoX,Zhang Y,et al. qurVC-like gene located in a novel complex class 1 integron harboring the ISCR1 element in an Aeromonas punctata strain from an aquatic environment in Shandong province,China[J]. Antimicrob Agents Chemother,2010,54(8):3471-3474.
[19] Poiel L,Cabanne H,Vahaboglu,et al. Genetic environment and expression of the extended-spectrum beta-lactamase blaPER-1 gene in gram-negative bacteria[J]. Antimicrob Agents Chemother,2005,49(5):1708-1713.
|
扩展功能
|
|
| 本文信息 | |
| PDF全文 | |
| HTML全文 | |
| 参考文献 | |
| 服务与反馈 | |
| 加入引用管理器 | |
| 引用本文 | |
| Email Alert | |
| 本文作者相关文章 | |
| 陈霞 | |
| 李桂喜 | |
| 禹惠兰 | |
| 袁敏 | |
| 侯小平 | |
| 李娟 | |
| PubMed | |
| Article by CHEN Xia | |
| Article by LI Gui-xi | |
| Article by YU Hui-lan | |
| Article by YUAN Min | |
| Article by HOU Xiao-ping | |
| Article by LI Juan | |