Multiple-locus variable-number tandem repeats analysis typing of Shiga toxin-producing Escherichia coli isolates
ZHAO Ai-lan, BAI Xiang-ning, MENG Qiong, LIU Kai, XIONG Yan-wen
State Key Laboratory for Communicable Diseases Prevention and Control, Institute for Communicable Disease Prevention and Control, Chinese Center for Disease Control and Prevention, Beijing 102206, China
Abstract
Objective To evaluate the performance of multiple-locus variable number tandem repeats analysis (MLVA) in typing of Shiga toxin producing Escherichia coli (STEC) isolates, and understand the molecular epidemiological characteristics of the stains. Methods Totally 70 STEC isolates were typed by MLVA using 7 VNTR loci. A dendrogram was constructed using BioNumerics based on allele numbers. Results Using the MLVA protocol, 46 MLVA types were obtained from 70 STEC isolates. O157 and non-O157 isolates were divided into A and B groups. Isolates of different sources, serotypes or Shiga toxin gene types are well divided with some exceptions. Conclusion Though it needs to be improved, the MLVA protocol based on 7 VNTR loci is useful to differentiate STEC isolates, to trace the source of outbreak and so on.
Keywords:
Shiga toxin producing Escherichia coli
multiple locus variable number tandem repeats analysis
molecular typing
产志贺毒素大肠埃希菌分离株多位点可变数目串联重复序列分析
赵爱兰, 白向宁, 孟琼, 刘凯, 熊衍文
中国疾病预防控制中心传染病预防控制所, 传染病预防控制国家重点实验室, 北京 102206
摘要
目的 探讨多位点可变数目串联重复序列分析[multiple-locus VNTR (variable-number tandem repeats) analysis,MLVA]分型方法对产志贺毒素大肠埃希菌分离株的分型效果,初步了解菌株分子流行病学特征。 方法 采用7个VNTR位点对70株产志贺毒素大肠埃希菌分离株进行分析,并根据多态性位点的重复数目利用BioNumerics软件进行聚类分析。 结果 70株菌株被分为46种MLVA型别,O157与非O157菌株可分为A、B两个明显不同的群。除少部分菌株外,不同宿主来源、志贺毒素型别及血清型的非O157菌株与MLVA型别存在较好的相关性。 结论 7个VNTR位点的MLVA分型方案对于产志贺毒素大肠埃希菌的菌株分型、暴发溯源等具有一定的参考价值,但仍需进一步完善。
内容大纲
-
1 材料与方法
- 1.1 材料
- 1.2 方法
- 1.2.1 细菌基因组DNA制备及引物合成
- 1.2.2 目的VNTR位点PCR扩增
- 1.2.3 PCR扩增产物的检测与分析
- 2.1 VNTR 各位点多态性分析
- 2.2 MLVA分型分析
- 2.3 不同MLVA型别菌株的血清型情况
- 2.4 不同MLVA型别菌株携带志贺毒素基因情况
- 2.5 不同宿主来源STEC菌株的MLVA分型情况
STEC的宿主以牛、羊等反刍类动物为主,欧洲研究报道显示健康牛的STEC携带率为10%~50%,健康羊的携带率为36%~67%[7],这些动物可作为人类感染的重要传染源。我国目前没有详细的非O157 STEC人感染情况和动物携带情况及菌株特点分析等相关数据报道,但加强非O157 STEC感染的实验室检测和菌株分子流行病特征分析,有助于及时发现暴发疫情,追溯感染来源从而及时控制疫情。美国疾病预防控制中心(CDC)建立了以脉冲场凝胶电泳(pulse-field gel electrophoresis,PFGE)技术为核心的实验室分子分型监测网络——PulseNet,并正在发展多位点可变数目串联重复序列分析 (variable-number tandem repeats) analysis,MLVA]的第二代监测技术,其中有些病原菌如STEC O157 ∶ H7的MLVA分型方案已经建立[8]。Lindstedt等[9]筛查了一些新的VNTR位点,建立了能用于EIEC、ETEC、EPEC、EAEC、STEC及志贺菌分型分析的MLVA方法。本研究对分离自我国不同地区、不同宿主来源以及不同血清型和毒力基因特征的STEC分离株进行了MLVA分析,初步探讨了该方法的分型能力、我国STEC分离株的分子流行病学特征,为包括非O157 STEC在内的监测和防控提供分子流行病学依据。
1 材料与方法
1.1 材料 1.1.1 菌株来源
本研究中70株STEC代表性菌 株分别分离自腹泻患者粪便(8株)、羊粪(25株)、牦牛粪(18株)、猪粪或猪肠内容物(14株)、肉牛粪(4株)以及鸡粪(1株),其中O157 ∶ H7菌株13株,均为国家监测点的监测菌株,非O157 STEC菌株57株,均为本实验室分离。菌株分离时间跨度为1988-2012年,标本来源地包括北京、天津、江苏、河南、山东、湖南、四川、黑龙江、青海、贵州等。所有菌株经API 20E生化条鉴定为大肠埃希菌,血清型鉴定使用丹麦血清研究院(Statens Serum Institut, Denmark)和天津生物芯片有限公司生产血清套装,鉴定步骤按照说明书进行。多重PCR方法对菌株进行stx1/stx2毒力基因检测[8]。参考菌株为STEC O157 ∶ H7 EDL933。
1.1.2 主要试剂与仪器
细菌基因组提取试剂盒(Bacteria Gene DNA kit)购自康为世纪公司。Taq DNA聚合酶、10×PCR buffer、dNTPs 及DL2000 DNA Marker购自大连TaKaRa公司;PCR仪(SensoQuest Labcycler)购自德国Senso公司。
1.2 方法
1.2.1 细菌基因组DNA制备及引物合成
严格按照DNA提取试剂盒说明书提取DNA模板,置-20 ℃保存备用。根据参考文献[10],合成CVN001、CVN002、CVN003、CVN004、CVN007、CVN0014、CVN0015 共7个VNTR位点的引物,并在上游引物5′末端标记相应荧光报告基团,见表1。引物由生工生物工程(上海)有限公司合成。
表1 7个VNTR位点的引物序列、重复序列长度和重复序列数目(O157 ∶ H7 EDL933为参考菌株)信息表
Table 1 Primer sequences, repeat length and copy numbers in E. coli O157 ∶ H7 EDL933
| 引物名称 | 引物序列 | 重复序列长度(bp) | 重复序列数目 |
| CVN001-F | 5′-HEX-AAC CGG CTG GGG CGA ATC C-3′ | 39 | 6 |
| CVN001-R | 5′-GGC GGC GGT GTC AGC AAA TC-3′ | ||
| CVN002-F | 5′-TET-AAC CGT TAT GAA RGR AAG TCC T-3′ | 18 | 12 |
| CVN002-R | 5′-TCG CCC AGT AAG TAT GAA ATC-3′ | ||
| CVN003-F | 5′-6FAM-AAA AAT CCG GAT GAG WTG GTC-3′ | 15 | 2 |
| CVN003-R | 5′-TTG CGT TGT CAG TAA TTT GTT CAG-3′ | ||
| CVN004-F | 5′-HEX-MGC TGC GGC RCT GAA GAA GA-3′ | 15 | 3 |
| CVN004-R | 5′-CCC GGC AGG CGA AGC ATT GT-3′ | ||
| CVN007-F | 5′-6FAM-ACC GTG GCT CCA GYT GAT TTC-3′ | 18 | 4 |
| CVN007-R | 5′-ACC AGT GTT GCG CCC AGT GTC-3′ | ||
| CVN014-F | 5′-6FAM-TCC CCG CAA TCA GCA AMA CAA AGA-3′ | 6 | 4 |
| CVN014-R | 5′-GCA GCR GGG ACA ACG GAA GC-3′ | ||
| CVN015-F | 5′-TET-TAG GCA TAG CGC ACA GAC AGA TAA-3′ | 6 | 3 |
| CVN015-R | 5′-GTA CCG CCG AAC TTC AAC ACT C-3′ |
1.2.2 目的VNTR位点PCR扩增
采用聚合酶链反应技术(Polymerase Chain Reaction, PCR)扩增各VNTR位点,PCR反应采用4个反应体系,体系一包括:10×buffer 2.0 μl、dNTP 0.125 μmol/L、CVN001和CVN003上、下游引物各0.5 μmol/L、Taq DNA聚合酶0.25 μl (1.25 U)、DNA模板1 μl,补充蒸馏水至20 μl;体系二包括引物CVN004和CVN014;体系三包括CVN007和CVN015,体系四包括CVN002,引物浓度和PCR体系均同体系一。PCR 循环条件为:94 ℃预变性5 min;94 ℃变性45 s,体系一、二、三采用63 ℃退火45 s,体系四 50 ℃退火45 s,72 ℃延伸60 s,共30个循环;最后72 ℃延伸10 min。
1.2.3 PCR扩增产物的检测与分析
PCR扩增产物送北京擎科生物技术有限公司,采用3730XL测序仪进行毛细管电泳,利用GeneScan 600 Liz DNA marker对扩增产物的大小进行校准和确认,GeneMapper v4.0分析软件进行片段大小分析,根据各位点片段大小计算各位点的重复次数,使用 BioNumerics (Version 4.0)分析软件进行聚类分析。
2 结果
2.1 VNTR 各位点多态性分析 本研究中CVN001、CVN002、CVN003、CVN004、CVN007、CVN0014、CVN0015共7个VNTR位点在13株STEC O157 ∶ H7菌株中的重复序列数目分别为1、3、2、2、2、5、2;在57株非O157 STEC菌株中重复序列数目分别为8、4、4、6、1、17、1。位点CVN002在2株菌中没有扩增,位点CVN003在36株菌中没有扩增,分别以np(no product)表示,排除PCR扩增问题,认为这两个位点在本研究部分菌株中不存在或消失。由此可见CVN001在本实验中的13株STEC O157 ∶ H7菌株中没有多态性,CVN007 和CVN0015在57株非O157 STEC菌株中没有多态性,其余位 点在70株菌中均呈现较好多态性,尤其是CVN014在STEC O157 ∶ H7和非O157 STEC中均呈现最多的重复序列数目,应成为后续进一步完善MLVA方法时的候选位点。
2.2 MLVA分型分析
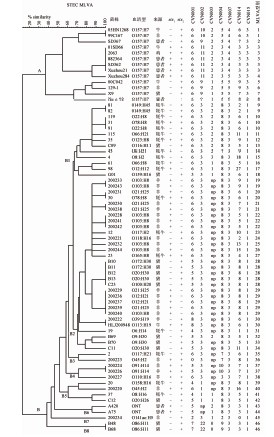
根据7个VNTR位点重复序列拷贝数,70株STEC菌株共分为46个MLVA型别,分别命名为MLVA1~MLVA46。进一步以BioNumerics软件对VNTR位点的拷贝数进行聚类分析,结果显示70株STEC菌株可分为明显的两个群(A、B),13株STEC O157 ∶ H7菌株均位于A群,57株非O157 STEC菌株均位于B群。B群菌株可进一步分为8个亚群,分别为B1~B8,其中B1占22.8%(13/57),B2占52.6%(30/57),B3~B8占24.6%(14/57),见图1。上述结果表明70株STEC代表性菌株具有较好的VNTR多态性,7个位点的VNTR方法,可以很好地区分O157与非O157 STEC菌株。
2.3 不同MLVA型别菌株的血清型情况
本研究中70株STEC菌株涵盖28种O ∶ H血清型,O157血清型与非O157血清型菌株分别为两个独立的克隆群。在27种非O157血清型菌株中,共有38种MLVA型别,某些相同血清型如O149 ∶ H45的菌株具有相同的MLVA型别;相同血清型如O103 ∶ H8、O21 ∶ H25,其中有的菌株为相同的MLVA型别,有的则属于不同的MLVA型别;有些不同血清型如O20 ∶ H30、O100 ∶ H20、O172 ∶ H30的菌株属于相同的MLVA型别。提示MLVA在区分非O157 STEC菌株的血清型方面,具有一定的区分能力,但也存在一定的局限性。
2.4 不同MLVA型别菌株携带志贺毒素基因情况
70株STEC分离株中,stx1基因阳性的20株,stx2基因阳性的34株,stx1+stx2基因均阳性的16株。志贺毒素基因类型与MLVA型别存在一定的相关性。除MLVA型别10、20、30存在不同志贺毒素类型的菌株外,其余所有相同MLVA型别的菌株志贺毒素类型相同。
2.5 不同宿主来源STEC菌株的MLVA分型情况
本研究中STEC菌株分离自腹泻患者和5种不同动物宿主,13株O157 ∶ H7菌株中人源菌株可与多种动物源菌株分为相同型别。57株非O157 STEC菌株中,2株人源菌株单独属于B6亚群;11株(11/18,61.1%)牦牛菌株属于B1亚群,其余7株属于B2、B3、B5亚群;17株(17/23,73.9%)株羊源菌株属于B2亚群,其余6株属于B3、B4、B5、B7亚群;13株(8/13,61.5%)株猪源菌株属于B2亚群,其余5株属于B1、B5、B8亚群。B2优势群中羊源菌株占56.7%(17/30),其次是猪源菌株占26.7%(8/30)。不同宿主来源菌株与MLVA型别存在明显的关联,除MLVA型别20、30存在不同宿主来源的菌株外,其余所有相同MLVA型别的菌株均具有相同的宿主来源,提示MLVA方法具有一定的菌株溯源能力。
图1 产志贺毒素大肠埃希菌分离株MLVA聚类图
Figure 1 Dendrogram of MLVA results for Shiga toxin-producing Escherichia coli isolates
3 讨论
近年来国际上报道非O157 STEC 感染率不断上升,许多国家甚至出现了非O157 STEC暴发 ,特别是2011年德国暴发大规模STEC O104 ∶ H4感染[13]。1999年我国苏皖地区暴发了O157 ∶ H7感染[14],随后我国在这些地区开展了O157 ∶ H7的监测,而非O157 STEC在我国的感染情况目前尚未开展系统工作,但我们仍应对非O157 STEC感染予以重视。
美国CDC建立的以PFGE技术为核心的实验室分子分型监测网络——PulseNet,已经成功处置和预警了多起细菌性传染病暴发疫情。目前有的病原菌如O157 ∶ H7已经发展了基于MLVA的第二代分子分型技术,有的病原菌MLVA技术仍在建立中。MLVA分型技术是依据不同菌株间细菌基因组中存在着VNTR位点拷贝数差异进行分子分型,具有分型能力强、简便、快速、稳定、数据交流便利等优点,可以识别暴发菌株,在细菌性传染病溯源及监测预警中可发挥重要作用。
本研究利用文献报道的针对不同致泻性大肠埃希菌和志贺菌的7个VNTR位点,对我国不同地区、不同宿主来源、不同血清型和毒力基因特点的代表性菌株进行了分析,该方案能很好的区分O157与非O157 STEC菌株。在区分非O157 STEC菌株方面,该方案能较好的区分不同血清型、不同志贺毒素类型以及不同动物来源的菌株。但仍有少部分血清型、志贺毒素类型和宿主来源不同的菌株未能被区分。分析这7个VNTR位点,少数位点对分析菌株的多态性有限,2个位点在部分菌株中无扩增产物。因此在应用该方案对暴发菌株进行溯源分析时,在条件允许的情况下,可进一步结合其他分型方案如PFGE,以做出更为准确的判断。同时,在目前GenBank等公共数据库中有越来越多STEC菌株测序基因组信息的情况下,可发掘新的更有区分能力的VNTR位点,进一步提高MLVA的分辨率,从而提高其对菌株甄别、暴发溯源等的应用价值。
参考文献
[1] Paton JC, Paton AW. Pathogenesis and diagnosis of Shiga toxin producing Escherichia coli infections[J]. Clin Microbiol Rev, 1998,11(3):450-479.
[2] Schaffzin JK,Coronado F,Dumas NBSchaffzin, et al. Public health approach to detection of non O157 Shiga toxin producing Escherichia coli: summary of two outbreaks and laboratory procedures[J]. Epidemiol Infect, 2012,140(2):283-289.
[3] Wahl E, Vold L, Lindstedt BA, et al. Investigation of an Escherichia coli O145 outbreak in a child day care centre extensive sampling and characterization of eae and stx1 positive E. coli yields epidemiological and socioeconomic insight[J]. BMC Infect Dis, 2011,11:238.
[4] Käppeli U, Hächler H, Giezendanner N, et al. Human infections with non O157 Shiga toxin producing Escherichia coli, Switzerland,2000-2009[J]. Emerg Infect Dis, 2011,17(2):180-185.
[5] Gould LH, Bopp C, Strockbine N, et al. Recommendations for diagnosis of Shiga toxin producing Escherichia coli infections by clinical laboratories[J]. MMWR Recomm Rep, 2009,58(RR-12):1-14.
[6] Karmali MA, Mascarenhas M, Shen S, et al. Association of genomic O island 122 of Escherichia coli EDL 933 with verocytotoxin producing Escherichia coli seropathotypes that are linked to epidemic and/or serious disease[J]. J Clin Microbiol, 2003,41(11):4930-4940.
[7] Oporto B, Esteban JI, Aduriz G, et al. Escherichia coli O157 H7 and non O157 Shiga toxin producing E. coli in healthy cattle, sheep and swine herds in Northern Spain[J]. Zoonoses Public Health, 2008,55(2):73-81.
[8] Wang T, Qi TN, Liu K, et al. Characterization of Escherichia coli O157:H7 isolated in China by multiple locus variable number of tandem repeat analysis[J]. Chinese Journal of Public Health, 2011,27(8):972-974. (in Chinese) 王涛,綦廷娜,刘凯,等. 大肠埃希菌O157:H7分离株MLVA分子分型[J]. 中国公共卫生,2011,27(8):972-974.
[9] Lindstedt BA, Brandal LT, Aas L, et al. Study of polymorphic variable number of tandem repeats loci in the ECOR collection and in a set of pathogenic Escherichia coli and Shigella isolates for use in a genotyping assay[J]. J Microbiol Methods, 2007,69(1):197-120.
[10] Wang T, Xiong YW, Bai XM, et al. Detection of enterohemorrhagic Escherichia coli O157:H7 by multiplex PCR[J]. Chinese Journal of Clinical Laboratory Science, 2010,28(4):264-266. (in Chinese)
王涛,熊衍文,白雪梅,等. 多重PCR检测肠出血性大肠埃希菌O157:H7[J]. 临床检验杂志,2010,28(4):264-266.
[11] Ethelberg S, Smith B, Torpdahl M, et al. Outbreak of non O157 Shiga toxin producing Escherichia coli infection from consumption of beef sausage[J]. Clin Infect Dis, 2009,48(8):78-81.
[12] Kappeli U, Hachler H, Giezendanner N, et al. Human infections with non O157 Shiga toxin producing Escherichia coli, Switzerland,2000-2009[J]. Emerg Infect Dis, 2011,17(2):180-185.
[13] Christina F, Dirk W, Jakob PC, et al. Epidemic Profile of Shiga Toxin Producing Escherichia coli O104:H4 Outbreak in Germany[J]. N Engl J Med, 2011,365:1771-1780.
[14] Xiong Y, Wang P, Lan R, et al. A novel Escherichia coli O157:H7 clone causing a major hemolytic uremic syndrome outbreak in China[J]. PLoS One, 2012,7(4):e36144.
[2] Schaffzin JK,Coronado F,Dumas NBSchaffzin, et al. Public health approach to detection of non O157 Shiga toxin producing Escherichia coli: summary of two outbreaks and laboratory procedures[J]. Epidemiol Infect, 2012,140(2):283-289.
[3] Wahl E, Vold L, Lindstedt BA, et al. Investigation of an Escherichia coli O145 outbreak in a child day care centre extensive sampling and characterization of eae and stx1 positive E. coli yields epidemiological and socioeconomic insight[J]. BMC Infect Dis, 2011,11:238.
[4] Käppeli U, Hächler H, Giezendanner N, et al. Human infections with non O157 Shiga toxin producing Escherichia coli, Switzerland,2000-2009[J]. Emerg Infect Dis, 2011,17(2):180-185.
[5] Gould LH, Bopp C, Strockbine N, et al. Recommendations for diagnosis of Shiga toxin producing Escherichia coli infections by clinical laboratories[J]. MMWR Recomm Rep, 2009,58(RR-12):1-14.
[6] Karmali MA, Mascarenhas M, Shen S, et al. Association of genomic O island 122 of Escherichia coli EDL 933 with verocytotoxin producing Escherichia coli seropathotypes that are linked to epidemic and/or serious disease[J]. J Clin Microbiol, 2003,41(11):4930-4940.
[7] Oporto B, Esteban JI, Aduriz G, et al. Escherichia coli O157 H7 and non O157 Shiga toxin producing E. coli in healthy cattle, sheep and swine herds in Northern Spain[J]. Zoonoses Public Health, 2008,55(2):73-81.
[8] Wang T, Qi TN, Liu K, et al. Characterization of Escherichia coli O157:H7 isolated in China by multiple locus variable number of tandem repeat analysis[J]. Chinese Journal of Public Health, 2011,27(8):972-974. (in Chinese) 王涛,綦廷娜,刘凯,等. 大肠埃希菌O157:H7分离株MLVA分子分型[J]. 中国公共卫生,2011,27(8):972-974.
[9] Lindstedt BA, Brandal LT, Aas L, et al. Study of polymorphic variable number of tandem repeats loci in the ECOR collection and in a set of pathogenic Escherichia coli and Shigella isolates for use in a genotyping assay[J]. J Microbiol Methods, 2007,69(1):197-120.
[10] Wang T, Xiong YW, Bai XM, et al. Detection of enterohemorrhagic Escherichia coli O157:H7 by multiplex PCR[J]. Chinese Journal of Clinical Laboratory Science, 2010,28(4):264-266. (in Chinese)
王涛,熊衍文,白雪梅,等. 多重PCR检测肠出血性大肠埃希菌O157:H7[J]. 临床检验杂志,2010,28(4):264-266.
[11] Ethelberg S, Smith B, Torpdahl M, et al. Outbreak of non O157 Shiga toxin producing Escherichia coli infection from consumption of beef sausage[J]. Clin Infect Dis, 2009,48(8):78-81.
[12] Kappeli U, Hachler H, Giezendanner N, et al. Human infections with non O157 Shiga toxin producing Escherichia coli, Switzerland,2000-2009[J]. Emerg Infect Dis, 2011,17(2):180-185.
[13] Christina F, Dirk W, Jakob PC, et al. Epidemic Profile of Shiga Toxin Producing Escherichia coli O104:H4 Outbreak in Germany[J]. N Engl J Med, 2011,365:1771-1780.
[14] Xiong Y, Wang P, Lan R, et al. A novel Escherichia coli O157:H7 clone causing a major hemolytic uremic syndrome outbreak in China[J]. PLoS One, 2012,7(4):e36144.
|
扩展功能
|
|
| 本文信息 | |
| PDF全文 | |
| HTML全文 | |
| 参考文献 | |
| 服务与反馈 | |
| 加入引用管理器 | |
| 引用本文 | |
| Email Alert | |
| 本文作者相关文章 | |
| 赵爱兰 | |
| 白向宁 | |
| 孟琼 | |
| 刘凯 | |
| 熊衍文 | |
| PubMed | |
| Article by ZHAO Ai-lan | |
| Article by BAI Xiang-ning | |
| Article by MENG Qiong | |
| Article by LIU Kai | |
| Article by XIONG Yan-wen | |