2. Collaborative Innovation Center for Diagnosis and Treatment of Infectious Diseases, Hangzhou 310003, Zhejiang, China
2. 感染性疾病诊治协同创新中心, 浙江 杭州 310003
-
1 材料与方法
- 1.1 菌株来源及数据准备
- 1.2 pstS1基因扩增及序列测定
- 1.3 pstS1基因中T/B细胞表位区的确定
- 1.4 数据分析
- 2.1 pstS1基因序列整理
- 2.2 pstS1基因序列整理
- 2.3 pstS1基因序列整理
- 2.4 pstS1基因中的单核苷酸多态性(SNP)
- 2.5 pstS1基因中T/B细胞表位区的变异
近年来,提高结核病(Tuberculosis, TB)诊断能力以及开发新的、有效的抗结核病疫苗成为结核病研究的热点,随着生物化学、免疫学以及分子生物学等学科的进步,已经发现有部分结核分枝杆菌(Mycobacterium tuberculosis)抗原可能在提高结核病诊断能力或研制新的疫苗方面具有重要作用[1]。Pst蛋白是一种细胞膜相关的复合体,属于ATP结合盒转运超家族(ATP binding cassette transporter superfamily)[2]。在结核分枝杆菌基因组中,共发现3个pst基因家族成员,分别为pstS1、pstS2和pstS3[3]。这3个基因具有较高的相似度,其中pstS1与pstS2和pstS3的相似度约为75%,pstS2与pstS3的相似度约为94%,都具有一个相似的脂蛋白信号序列。PstS蛋白位于结核分枝杆菌、牛分枝杆菌(Mycobacterium bovis)和BCG菌株的细胞表面[4],但也有研究表明,在BCG菌株中只能检测到少量的pstS1基因表达[5]。
2010年,Comas等[6]通过对来自世界不同地区的21株具有代表性的结核分枝杆菌复合群(Mycobacterium tuberculosis complex, MTBC)菌株的测序发现, 结核分枝杆菌基因中人T细胞表位具有高度的保守性,pstS1是其中少数几个具有碱基突变的基因之一。且PstS1蛋白抗原可能在刺激T细胞和B细胞活化、增殖方面具有重要作用。
本研究选取了180株国内的MTBC临床分离菌株以及11株不同来源的BCG菌株,扩增了其pstS1基因,测序后分析该基因及其中T/B细胞表位序列的多态性,并结合已经公布的其他MTBC菌株的数据,验证了相关多态性的特异性。
首先,从2346株国内结核分枝杆菌临床分离株中,挑选了180株作为本次研究的样本,这些菌株涵盖了所有已经在我国发现的Spoligotyping类型[7],这些菌株的省份来源及相关分型数据见表1。另外,还选取世界不同来源的11株BCG疫苗株,具体菌株信息见表 1。
表 1 180株临床菌株的来源、分型信息及BCG菌株名称Table 1 Sources and genotypes of 180 clinical isolates and names of BCG strains
| 省份 | 菌株数 (株) | Spoligotyping 分型结果 | 菌株数 (株) | BCG 菌株序号 | 菌株名称 |
| 安徽 | 12 | Beijing | 92 | 01 | BCG Birkhaug |
| 陕西 | 17 | T | 13 | 02 | BCG China |
| 北京 | 11 | U | 28 | 03 | BCG Danish |
| 福建 | 29 | MANU | 11 | 04 | BCG Frappier |
| 甘肃 | 12 | Haarlem | 5 | 05 | BCG Glaxo |
| 广西 | 29 | EAI | 2 | 06 | BCG Moreau |
| 四川 | 1 | LAM | 2 | 07 | BCG Phipps |
| 河南 | 12 | H37Rv family | 1 | 08 | BCG Prague |
| 湖南 | 7 | BCG | 2 | 09 | BCG Swedens |
| 西藏 | 11 | S | 1 | 10 | BCG Tice |
| 新疆 | 13 | CAS | 4 | 11 | BCG Russia |
| 吉林 | 14 | New | 19 | - | - |
| 浙江 | 12 | - | - | - | - |
| 合计 | 180 | - | 180 | - | - |
同时,从美国国立生物技术信息中心(National Center of Biotechnology Information, NCBI)网站下载了1株牛分枝杆菌(M. bovis AF2122/97,NC_002945)和3株BCG菌株(BCG Pasteur,NC_008769;BCG Tokyo,NC_012207以及BCG Mexico, NC_016804)中的pstS1的等位基因序列, 以及作为标准参照序列的结核分枝杆菌H37Rv株的pstS1基因序列。
1.2 pstS1基因扩增及序列测定本实验中扩增pstS1基因使用的PCR引物,为依据结核分枝杆菌H37Rv株的基因序列使用DNAstar软件设计,正、反向引物从5′到3′方向分别为ACC ACC GAG AAG ATC GAA GA和AAG AAA TGC AAC CCG TTG AG。PCR扩增的总反应体系为20 μl,其中包括2×PCR缓冲液10 μl,上、下游引物各100 nmol/L,4种dNTP各200 μmol/L以及0.5 U的DNA聚合酶。PCR反应条件为94 ℃预变性5 min,然后94 ℃变性45 s,62 ℃复性45 s,72 ℃延伸1 min,重复35个循环,最后72 ℃延伸10 min终止反应[8]。扩增产物送测序公司进行双向测序和拼接。
1.3 pstS1基因中T/B细胞表位区的确定首先检索免疫表位数据库(Immune Epitope Database, IEDB)[9],获得结核分枝杆菌中存在的人T/B细胞表位序列,再与H37Rv株的pstS1基因序列进行比对,最终获得在该基因中存在的人T/B细胞表位信息。
1.4 数据分析首先,对PCR产物测序和网络下载获得的pstS1基因相关序列,进行核酸序列方向一致性调整,使用Clustalw软件[10]进行alignment操作;再依据H37Rv株pstS1基因序列,截取所有测序菌株与之对应的基因全长序列;然后,对获得的基因全长数据进行分析,去除有碱基插入或缺失的菌株数据后,分别截取其中的T、B表位区(重合表位只截取一次)和非表位区;最后,用Mega 5软件[11]分别计算这些序列区中的同义突变率(dS)和非同义突变率(dN)及其比值。
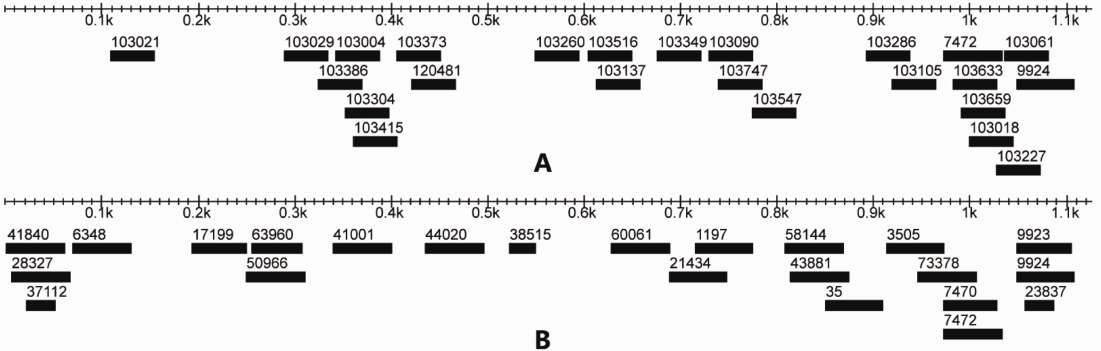
经检索IEDB及与H37Rv株pstS1基因序列进行比对后,本次研究共在H37Rv株pstS1基因中发现了24个B细胞表位和23个T细胞表位,见图1和表2。
注:A:B细胞表位分布; B:T细胞表位分布。图中标注的数字为表位的IEDB编号。图 1 结核分枝杆菌H37Rv株pstS1基因中T/B细胞表位位置分布
Figure 1 Distribution of T and B epitopes in pstS1 gene of H37Rv
表 2 H37Rv株pstS1基因序列中T/B细胞表位的分布信息
Table 2 T and B epitopes founded in H37Rv pstS1 gene
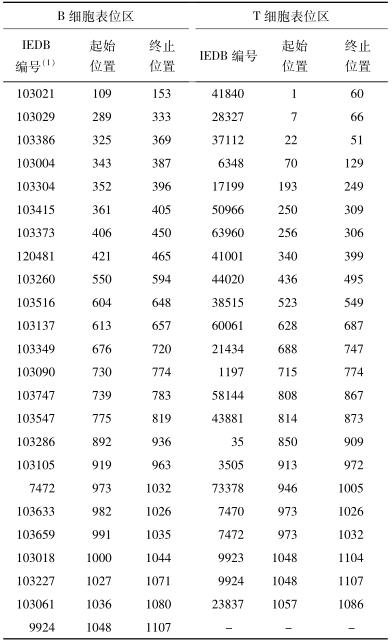 |
| 注:(1)编号为IEDB数据库中的表位编号。 |
经整理后,共获得196个菌株的pstS1基因序列,其中有178株结核分枝杆菌临床分离株、16株BCG菌株(包括11株世界不同国家来源的BCG菌株,2株临床使用株,以及前述3株NCBI已经公布数据的菌株)、1株牛分枝杆菌以及标准参照株H37Rv株。
2.3 pstS1基因中的碱基插入与缺失突变在所有196个菌株的pstS1基因序列中,共发现2个单碱基插入突变,未发现缺失突变。其中,FJ05132株在第405个碱基位置插入一个腺嘌呤脱氧核糖核苷酸(A),使第191位密码子成为终止密码子; JL06035株在第626个碱基位置插入一个胞嘧啶脱氧核糖核苷酸(C),使得第228位密码子成为终止密码子,均造成该基因所编码肽链的提前终止(图2)。
注:(1)6株结核分枝杆菌临床分离、所有的牛分枝杆菌以及BCG菌株在氨基酸水平上均存在变异,而本次研究中的其他菌株与标准参照株H37Rv一致,未在图中进行标注; (2)T/B细胞表位区在H37Rv株的pstS1基因序列上进行了框注。图 2 pstS1基因中的T/B细胞表位区及变异分布
Figure 2 Mutations and T/B epitope regions in pstS1 gene
2.4 pstS1基因中的单核苷酸多态性(SNP)
经序列比对分析发现,本研究所有的16株BCG菌株及1株牛分枝杆菌菌株中,均存在特异性的GTT352GCT突变,使得该密码子编码的氨基酸由缬氨酸(Val)变为丙氨酸(Ala);在2株东非-印度型(East AfricanIndian,EAI)的菌株(FJ05406和FJ06051)中,均存在特异性的ACC341ATC突变,致使该密码子编码的氨基酸由苏氨酸(Thr)变为异亮氨酸(Ile); JL06018株存在CCG50TCG突变,而ShanX05296株存在TTG364GTG突变,均造成相应密码子编码氨基酸的改变(表3和图2)。
表 3 MTBC菌株pstS1基因中的变异情况Table 3 T and B epitopes founded in H37Rv pstS1 gene
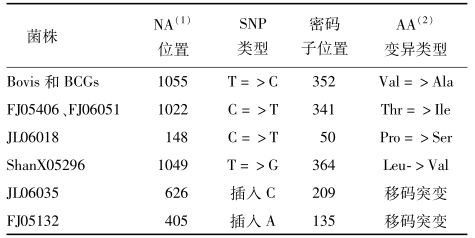 |
| 注:(1)NA代表核酸;(2)AA代表氨基酸。 |
2株(FJ05132和JL06035)存在单碱基插入突变的菌株, 由于出现移码突变(FrameShift mutation),对插入突变点后的细胞表位区造成巨大影响,其中:FJ05132株的突变影响了7917%(19/24)的B细胞表位和6522%(15/23)T细胞表位,而JL06035株的突变影响了6250%(15/24)的B细胞表位和5652%(13/23)T细胞表位;而前述4种SNP变异,则不同程度的影响了T/B细胞表位区。整个pstS1基因的 dN 值为0000 14,其中T细胞表位区与非T细胞表位区的 dN 值分别为0000 17和0000 05,B细胞表位区与非B细胞表位区的 dN 值分别为0000 23和0,由于未发现同义突变位点,故所有序列区的 dS 值均为零(表4、5和图2)。
表 4 MTBC菌株pstS1基因中的变异对T/B表位区的影响Table 4 Effects of mutations in pstS1 gene on T/B epitope region in MTBC strains
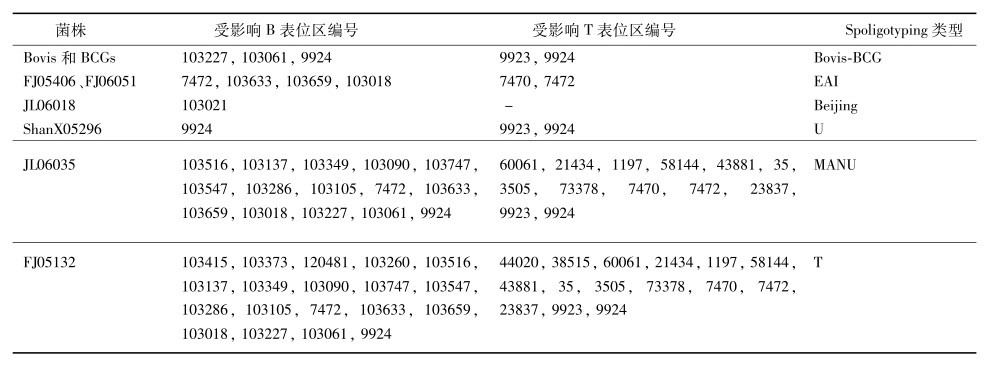 |
| 注:(1)表位编号为IEDB数据库中的编号。 |
表 5 测序菌株中pstS1基因不同序列区中的变异情况
Table 5 Mutations in different regions of pstS1 gene
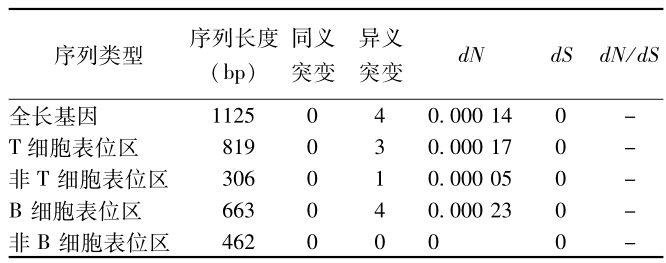 |
| 注:(1)2株含有单碱基插入变异的数据未纳入分析。 |
在本研究中,根据地区和基因型分布等条件,随机从国内不同地区来源、不同Spoligotyping类型的2346株临床分离MTBC菌株中,选取了180株,采用PCR和DNA测序方法,对其pstS1基因及其人T、B细胞抗原表位进行了分析。菌株代表性较好,得出的结果有较高可信度。
结核分枝杆菌的PstS1蛋白是其无机磷酸盐吸收系统中的磷离子结合亚单位,属于ABC转运超家族,位于细胞膜上,具有很强的免疫原性和免疫刺激性[12,13],同时,PstS1是糖化脂蛋白,可以存在于结核分枝杆菌菌体内,也可以分泌到培养液中。目前,通过检测结核分枝杆菌的PstS1蛋白,已经可以达到很高的结核病诊断特异性(>98%)[14],但其检测灵敏度却变化较大(45%~80%之间)[14,15,16]。本研究发现存在于pstS1基因中的变异,特别是移码突变的存在,均造成该基因编码肽链的改变,进而可能影响该基因编码的蛋白功能,造成检测灵敏度发生变化。此外,由于我们已经在研究中发现此基因具有相对较高的变异程度,将来在研制含有此抗原的新疫苗时,需要考虑到菌株间变异对其免疫效果的影响。
通过对人致病的病毒、细菌及寄生虫的研究已经发现,编码其抗原性物质的基因具有高度的变异性,这是一种逃避人体免疫系统的多向性选择[17,18,19]。虽然,之前的报告认为结核分枝杆菌的T细胞表位具有较高的保守性,并据此推断结核分枝杆菌缺少抗原变异及免疫逃逸[6]。但是在对pstS1基因的研究中发现,无论其T细胞表位(15/23, 6522%),还是B细胞表位(19/24,7917%),均表现出一个较高的变异水平,而且,T细胞表位区与B细胞表位区均高于对应的非表位区以及整个基因编码区的 dN 值(表5)。因此,pstS1基因可能在结核分枝杆菌免疫逃逸中发挥重要作用,但将来仍需要更多实验进行验证。
在 16株不同来源的BCG菌株及1株牛分枝杆菌(NC_002945)菌株的pstS1基因中,均存在一个共同的特异性SNP位点(T1055C),造成牛分枝杆菌和BCG菌株的PstS1肽链中第352氨基酸残基由Val变成Ala,且这个突变位于功能域Ⅰ(domain Ⅰ)的羧基端,并靠近磷离子(Pi)结合位点(PDB_ID: 4LAT)。尽管突变后编码的氨基酸与原氨基酸性质相近,对此蛋白功能的影响相对较小,但仍可能作为一个鉴别牛分枝杆菌与结核分枝杆菌菌株的生物标志。
另外,本研究中的FJ06051和FJ05406株为EAI基因型的菌株,同样具有一个特异性的SNP位点(C1022T, Thr341Ile),但是,由于在此次研究中取得EAI基因型菌株较少,仍然需要进一步的工作进行验证。
综上所述,MTBC菌株pstS1基因中的变异,可能会对该基因所编码蛋白的功能造成影响,并可能在人体内的免疫逃逸过程中发挥作用;同时,该基因中一个特异性的SNP位点(T1055C)可能被用来鉴别牛分枝杆菌与结核分枝杆菌菌株。
[2] Braibant M, Gilot P. The ATP binding cassette (ABC)transport systems of Mycobacterium tuberculosis[J]. FEMS Microbiol Rev, 2000,24(4):449-467.
[3] Cole S, Brosch R, Parkhill J, et al. Deciphering the biology of Mycobacterium tuberculosis from the complete genome sequence[J]. Nature, 1998,393(6685):537-544.
[4] Lefevre P, Braibant M, de Wit L, et al. Three different putative phosphate transport receptors are encoded by the Mycobacterium tuberculosis genome and are present at the surface of Mycobacterium bovis BCG[J]. J Bacteriol, 1997,179(9):2900-2906.
[5] Andersen AB, Hansen EB. Structure and mapping of antigenic domains of protein antigen b, a 38 000-molecular-weight protein of Mycobacterium tuberculosis[J]. Infect Immun, 1989,57(8):2481-2488.
[6] Comas I, Chakravartti J, Small PM, et al. Human T cell epitopes of Mycobacterium tuberculosis are evolutionarily hyperconserved[J]. Nat Genet, 2010,42(6):498-503.
[7] Dong HY, Liu ZG, Lyu B, et al. Spoligotypes of Mycobacterium tuberculosis from different Provinces of China[J]. J Clin Microbiol, 2010,48(11):4102-4106.
[8] Liu HC, Jiang Y, Dou XF, et al.pstS1 polymorphisms of Mycobacterium tuberculosis strains may reflect ongoing immune evasion[J]. Tuberculosis, 2013,93(5):475-481.
[9] Salimi N, Fleri W, Peters B, et al. The immune epitope database: a historical retrospective of the first decade[J]. Immunology, 2012,137(2):117-123.
[10] Thompson JD, Higgins DG, Gibson TJ. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice[J]. Nucleic Acids Res, 1994,22(22):4673-4680.
[11] Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Mol Biol Evolut, 2011,28(10):2731-2739.
[12] Vordermeier H, Coombes A, Jenkins P, et al. Synthetic delivery system for tuberculosis vaccines: immunological evaluation of the M. tuberculosis 38 kDa protein entrapped in biodegradable PLG microparticles[J]. Vaccine, 1995,13(16):1576-1582.
[13] Shi JF, Song GZ, Pan JC, et al. Study on expression, purification and immunoreactivity of Mycobacterium tuberculosis recombinant PstS1[J]. Chinese Preventive Medicine,2011,12(4):307-310. (in Chinese) 石君帆,宋广忠,泮结超,等. 结核杆菌 H37Rv 株重组 PstS1 蛋白的表达纯化及其免疫反应性分析[J]. 中国预防医学杂志,2011,12(4):307-310.
[14] Bothamley GH, Rudd R, Festenstein F, et al. Clinical value of the measurement of Mycobacterium tuberculosis specific antibody in pulmonary tuberculosis[J]. Thorax, 1992,47(4):270-275.
[15] Daniel TM, Debanne SM. The serodiagnosis of tuberculosis and other mycobacterial diseases by enzyme-linked immunosorbent assay[J]. Am Rev Respir Dis, 1987,135(5):1137-1151.
[16] Espitia C, Cervera I, Gonzalez R, et al. A 38-kD Mycobacterium tuberculosis antigen associated with infection. Its isolation and serologic evaluation[J]. Clin Exp Immunol,1989,77(3):373-377.
[17] Kawashima Y, Pfafferott K, Frater J, et al. Adaptation of HIV-1 to human leukocyte antigen class Ⅰ[J]. Nature, 2009,458(7238):641-645.
[18] Jeffares DC, Pain A, Berry A, et al. Genome variation and evolution of the malaria parasite Plasmodium falciparum[J]. Nat Genet, 2007,39(1):120-125.
[19] Urwin R, Russell JE, Thompson EA, et al. Distribution of surface protein variants among hyperinvasive meningococci: implications for vaccine design[J]. Infect Immun, 2004,72(10):5955-5962.
|
扩展功能
|
|
| 本文信息 | |
| PDF全文 | |
| HTML全文 | |
| 参考文献 | |
| 服务与反馈 | |
| 加入引用管理器 | |
| 引用本文 | |
| Email Alert | |
| 本文作者相关文章 | |
| 刘海灿 | |
| 蒋毅 | |
| 李马超 | |
| 赵秀芹 | |
| 万康林 | |
| PubMed | |
| Article by LIU Hai-can | |
| Article by JIANG Yi | |
| Article by LI Ma-chao | |
| Article by ZHAO Xiu-qin | |
| Article by WAN Kang-lin | |