-
1 材料与方法
2 结果
- 2.1 大肠埃希菌产ESBLs菌株与非产ESBLs菌株毒力因子分布
- 2.2 ESBLs基因PCR扩增结果及相关统计分析
- 2.2.1 对92株产ESBLs大肠埃希菌进行超广谱
- 2.2.2 不同基因型产ESBLs大肠埃希菌毒力因子分布
大肠埃希菌是临床上常见的致病菌,随着三代头孢菌素等广谱β内酰胺类抗生素在临床上的广泛应用,产超广谱β内酰胺酶(Extended Spectrum Beta-Lactamase,ESBLs)大肠埃希菌感染日益增多。ESBLs由质粒介导,易在同种属甚至不同种属细菌间传递造成暴发流行,由于各个国家和地区使用的抗菌药物存在差异,流行的ESBLs基因型各不相同。ESBLs的传播性以及它们的多重耐药性给临床抗感染治疗造成了极大困难,及时准确检出ESBLs对防止ESBLs流行、控制院内感染和指导临床治疗有着非常重要的意义。
本研究对浙江省温州医科大学附属第一医院临床分离的产超广谱β-内酰胺酶大肠埃希菌的产生率、超广谱β-内酰胺酶的基因型及所含的毒力因子进行检测,分析ESBLs与毒力因子的相关性,为提高抗菌药物治疗的有效率提供依据。
188株大肠埃希菌为温州医科大学附属第一医院临床送检标本中分离得到,其中64株分离自尿液标本,62株分离自脓液标本,另62株分离自血液标本,全部菌株应用法国生物梅里埃公司产品VITEK 2 Compact型全自动微生物分析仪作菌种鉴定,均为非重复菌株。质控菌株大肠埃希菌(ATCC 25922)和肺炎克雷伯菌(ATCC 700603)分别为ESBLs检测的阴性和阳性质控菌株。
VITEK 2 Compact 全自动微生物分析仪及其配套的细菌鉴定和药敏检测卡(法国生物梅里埃公司); LB液体培养基(OXOID公司);PCR试剂(上海生工生物工程技术有限公司);50×TAE电泳试剂为实验室自配;琼脂糖粉为西班牙进口分装。隔水式恒温培养箱 (上海一恒科技有限公司);THZ-C恒温振荡摇床 (江苏太仓实验设备厂);BECKMAN低温离心机(Beckman公司);MyCycler Thermal Cycler 170-9703 PCR 扩增仪(BIO-RAD 公司);紫外凝胶分析系统(SynGene公司)。
按CLSI推荐的标准纸片扩散确证法测定ESBLs。同时用大肠埃希菌(ATCC 25922)和肺炎克雷伯菌(ATCC 700603)分别作阴性和阳性的质控对照。
采用聚合酶链反应(PCR)扩增。
(1)引物设计:参照文献[1-4],由上海生工生物工程技术有限公司合成。序列及预期PCR扩增产物大小见表1、2。
(2)模板制备:将血平板上过夜培养的单个菌落挑于含1 ml LB的1.5 ml EP管中,过夜培养,然后于沸水中煮 10 min,12 000 r/min离心 3 min,上清液作为模板。
(3)PCR反应体系及反应条件:反应体系(25 μl):上、下游引物各 0.2 μmol/L,10×buffer缓冲液 2.5 μl,dNTP 100 μmol/L, Mg2+ 1.5 mmol/L,Taq酶1.0 U,模板2 μl,加灭菌ddH2O至25 μl。
(4)基因PCR扩增反应条件:94 ℃预变性5 min;94 ℃ 30 s;52 ℃ 30 s;72 ℃延伸60 s;共30个循环,72 ℃再延伸10 min。扩增产物在含0.5 μg/ml EB的1.0%琼脂糖凝胶于1×TAE 中电泳, 紫外透射反射仪观察结果,由紫外凝胶分析系统进行分析。以出现预期产物大小的条带判为阳性。
表1 毒力基因PCR扩增的引物Table 1 Primers for PCR amplification of virulence genes
| 基因类型 | 序列 (5′~3′) | 预期产物大小(bp) | 退火温度(℃) |
| papC | GTG GCA GTA TGA GTA ATG ACC GTT A | 203 | 54 |
| ATA TCC TTT CTG CAG GGA TGC AAT A | |||
| sfa/foc | CTC CGG AGA ACT GGG TGC ATC TTA C | 408 | 55 |
| CGG AGG AGT AAT TAC AAA CCT GGC A | |||
| afa/dra | GGC AGA GGG CCG GCA ACA GGC | 592 | 60 |
| CCC GTA ACG CGC CAG CAT CTC | |||
| iutA | GGC TGG ACA TCA TGG GAA CTG G | 302 | 58 |
| CGT CGG GAA CGG GTA GAA TCG | |||
| aer | TAC CGG ATT GTC ATA TGC AGA CCG | 602 | 55 |
| AAT ATC TTC CTC CAG TCC GGA GAA G | |||
| fimH | TCG AGA ACG GAT AAG CCG TGG | 506 | 58 |
| GCA GTC ACC TGC CCT CCG GTA | |||
| hly | AAC AAG GAT AAG CAC TGT TCT GGC T | 1177 | 54 |
| ACC ATA TAA GCG GTC ATT CCC GTC A | |||
| cnf1 | AAG ATG GAG TTT CCT ATG CAG GAG | 498 | 52 |
| CAT TCA GAG TCC TGC CCT CAT TAT T | |||
| kpsMTⅡ | GCG CAT TTG CTG ATA CTG TTG | 270 | 53 |
| CAT CCA GAC GAT AAG CAT GAG CA | |||
| fyuA | TGA TTA ACC CCG CGA CGG GAA | 785 | 58 |
| CGC AGT AGG CAC GAT GTT GTA |
表2 ESBLs基因PCR扩增引物
Table 2 Primers for PCR amplification of ESBLs genes
| 引物名称 | 引物序列 | 扩增产物大小(bp) | 退火温度(℃) |
| TEM | P1: ATA AAA TTC TTG AAG ACG AAA P2: GAC AGT TAC CAA TGC TTA ATC A | 1080 | 52 |
| SHV | P1: GGT TAT GCG TTA TAT TCG CC P2: TTA GCG TTG CCA GTG CTC | 865 | 57 |
| VEB | P1: CGA CTT CCA TTT CCC GAT GC P2: GGA CTC TGC AAC AAA TAC GC | 643 | 50 |
| PER | P1: ATG AAT GTC ATT ATA AAA GC P2: AAT TTG GGC TTA GGG CAG AA | 925 | 50 |
| GES | P1:ATG CGC TTC ATT CAC GCA C P2:CTA TTT GTC CGT GCT CAG G | 864 | 54 |
| CTX-M-1 | P1: AAA AAT CAC TGC GTC AGT TCA C P2: ACA AAC CGT TGG TGA CGA TT | 867 | 56 |
| CTX-M-2 | P1: TTA ATG ATG ACT CAG AGC ATT C P2: GAT ACC TCG CTC CAT TTA TTG | 902 | 55 |
| CTX-M-8 | P1: ACA TCG CGT TAA GCG GAT P2: AAC CCA CGA TGT GGG TAG C | 691 | 55 |
| CTX-M-9 | P1: TAT TGG GAG TTT GAG ATG GT P2: TCC TTC AAC TCA GCA AAA GT | 933 | 53 |
| OXA-10 | P1: GTC TTT CGA GTA CGG CAT TA P2: ATT TTC TTA GCG GCA ACT TAC | 720 | 51 |
采用四格表资料的χ2检验分析,应用SPSS 13.0软件进行统计分析,P<0.05为差异有统计学意义。
产ESBLs大肠埃希菌株中,毒力因子iutA、fyuA的检出率较高,均>50%;毒力因子fimH、aer的检出率为20%~40%,而毒力因子papC、sfa/foc、afa/dra、kpsMTII的检出率均较低<20%;经χ2检验产ESBLs菌株与非产ESBLs菌株的毒力因子分布率差异均无显著性(P>0.5),见表3。
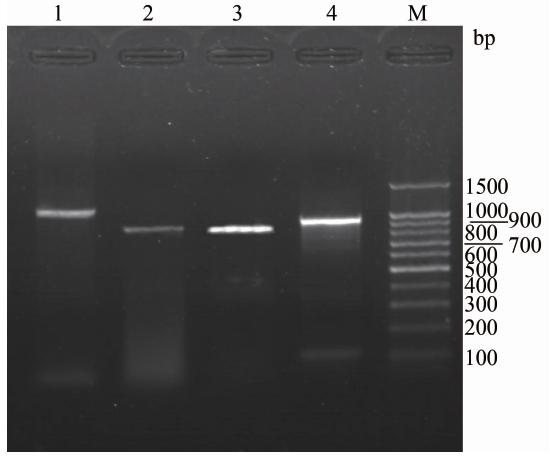
β-内酰胺酶基因TEM、SHV、VEB、PER、GES、CTX-M-1、CTX-M-2、CTX-M-8、CTX-M-9、OXA-10检测,其中共检测到ESBLs基因阳性85株,检出率为92.4%。85株ESBLs基因阳性菌株中有64株携带CTX-M-9型基因,32株携带CTX-M-1型基因,23株携带TEM型基因,1株携带SHV型基因,其基因分布见表4,其余基因均未检出。基因电泳结果见图1。
表3 大肠埃希菌产ESBLs菌株与非产ESBLs菌株的毒力因子分布Table 3 Distributions of virulence factors of ESBLs producing E. coli strains and non ESBLs producing E. coli strains
| 毒力 因子 |
|
|
| papC | 12 | 13.0 | 12 | 12.5 |
| sfa/foc | 6 | 6.5 | 6 | 6.3 |
| afa/dra | 3 | 3.2 | 6 | 6.3 |
| iutA | 54 | 58.6 | 57 | 59.4 |
| aer | 36 | 39.1 | 38 | 39.6 |
| fimH | 19 | 20.6 | 21 | 21.9 |
| kpsMTII | 3 | 3.2 | 8 | 8.3 |
| fyuA | 57 | 61.9 | 51 | 53.1 |
表4 85株产ESBLs大肠埃希菌的ESBLs基因型分布
Table 4 Distribution of ESBLs genotyes of 85 strains of ESBLs producing E. coli
| 基因型 | 菌株数 | 构成比(%) |
| TEM | 3 | 3.5 |
| SHV | 1 | 1.2 |
| CTX-M-1 | 15 | 17.6 |
| CTX-M-9 | 35 | 41.2 |
| TEM+CTX-M1 | 2 | 2.4 |
| TEM+CTX-M9 | 14 | 16.5 |
| CTX-M-9+CTX-M-1 | 11 | 12.9 |
| TEM+CTX-M-9+CTX-M-1 | 4 | 4.7 |
| 合计 | 85 | 100.0 |
注1~4分别为TEM:1080 bp、 SHV:865 bp、CTX-M-1:867 bp、CTX-M-9:933 bp,M为Marker。
图1 不同基因型PCR扩增产物琼脂糖电泳结果
Figure 1 Agarose electrophoresis results of PCR amplification products of different genotypes
对9种毒力因子在85株ESBLs基因阳性菌株中的检出率进行分析,见表5。毒力因子papc在CTX-M-9基因阳性菌株中的检出率为21.9%,高于其在TEM基因阳性菌株及CTX-M-1基因阳性菌株中的检出率;而毒力因子aer在CTX-M-1基因阳性菌株中的检出率则要高于其在CTX-M-9基因阳性菌株及TEM基因阳性菌株中的检出率;毒力因子fyuA在CTX-M-9基因阳性菌株及CTX-M-1基因阳性菌株中的检出率则要高于其在TEM基因阳性菌株中的检出率。经 χ2检验差异有统计学意义(P<0.05)。
表5 不同亚型ESBLs大肠埃希菌毒力因子分布率
Table 5 Distribution of virulence factors of E. coli with different subtypes of ESBLs
| 毒力 因子 | TEM | CTX-M-9 | CTX-M-1 | |||
| 标本数 | 构成比 (%) | 标本数 | 构成比 (%) | 标本数 | 构成比 (%) | |
| papC | 1 | 4.3 | 14 | 21.9 | 0 | 0.0 | ||
| sfa/foc | 0 | 0.0 | 0 | 0.0 | 0 | 0.0 | ||
| afa/dra | 1 | 4.3 | 0 | 0.0 | 0 | 0.0 | ||
| iutA | 10 | 43.4 | 35 | 54.7 | 12 | 37.5 | ||
| aer | 7 | 30.4 | 28 | 43.8 | 20 | 62.5 | ||
| fimH | 7 | 30.4 | 14 | 21.9 | 10 | 31.2 | ||
| kpsMTII | 2 | 8.7 | 3 | 4.7 | 0 | 0.0 | ||
| PAI | 0 | 0.0 | 0 | 0.0 | 0 | 0.0 | ||
| fyuA | 11 | 47.8 | 50 | 78.1 | 23 | 71.9 |
大肠埃希菌是导致各种医院内感染的重要病原菌,近年来,随着广谱抗菌药物尤其是第三代头孢菌素在临床上的广泛应用,使大肠埃希菌等革兰阴性菌对其耐药性迅速增加,其中重要原因是产生超广谱β-内酰胺酶(ESBLs)。ESBLs的出现给临床抗感染治疗带来众多问题:如患者住院时间延长,治疗经费增高,死亡率上升,院内感染概率升高等。产ESBLs大肠埃希菌对大多数广谱β-内酰胺类抗生素高度耐药,对喹诺酮类、氨基甙类、磺胺类药物多重耐药。自20世纪80年代ESBLs细菌引起的感染流行以来,该类细菌流行逐年上升,特别是肠杆菌科细菌,是目前临床治疗的难题之一。
本实验188株大肠埃希菌中检出产ESBLs菌株92株,占48.9%(92/188),高于黄印启等[5]报道的数据。说明不同时间、不同地区,由于抗菌药物的使用种类和使用量不同等原因,ESBLs的检出率存在差异。
按遗传学特性分类,可将ESBLs分为5类,TEM型、SHV型、CTX-M型、OXA型和其他。现已发现ESBLs基因型近200种,其中CTX-M型36种,它是由质粒介导产生的A类β内酰胺酶,于1989年首次德国和阿根廷自大肠埃希菌分离,具有ESBLs一般特性,其特点为对CTX的水解活性远高于头孢他啶,而命名为头孢噻肟酶。由于各地区抗生素的使用策略不同,各国家、地区所分离的占优势的ESBLs耐药基因型也不尽相同[6-8]。CTX-M型酶在南美、东亚和东欧地区流行。早期中国大陆地区以CTX-M-3和CTX-M-14为主,其他类型较少见[9-10],但近年来随着细菌耐药情况的变化,有报道浙江地区以CTX-M-15为主[11]。本实验结果显示:85株ESBLs基因阳性大肠埃希菌中CTX-M-9群比例最高(64株)占75.3%,TEM群(23株)占27.1%,CTX-M-1群(32株)占37.6%,SHV群(1株)占1.2%。未检测到基因型CTX-M-2型,CTX-M-8型,VEB型,GES(IBC)型及OXA-10存在。说明浙江省温州医科大学附属第一医院CTX-M-9酶流行较严重,与以往报道不相一致,这种ESBLs分离率的差异可能与不同医院、地区使用三代头孢菌素的种类数量及时间不同而造成对ESBLs的筛选及诱导不同有关,所以不同地区基因型的差异也可能与不同的质粒参与介导ESBLs基因转移有关。
近年来,大肠埃希菌毒力因子的研究已成为越来越受关注的话题 。Karisik等[14] 曾对产CTX-M型ESBLs及不产ESBLs的大肠埃希菌的毒力基因进行检测,发现毒力基因fimH、fyuA、iutA、kpsMTII、traT等在产CTX-M型ESBLs及不产CTX-M型ESBLs大肠埃希菌中有差异,在产CTX-M型ESBLs中检出率高。但国内尚未报道产ESBLs大肠埃希菌的毒力因子情况。本研究结果显示:产ESBLs大肠埃希菌株中, 毒力因子pap、sfa/foc、afa/dra、kpsMTII的检出率均较低,而毒力因子fimH、iutA、aer检出率较高;同时,非ESBLs大肠埃希菌株中,papC阳性株占12.5%,sfa/foc 阳性株占6.3%,kpsMTII阳性株占8.3%,afa/dra阳性株占6.3%,均较低,iutA阳性株占59.4%,aer阳性株占39.6%,fimH阳性株占21.9%, fyuA阳性株占53.1%,要高于上述毒力因子,但产ESBLs菌株与非产ESBLs菌株的毒力因子分布率差异均无统计学意义(P>0.5)。可见ESBLs的表型跟各个毒力因子的存在与否无关。表4显示ESBLs不同基因型毒力因子的分布情况,结果显示:毒力因子papc在CTX-M-9基因阳性菌株中的检出率为21.9%,高于其在TEM基因阳性菌株及CTX-M-1基因阳性菌株中的检出率;而毒力因子aer在CTX-M-1基因阳性菌株中的检出率则要高于其在CTX-M-9基因阳性菌株及TEM基因阳性菌株中的检出率;毒力因子fyuA在CTX-M-9基因阳性菌株及CTX-M-1基因阳性菌株中的检出率则要高于其在TEM基因阳性菌株中的检出率,差异有统计学意义。ESBLs不同基因型毒力因子分布存在差异性,毒力因子是否与产ESBLs的大肠埃希菌耐药有一定关系,有待进一步研究。
[2] Sehembri MA, Kjaergaard K, Sokurenko EV, et al.Molecular characterization of the Escherichia coli FimH adhesin[J]. J lnfect Dis,2001, 183(Suppl1):S28-31.
[3] Johnson JR, Stell AL.Extended virulence genotypes of Escherichia coli strains from patients with urosepsis in relation to phylogeny and host compromise[J]. J Infect Dis,2000, 181(1):261-272.
[4] Hou M, He JD, Chen JY, et al.Ralationship between the distribution of virulence factors of Escherichia coli leading to pyelonephritis and the resistance of Escherichia coli[J].Tianjin Medical,2008, 36(5):321-323.(in Chinese) 侯敏, 贺靖冬, 陈锦英, 等.致肾盂肾炎大肠杆菌毒力因子的分布及其与耐药性的关系[J]. 天津医药, 2008, 36(5):321-323.
[5] Huang YQ, Wu XY, Feng ZJ, et al.The effects of E.coli resistance to antibacterial therapy for urinary tract infection[J]. Hebei Medical Journal,2009, 31(10):1164-1166.(in Chinese) 黄印启, 武湘云, 冯忠军, 等. 大肠埃希菌耐药性对尿路感染抗菌治疗的影响[J].河北医药, 2009, 31(10):1164-1166.
[6] Jorgensen JH, McElmeel ML, Fulcher LC, et al.Detection of CTX-M-Type Extended-Spectrum Beta-Lactamase(ESBLs)by testing with Microscan overnight and ESBLs confirmation panels[J].J Clin Microbiol,2010, 48(1):120-123.
[7] Yan JJ, Tsai SH, Chuang CL, et al.OXA-type beta-lactamases among extended-spectrum cephalosporin-resistent Pserdomonas aeruginosa isolates in a univercity hospital in souther Taiwan[J].J Microbiol Immunol Infect,2006, 39:130-134.
[8] Rahman NMW, Lutfor AB, Jhora ST, et al.Detection of CTX-M gene in extended spectrum beta lactamase(ESBL) producing Escherichia coli and Klebsiella species of different hospitals[J].Bangladesh J Med Microbiol,2010, 4(2):28-31.
[9] Zhu DM, Xiong ZZ, Wang F, et al. Extended Spectrum Beta-Lactamase and bacterial drug resistance[J]. Chinese Journal of Infectious Diseases,2000, 18(3):151-154.(in Chinese) 朱德妹, 熊自忠, 汪复, 等. 超广谱β-内酰胺酶和细菌耐药性[J]. 中华传染杂志, 2000, 18(3):151-154.
[10] Yu YS, Zhou WL, Chen YA, et al. Epidenmiological and antlbiotic resistant study on extended-spectrumβ-lactamase producing Escherichia coli and Klebsiella pneumoniae in Zhejiang province[J]. Chin Med,2002, 115(10):1479-1482.
[11] Zhang R, Zhou HW, Cai JC, et al.Serotypes and extended-sprctrum-β-lactamase types of clinical isolates of Shigella spp from the Zhejiang province of China[J].Diagn Microbiol Infect Dis,2011, 69(1)98-104.
[12] Johnson JR.Virulence factors in Escherichia coli[J].J Chin Microbiol,2005, 43(12):6221-6222.
[13] Sharma S, Bhat GK, Shenoy S, et al.Virulence factors and drug resistance in Echerichia coli isolate from extraintestinal infections[J].Indian J Microbiol,2007, 25(4):369-373.
[14] Karisik E, Ellington MJ, Pike R, et al.Differentia expression of CTX-M-15-β-lactamase between two major Escherichia coli strains responsible for outbreaks in the UK[J].Clin Microbiol Infect Dis,2006, 16(4):1-4.
|
扩展功能
|
|
| 本文信息 | |
| PDF全文 | |
| HTML全文 | |
| 参考文献 | |
| 服务与反馈 | |
| 加入引用管理器 | |
| 引用本文 | |
| Email Alert | |
| 本文作者相关文章 | |
| 徐春泉 | |
| 吴庆 | |
| 张雪青 | |
| 陈栎江 | |
| 王赛芳 | |
| 虞丹丹 | |
| PubMed | |
| Article by XU Chun-quan | |
| Article by WU Qing | |
| Article by ZHANG Xue-qing | |
| Article by CHEN Li-jiang | |
| Article by WANG Sai-fang | |
| Article by YU Dan-dan | |