扩展功能
文章信息
- 徐征, 朱兵清, 高源, 徐丽, 邵祝军
- XU Zheng, ZHU Bing-qing, GAO Yuan, XU Li, SHAO Zhu-jun
- 中国不可分群脑膜炎奈瑟菌的分子分型分析
- Molecular typing of non sero-groupable Neisseria meningitidis in China
- 疾病监测, 2014, 29(9): 688-692
- Disease Surveillance, 2014, 29(9): 688-692
- 10.3784/j.issn.1003-9961.2014.09.005
-
文章历史
- 收稿日期:2014-7-7
脑膜炎奈瑟菌(Neisseria meningitidis,Nm)是引起流行性脑脊髓膜炎(流脑)的病原菌,也是细菌性脑膜炎的主要病原菌之一[1]。根据荚膜多糖的结构,脑膜炎奈瑟菌分为A、B、C、X、Y、Z、E、W、L、H、I和K共12个血清群。另有一些菌株不能归为以上血清群,称为不可分群菌株(non-serogroupable,NG)[2]。能引起侵袭性流脑病例的菌株多具有完整的荚膜,主要包括A、B、C、W、Y和X 6个血清群[3, 4]。
一直以来,NG菌株在健康人群中携带率较高,被认为没有致病性或仅在免疫低下的人群中引起感染[5]。但是,由NG菌株致病的情况也屡见报道[6, 7],2010年,我国也发现了首例由NG菌株感染引起的脑膜炎病例[8]。
本研究采用普通聚合酶链反应 (polymerase chain reaction,PCR)方法,对我国2005 2011年分离的104株NG脑膜炎奈瑟菌菌株进行了基因分群,并运用多位点序列分型(multilocus sequence typing,MLST)方法,对这些菌株进行了分子分型分析。 1 材料与方法 1.1 材料 1.1.1 菌株
104株NG脑膜炎奈瑟菌分离自我国17个省(直辖市)的健康带菌者,分离时间为2005 2011年;其中1株分离自北京2009年脑膜炎患者脑脊液。 1.1.2 主要仪器、试剂
PCR仪为Bio-Rad DNAEngine,凝胶成像仪为Gel Doc XR,均为Bio-Rad公司产品;Taq DNA聚合酶、dNTPs及100 bp DNA Marker购于TaKaRa生物工程公司。DNA提取采用Promega Wizard基因组DNA纯化试剂盒。PCR引物由上海生物工程有限公司合成。 1.2 方法 1.2.1 菌株鉴定和DNA提取
所有脑膜炎奈瑟菌经涂片革兰染色、API NH(法国生物梅里埃公司)生化鉴定和血清学玻片凝集检测(美国Remel公司分群血清),证实为NG脑膜炎奈瑟菌。收集哥伦比亚巧克力平板上新鲜培养的菌苔,用Promega试剂盒提取染色体DNA。 1.2.2 基因分群PCR
用于A、B、C、W、Y、E、X、Z、H、I、L、K基因群鉴定的PCR方法参照文献 ,荚膜基因缺失菌株(capsule null locus,cnl)的鉴定采用一对覆盖整个荚膜基因A区和C区的引物(位于荚膜基因C区上游的tex和A区下游的galE基因中)[12, 13]。所有引物序列见表 1。质控阳性菌株号分别为A(CMCC29019)、B(CMCC 29021)、C(CMCC 371101)、E(CMCC 29034)、W(CMCC 29037)、Y(CMCC 29028)、H(CMCC 29031)、I(CMCC 29044)、K(CMCC 29046)、Z(ATCC35562)、L(ATCC43828)和X(CMCC 29040)。
| 基因群 | 引物名称 | 序列(5′~3′) | 目的基因 | 扩增片段长度(bp) | 参考文献 |
| A | Orf2-F | cgc aat agg tgt ata tat tct tcc | Orf2 | 400 | [9] |
| Orf2-R | cgt aat agt ttc gta tgc ctt ctt | ||||
| B | siaD-F | gga tca ttt cag tgt ttt cca cca | siaD | 450 | [9] |
| siaD-R | gca tgc tgg agg aat aag cat taa | ||||
| W | W-F | cag aaa gtg agg gat ttc cat a | synF | 120 | [9] |
| W-R | cac aac cat ttt cat tat agt tac tgt | ||||
| Y | Y-F | ctc aaa gcg aag gct ttg gtt a | synG | 120 | [9] |
| Y-R | ctg aag cgt ttt cat tat aat tgc taa | ||||
| C | synE-F | tcc aag att ccc agt ggt ttg c | synE | 711 | [9] |
| synE-R | act tca tct gaa aca gga tag cg | ||||
| E | E-F | att acg ctg acg gca tgt gga | ctrA-29E | 667 | [10] |
| E/X/Z-R | ttg tcg cgg att tgc aac ta | ||||
| X | X-F | gtc ttt gta taa ggc cca ag | ctrA-X | 625 | [10] |
| E/X/Z-R | ttg tcg cgg att tgc aac ta | ||||
| Z | Z-F | tat gcg gtg ctg ttc gct atg | ctrA-Z | 667 | [10] |
| E/X/Z-R | ttg tcg cgg att tgc aac ta | ||||
| H | wnmA-F | tga tct acc caa ggc aca tac | wnmA | 1454 | |
| GalEGC1 | gga cgg cgg cag acg agt tgc | AC区 |
MLST是通过测定7个管家基因的核苷酸序列来发现细菌变异的分型方法。使用普通PCR方法扩增7个等位基因并测定其序列,方法参照文献[14]。PCR产物由天一辉远基因公司测序。测序结果递交MLST网上数据库进行比对,获得等位基因序列号、ST型和序列群。用Excel软件进行数据的整理分析。 1.2.4 菌株进化关系分析
采用BioNumerics构建多位点序列分型的最小生成树,分析菌株基因群结构和遗传进化关系。 2 结果 2.1 基因群分布
通过基因分群PCR鉴定,104株NG菌株中,36株为cnl菌株(34.6%),20株为B群(19.2%),14株为C群(13.4%),9株为W群(8.6%),9株为E群(8.6%),3株为X群(2.9%),3株为Y群(2.9%)(表 2)。10株菌株没有鉴定出基因群(所有分群引物扩增结果均为阴性),未发现其他基因群的菌株。
| 基因群 | 菌株数量 | ST序列群(菌株数) | ST型别(菌株数) | ST型数 |
| cnl | 36 | ST-198(36) | 2146(25);7960(1);8237(1);8239(1); 8243(2);10611(2);8717(1);10589(3) | 8 |
| B | 20 | ST-4821(2); ST41/44(1); UAb(17) | 3200(1);4821(1)10590(1)5542(1);5615(1);5819(2);5863(2);6927(2);6934(2);7157(1);7962(1);8490(1);8718(1);10591(1);10592(1);10746(1) | 16 |
| C | 14 | ST-4821(9); UA (5) | 4821(5);5473(4)5542(1);8714(1);8721(1);10755(1);10757(1) | 7 |
| W | 9 | ST-11(3); ST-174(6) | 11(3)6933(6) | 2 |
| Y | 3 | ST-175(1); ST-92(2) | 175(1)92(1);10606(1) | 3 |
| E | 9 | UA(9) | 5586(5);8238(1);10610(1);10616(1);10617(1) | 5 |
| X | 3 | UAb(3) | 5470(1);8438(1);8676(1) | 3 |
| NT | 10 | ST-4821(3); ST-175(2); UA(5) | 3200(1);4821(2)175(2)5542(1);5819(1);10108(1);10474(1);10615(1) | 8 |
| 注:(1)NT为基因未分群;UA为无序列群分类。 |
104株NG菌株共分为45种ST型,包括14种新的ST型,其中17个ST型可归为7个序列群(65株),ST-198序列群以ST-2146型为主(25株),C群的ST-4821序列群以ST-4821型为主(5株)。总体而言,数量最多的序列群为ST-198序列群,数量其次的为ST-4821序列群(图 1)。由图 1可以看出cnl菌株均属于ST-198序列群。ST-4821序列群涵盖B、C基因群和基因未分群,除ST-4821序列群外,已定义的同一序列群菌株都属于相同的基因群(不包括未分群)。反之,同一基因群的菌株可以包括不同的序列群。B群包括ST-4821和ST41/44两种序列群;C群包括ST-4821序列群;W 群包括ST-11和ST-174两种ST序列群;Y群包括ST-175和ST-92序列群;E群和X群的菌株都未能归入已定义的ST序列群。10株未能鉴定出基因群的菌株中,5株属于ST-4821和 ST-175序列群,另外5株未能归入已定义的ST序列群。
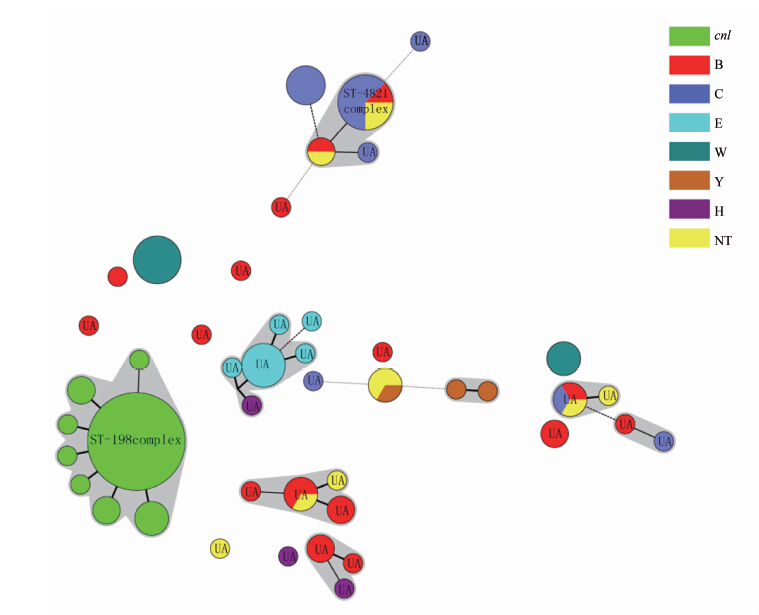
|
| 图 1 中国NG脑膜炎奈瑟菌多位点序列分型最小生成树 Figure 1 Minimum spanning tree of sequence type of 104 NG N.meningitidis strains 注:图中每个圈代表一种ST型,圈的大小代表菌株数量的多少,不同的颜色代表不同的基因群。 |
NG脑膜炎奈瑟菌株多为健康人群携带,较少致病。NG脑膜炎奈瑟菌形成的机制有3种,对于B、C、Y基因群,siaD基因滑链错配或者点突变使其终止密码子提前出现,从而使荚膜的生物合成和转运障碍,导致荚膜的缺失;其次IS元件的插入也可导致荚膜基因的不表达;第三种机制为荚膜基因A区和C区缺失所形成的cnl菌株[12]。前两种情况形成的菌株仍然可以用PCR进行基因分群。
本次研究对来自全国不同地区共104株NG脑膜炎奈瑟菌进行了基因群鉴定和MLST分型。基因群鉴定结果显示,我国NG脑膜炎奈瑟菌基因群主要为B、C、W、E,这与我国健康人群鼻咽部分离的可分群菌株的血清群一致[15, 16]。值得注意的是A群菌株,早期的研究结果显示,欧洲NG脑膜炎奈瑟菌中未发现A基因群菌株,但因为血清可分群的A群脑膜炎奈瑟菌在该地区非常少见,所以并未引起研究者的关注[12]。在我国,A群是目前我国主要流行菌群之一,而且在中西部还是优势菌群[16],然而本研究中我们也未发现基因群为A的菌株,提示A群菌株在荚膜基因的稳定性上不同于其他血清群。
我国血清可分群B群和C群的脑膜炎奈瑟菌的流行都以ST-4821序列群为主[17],本研究中的B群与C群的ST序列群均包括ST-4821序列群,基因群为W的NG脑膜炎奈瑟菌主要的ST序列群为ST-11和ST-174,并以ST-174序列群为主,ST-174和ST-11也是可分群W的主要序列群[15, 18]。这一结果说明我国NG脑膜炎奈瑟菌株与可分群脑膜炎奈瑟菌株的主要ST群一致。
本研究中,cnl菌株的MLST分型全部为ST-198序列群,而所有ST-198序列群也都是cnl菌株。cnl菌株缺少荚膜基因A区和C区,作者认为cnl菌株已不表达荚膜。由MLST数据库检索可知,国外cnl菌株并不全属于ST-198序列群,如第1株全基因组测序的cnl菌株(α14)属于ST-53序列群;国外ST-198序列群菌株也涵盖B、W、E、X等多个血清群。这一结果提示中国的ST-198序列群的cnl菌株在进化上可能属于一个独立的分支,这也是我国第一次对cnl菌株进行分析。
脑膜炎奈瑟菌的荚膜是脑膜炎奈瑟菌的主要毒力因子之一,可以抵御宿主补体介导的溶菌作用及单核巨噬细胞调理吞噬作用。然而,荚膜也是宿主黏膜免疫的靶成分,因此荚膜缺失反而有利于菌株的定植和侵袭[19, 20]。近几年的NG致病病例在全球范围内都有发生,提示我们需要对中国NG脑膜炎奈瑟菌的研究应引起重视。同时,NG脑膜炎奈瑟菌在人群中携带而又极少致病,提示其在维持机体内微生态系统平衡中是否起到一定的作用。本研究结果也显示,NG菌株与血清可分群菌株之间存在紧密的关系,因此,研究NG菌株也是阐释可分群菌株致病及流行特征的另一种角度和途径。
| [1] | Rosenstein NE, Perkins BA, Stephens DS, et al. Meningococcal disease[J]. N Engl J Med,2001,344(18):1378-1388. |
| [2] | Zhang YT,Xu L,Gao Y, et al. PCR genotyping of ungroupable Neisseria meningitidis[J].Disease Surveillance,2009,24(10):779-781.(in Chinese) 张艳亭,徐丽,高源,等. 不可分群脑膜炎奈瑟菌PCR基因分型研究[J].疾病监测,2009,24(10):779-781. |
| [3] | Goulding JN, Hookey JV, Stanley J, et al. Fluorescent amplified-fragment length polymorphism genotyping of Neisseria meningitidis identifies clones associated with invasive disease[J]. J Clin Microbiol,2000,38(12):4580-4585. |
| [4] | Xie O, Pollard AJ, Mueller JE, et al. Emergence of serogroup X meningococcal disease in Africa: need for a vaccine[J]. Vaccine,2013,31(27):2852-2861. |
| [5] | Vogel U, Claus H, von Muller L, et al. Bacteremia in an immunocompromised patient caused by a commensal Neisseria meningitidis strain harboring the capsule null locus (cnl)[J]. J Clin Microbiol,2004,42(7):2898-2901. |
| [6] | Hoang LM, Thomas E, Tyler S, et al. Rapid and fatal meningococcal disease due to a strain of Neisseria meningitidis containing the capsule null locus[J]. Clin Infect Dis,2005,40(5):e38-42. |
| [7] | Findlow H, Vogel U, Mueller JE, et al. Three cases of invasive meningococcal disease caused by a capsule null locus strain circulating among healthy carriers in Burkina Faso[J]. J Infect Dis,2007,195(7):1071-1072. |
| [8] | Zhou H, Shao Z, Li Q, et al. Neisseria meningitidis strain of unknown serogroup, China[J]. Emerg Infect Dis,2011,17(3):569-571. |
| [9] | Zhang L, Shao ZJ, Xu L, et al. A novel method for detection and sero-grouping of Nesseria meningitidis[J]. Chinese Journal of Epidemiology,2006,27(5):399-401.(in Chinese) 张力,邵祝军,徐丽,等. 鉴别脑膜炎奈瑟菌A、B、C、Y、W群的多重聚合酶链反应诊断方法[J].中华流行病学杂志,2006, 27(5):399-401. |
| [10] | Bennett DE, Mulhall RM, Cafferkey MT. PCR-based assay for detection of Neisseria meningitidis capsular serogroups 29E, X, and Z[J]. J Clin Microbiol,2004,42(4):1764-1765. |
| [11] | Zhu H, Wang Q, Wen L, et al. Development of a multiplex PCR assay for detection and genogrouping of Neisseria meningitidis[J]. J Clin Microbiol,2011,50(1):46-51. |
| [12] | Dolan-Livengood JM, Miller YK, Martin LE, et al. Genetic basis for nongroupable Neisseria meningitidis[J]. J Infect Dis,2003,187(10):1616-1628. |
| [13] | Harrison OB, Claus H, Jiang Y, et al. Description and nomenclature of Neisseria meningitidis capsule locus[J]. Emerg Infect Dis,2013,19(4):566-573. |
| [14] | Maiden MC, Bygraves JA, Feil E, et al. Multilocus sequence typing: a portable approach to the identification of clones within populations of pathogenic microorganisms[J]. Proc Natl Acad Sci USA,1998,95(6):3140-3145. |
| [15] | Yang M, Zhou HJ, Yuan H, et al. Analysis of molecular subtypes and microflora structure of Neisseria meningitidis strains isolated in Jiangxi province[J]. Chinese Journal of Preventive Medicine,2013,47(4):342-346. (in Chinese) 杨梦,周海健,袁辉,等. 江西省脑膜炎奈瑟菌分离株分子分型和菌群结构分析[J]. 中华预防医学杂志,2013,47(4):342-346. |
| [16] | Shao ZJ.Tending to prevalence and evolution of Cerebrospinal Meningitis in China[J]. Chinese Journal of Preventive Medicine,2013,47(10):891-893. (in Chinese) 邵祝军. 中国流行性脑脊髓膜炎流行变异趋势[J]. 中华预防医学杂志,2013,47(10):891-893. |
| [17] | Zhang J, Zhou HJ, Xu L, et al. Molecular characteristics of Neisseria meningitidis isolated during an outbreak in a jail: association with the spread and distribution of ST-4821 complex serogroup C clone in China[J]. Biomed Environ Sci, 2013,26(5):331-337. |
| [18] | Zhou H, Liu W, Xu L, et al. Spread of Neisseria meningitidis serogroup W clone,China[J]. Emerg Infect Dis,2013,19(9):1496-1499. |
| [19] | Hammerschmidt S, Hilse R, van Putten JP, et al. Modulation of cell surface sialic acid expression in Neisseria meningitidis via a transposable genetic element[J]. EMBO J, 1996, 15(1):92-98. |
| [20] | Moxon ER, Rainey PB, Nowak MA, et al. Adaptive evolution of highly mutable loci in pathogenic bacteria[J]. Curr Biol,1994,4(1):24-33. |
 2014, Vol. 29
2014, Vol. 29


