扩展功能
文章信息
- 朱健铭, 姜如金, 吴康乐, 翁幸鐾, 孔海深
- ZHU Jian-ming, JIANG Ru-jin, WU Kang-le, WENG Xing-bei, KONG Hai-shen
- 肺炎克雷伯菌泛耐药株的质粒耐药元件研究
- Resistant elements in plasmid of extensive drug resistant Klebsiella pneumonia
- 疾病监测, 2015, 30(2): 134-139
- Disease Surveillance, 2015, 30(2): 134-139
- 10.3784/j.issn.1003-9961.2015.02.013
-
文章历史
- 收稿日期:2014-10-17
2. 宁波市第一医院检验科, 浙江 宁波 315010;
3. 浙江大学医学院附属第一医院传染病诊治国家重点实验室, 浙江 杭州 310003
2. Medical Laboratory, No.1 Hospital of Ningbo, Ningbo 315010, Zheijiang, China;
3. State Key Laboratory for Communicable Disease Diagnosis and Treatment, No.1 Affiliated Hospital, School of Medicine, Zhejiang University, Hangzhou 310003, Zhejiang, China
肺炎克雷伯菌(Klebsiella pneumoniae)是医院内获得性感染的重要条件致病菌,可引起尿路感染、肺炎、败血症及软组织感染等疾病。近年来,随着抗菌药物的广泛使用,肺炎克雷伯菌的临床分离率和耐药性不断上升,尤为严重的是对碳青霉烯类耐药的肺炎克雷伯菌持续增加[1]。质粒是染色体外能够自主复制的环状DNA分子,在细菌耐药过程中发挥了重要作用,其不仅可以编码抗菌药物耐药基因、毒力基因等遗传信息,导致其宿主菌的耐药性和致病性的产生[2],还可以通过水平转移在相同或不同种属的细菌之间播散,易化了潜在的有利基因如抗菌药物耐药编码基因在细菌群体之间快速播散[3];此外,质粒能够捕获外源性DNA,以利于细菌定植于新环境,在宿主菌的适应和进化中也有着重要的作用[3]。
笔者采用第二代高通量测序技术平台对从重症监护病房(ICU)患者中分离到的1株泛耐药肺炎克雷伯菌(JM45株)进行全基因组测序,并利用生物信息学手段进行序列分析。全基因组测序除获得1条完整的基因组(染色体)序列外,并获得2条质粒序列。质粒1(pJM45-1)序列大小为317 156 bp(GC含量53.0%),质粒2(pJM45-2)序列大小为12 209 bp(GC含量55.3%),其中质粒pJM45-1为耐药性质粒。笔者已报道过该菌株对β-内酰胺类、氨基糖苷类和喹诺酮类药物的耐药机制[4, 5, 6]。为更全面地了解该菌株的耐药遗传基础,本研究将质粒pJM45-1之耐药元件进行分析,发现该质粒为可接合转移质粒,并携带254个基因,其中有83个耐药相关基因包括接合性质粒转移编码基因、多种抗菌药物耐药编码基因和重金属离子耐受编码基因、毒素编码基因以及大量的转座子和插入序列。pJM45-1质粒上携带众多的耐药编码基因,是细菌进化为泛耐药菌的主要动力;而pJM45-1又是可接合转移质粒,可将耐药基因在细菌间进行水平转移,引起耐药性的播散。
1 材料与方法 1.1 菌株来源和药敏试验肺炎克雷伯菌菌株JM45分离于2010年4月ICU患者的血液样本。患者男性,72岁,由于结肠癌术后脑梗死住院,同时伴有呼吸衰竭以及呼吸机相关肺炎,抗感染治疗方案为亚胺培南以及头孢哌酮/舒巴坦,1周后患者死亡。细菌的分离鉴定参照《全国临床检验操作规程》(第3版)进行,并经法国生物梅里埃公司VITEK2-Compact细菌鉴定仪鉴定和16S rDNA及gyrA基因测序进一步确认。药敏试验采用法国生物梅里埃公司的E-test条测量25种抗菌药物的最小抑菌浓度(MIC),分别为哌拉西林、头孢噻吩、头孢呋辛、头孢噻肟、头孢他啶、头孢吡肟、头孢西丁、氨曲南、亚胺培南、美罗培南、厄他培南、哌拉西林/他唑巴坦、头孢哌酮/舒巴坦、阿米卡星、妥布霉素、庆大霉素、诺氟沙星、氧氟沙星、环丙沙星、左氧氟沙星、四环素、氯霉素、甲氧苄啶/磺胺甲恶唑、磷霉素、多黏菌素B。根据2013年美国临床和实验室标准化协会(Clinical and Laboratory Standards Institute,CLSI)M100-S22文件解释标准判断试验药物敏感或耐药性[7]。药敏试验质控菌株为大肠埃希菌ATCC25922、金黄色葡萄球菌ATCC25923、铜绿假单胞菌ATCC27853和大肠埃希菌ATCC35218,购自杭州滨和微生物试剂有限公司。
1.2 基于焦磷酸测序技术的高通量测序采用天根生化科技(北京)有限公司的细菌基因组DNA提取试剂盒提取细菌总DNA,取5 μg的基因组总DNA,通过雾化(nebulization)将DNA打碎成小片段,利用美国Agencourt公司AMPure beads进行纯化。纯化的DNA利用美国罗氏应用科技的GS DNA Library Preparation 试剂盒制备成DNA 文库,并利用美国罗氏应用科技的GS emPCR 试剂盒固定在DNA捕获磁珠上,在一个油包水的体系里进行扩增,最后在罗氏454 公司的Genome Sequencer FLX 系统上进行高通量测序。高通量测序得到的片段读长约为400 bp,再利用罗氏454公司开发的Newbler software拼接软件进行序列拼接和组装,对于拼接后中断序列通过PCR扩增并测序的方法补充。最后得到1条完整的基因组序列及2条质粒序列。全基因组测序在国家人类基因组南方研究中心上海翰宇生物科技有限公司完成。
1.3 质粒基因组功能注释得到质粒基因组全序列后,首先采用软件Glimmer 3.02、GeneMark和Z-Curve program进行基因预测,插入序列(IS)结构的寻找则基于IS Finder数据库(http://www-is.biotoul.fr)。再通过BlastP比较美国国立生物技术信息中心(NCBI)(http://www.blast/ncbi.nlm.nih.gov)的非冗余蛋白质序列数据库(Nr)和日本KEGG蛋白数据库(http://www.genome.jp/kegg)进行基因功能注释,需要进一步确认功能的基因通过InterProScan软件明确功能域。并利用NCBI蛋白质结构域保守序列数据库(http://www.ncbi.nlm.nih.gov/Structure/cdd/cdd.shtml)进行直系同源体簇(COG)分类。
1.4 质粒圈图绘制利用网上在线工具(http://www.infosake.com/plasmid/plasmidFrame.php)绘制。
1.5 pJM45-1质粒序列比对及分子进化分析将pJM45-1质粒序列与已在NCBI登录的质粒序列作Blastn(http://www.blast/ncbi.nlm.nih.gov)比对。并点取已在NCBI登录的质粒序列,应用在线工具Distance tree of results作聚类分析(Fast Minimum Evolutin法)。
1.6 序列递交将肺炎克雷伯菌JM45的pJM45-1质粒全序列进行基因功能注释后,递交至NCBI GenBank数据库。
2 结果 2.1 肺炎克雷伯菌JM45的耐药谱肺炎克雷伯菌JM45除对多黏菌素B敏感外(MIC值为2 μg/ml),对其他24种抗菌药物均表现为耐药(MIC值均超过了E-test条最大检测值),提示多重耐药及交叉耐药情况相当严重,系泛耐药株。
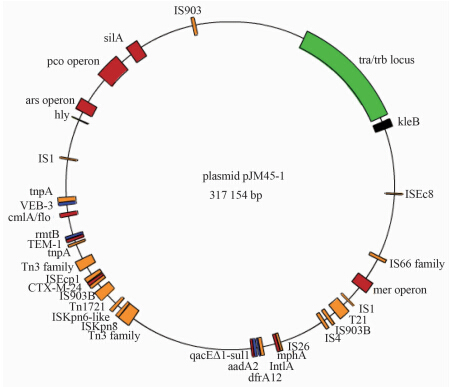
2.2 pJM45-1质粒耐药元件位置接合性质粒转移编码基因、抗菌药物耐药编码基因、重金属离子耐受编码基因、毒素编码基因和转座子以及插入序列在质粒圈图中相对位置,见图 1。
|
| 图 1 pJM45-1质粒接合性质粒转移编码基因、抗菌药物耐药编码基因、重金属离子耐受编码基因、毒素编码基因和转座子以及插入序列在质粒圈图中相对位置 Fig. 1 Locations of encoding genes of transfer genes of conjuagal plasmids,resistant genes to antibiotics and heavy metal ions,virulence factors and transposons and insertion sequences in plasmid pJM45-1 注:图中绿色为接合性质粒转移编码基因区,红色与蓝色为抗菌药物耐药编码基因与重金属离子耐受编码基因区,黑色为毒素编码基因区,桔黄色为转座子与插入序列区。 |
pJM45-1质粒序列全长317 156 bp,GC含量53.0%,共有254个各种编码基因,其中有83个耐药相关编码基因,包括33个接合性质粒转移编码基因、10个抗菌药物耐药编码基因、17个重金属离子耐受编码基因、2个毒素编码基因、8个转座子、12个插入序列以及1个Ⅰ类整合子整合酶编码基因。83个耐药相关编码基因的具体位置及功能注释,见表 1。pJM45-1质粒序列中254个各种编码基因已进行功能注释并登录于美国NCBI GenBank(登录号:CP006657)。
| Locus tag | 起始点 | 终点 | 长度(bp) | 基因名称 | 功能注释 | Locus tag | 起始点 | 终点 | 长度(bp) | 基因名称 | 功能注释 |
| N559_5135 | 2 668 | 2 261 | 408 | ISEc8 | 插入序列 | N559_5288 | 196 706 | 196 026 | 681 | pco peron | 铜离子耐受操纵子 |
| N559_5151 | 23 358 | 22 384 | 975 | IS66 family | 插入序列 | N559_5289 | 197 690 | 196 761 | 930 | ||
| N559_5158 | 30 287 | 30 652 | 366 | mer operon | 汞离子耐受操纵子 | N559_5290 | 198 075 | 197 695 | 381 | ||
| N559_5159 | 30 971 | 31 396 | 426 | N559_5291 | 200 828 | 199 011 | 1818 | ||||
| N559_5160 | 31 435 | 33 120 | 1686 | N559_5292 | 201 062 | 201 511 | 450 | ||||
| N559_5161 | 33 138 | 33 503 | 366 | N559_5297 | 209 056 | 205 910 | 3147 | silA | 银离子外排泵蛋白 | ||
| N559_5162 | 33 500 | 33 736 | 237 | N559_5310 | 227 210 | 226 242 | 969 | IS903 | 插入序列 | ||
| N559_5165 | 39 189 | 38 686 | 504 | IS1 | 插入序列 | N559_5341 | 261 213 | 261 605 | 393 | tra/trbC | 转移位座 |
| N559_5168 | 43 932 | 40 966 | 2967 | Tn21 | 转座子 | N559_5342 | 261 819 | 262 514 | 696 | ||
| N559_5169 | 44 495 | 43 935 | 561 | N559_5343 | 263 023 | 263 391 | 369 | ||||
| N559_5171 | 47 481 | 46 513 | 969 | IS903.B | 插入序列 | N559_5344 | 263 405 | 263 710 | 306 | ||
| N559_5173 | 49 556 | 48 588 | 969 | IS4 family | 插入序列 | N559_5345 | 263 730 | 264 296 | 567 | ||
| N559_5181 | 63 636 | 64 340 | 705 | IS26 | 插入序列 | N559_5346 | 264 283 | 265 023 | 741 | ||
| N559_5182 | 64 462 | 65 367 | 906 | mphA | 红霉素磷酸转移酶 | N559_5347 | 265 023 | 266 447 | 1425 | ||
| N559_5184 | 70 212 | 69 199 | 1014 | IntIA | Ⅰ类整合子整合酶 | N559_5348 | 266 440 | 267 036 | 597 | ||
| N559_5185 | 70 357 | 70 854 | 498 | dfrA12 | 甲氧苄胺嘧啶还原酶 | N559_5349 | 267 059 | 267 628 | 570 | ||
| N559_5186 | 71 274 | 72 053 | 780 | aadA2 | 氨基糖苷类腺苷转移酶 | N559_5350 | 267 760 | 268 170 | 411 | ||
| N559_5187 | 72 217 | 72 564 | 348 | qacE△1-sul1 | 季胺类消毒剂外排泵/二氢叶酸合成酶 | N559_5351 | 268 175 | 268 465 | 291 | ||
| N559_5190 | 77 473 | 76 796 | 678 | tetA | 四环素外排泵 | N559_5352 | 268 489 | 268 707 | 219 | ||
| N559_5223 | 114 072 | 111 067 | 3006 | Tn3 | 转座子 | N559_5353 | 268 708 | 269 025 | 318 | ||
| N559_5224 | 114 235 | 114 792 | 558 | N559_5354 | 269 092 | 269 496 | 405 | ||||
| N559_5225 | 114 915 | 115 895 | 981 | ISKpn8 | 插入序列 | N559_5355 | 269 527 | 269 784 | 258 | ||
| N559_5226 | 118 282 | 117 302 | 981 | ISKpn6-like | 插入序列 | N559_5356 | 269 798 | 270 190 | 393 | ||
| N559_5230 | 121 833 | 124 799 | 2967 | Tn1721 | 转座子 | N559_5357 | 270 262 | 272 901 | 2640 | ||
| N559_5232 | 127 434 | 126 466 | 969 | IS903.B | 插入序列 | N559_5358 | 273 290 | 273 916 | 627 | ||
| N559_5233 | 128 344 | 127 469 | 876 | CTX-M-24 | 超广谱β-内酰胺酶 | N559_5359 | 273 927 | 274 919 | 993 | ||
| N559_5234 | 129 856 | 128 594 | 1263 | ISEcp1 | 插入序列 | N559_5360 | 274 932 | 275 570 | 639 | ||
| N559_5236 | 135 441 | 132 475 | 2967 | Tn3-family | 转座子 | N559_5361 | 275 567 | 277 573 | 2007 | ||
| N559_5241 | 140 178 | 140 882 | 705 | tnpA | 转座酶 | N559_5362 | 277 605 | 277 841 | 237 | ||
| N559_5242 | 141 593 | 142 453 | 861 | TEM-1 | 窄谱β-内酰胺酶 | N559_5363 | 278 069 | 278 395 | 327 | ||
| N559_5243 | 142 623 | 143 378 | 756 | rmtB | 16S rRNA 甲基化酶 | N559_5364 | 278 416 | 279 168 | 753 | ||
| N559_5247 | 150 409 | 149 195 | 1215 | cmlA/flo | 氯霉素/砜霉素外排泵 | N559_5365 | 279 179 | 279 418 | 240 | ||
| N559_5248 | 152 735 | 153 634 | 900 | VEB-3 | 超广谱β-内酰胺酶 | N559_5366 | 279 390 | 279 962 | 573 | ||
| N559_5250 | 155 125 | 154 421 | 705 | tnpA | 转座酶 | N559_5367 | 279 955 | 280 383 | 429 | ||
| N559_5259 | 167 169 | 166 666 | 504 | IS1 | 插入序列 | N559_5368 | 280 370 | 281 740 | 1371 | ||
| N559_5267 | 179 474 | 179 271 | 204 | hly | 溶血素 | N559_5369 | 281 740 | 284 589 | 2850 | ||
| N559_5273 | 182 035 | 182 397 | 363 | ars operon | 砷离子耐受操纵子 | N559_5370 | 285 312 | 286 043 | 732 | ||
| N559_5274 | 182 415 | 184 166 | 1752 | N559_5371 | 286 403 | 288 712 | 2310 | ||||
| N559_5275 | 184 214 | 185 503 | 1290 | N559_5372 | 294 053 | 294 778 | 726 | ||||
| N559_5276 | 185 516 | 185 941 | 426 | N559_5373 | 294 935 | 295 531 | 597 | ||||
| N559_5286 | 194 412 | 193 978 | 435 | pco peron | 铜离子耐受操纵子 | N559_5375 | 297 015 | 299 336 | 2322 | kleB | 克雷伯毒素B |
| N559_5287 | 196 029 | 194 629 | 1401 |
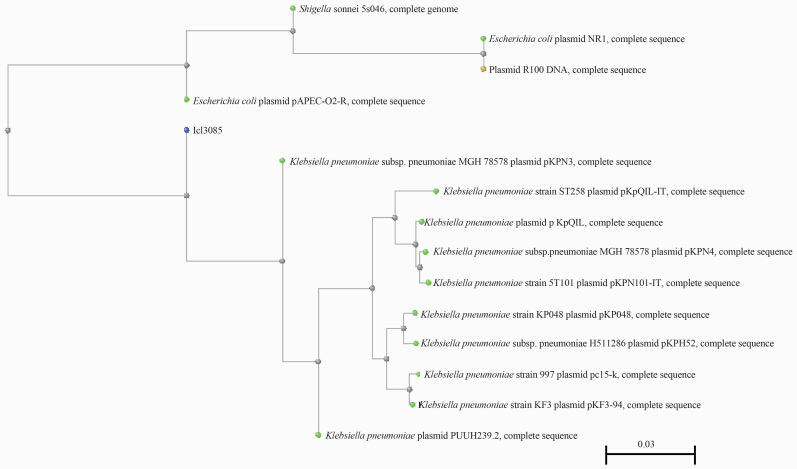
pJM45-1质粒与耐药质粒R100(登录号:AP000342.1)的全长94 281个序列中的45 995个序列有99%相同;与接合性质粒F质粒(登录号:AP001918.1)的全长99159个序列中的8322个序列有87%相同。分子进化分析显示pJM45-1质粒与源自肺炎克雷伯菌的质粒在同一簇(cluster),与源自其他肠杆菌的质粒不在一个簇,见图 2。
|
| 图 2 pJM45-1质粒与已在NCBI登录的质粒序列分子进化分析。 Fig. 2 Molecular evolutionary analysis of plasmid pJM45-1 and other plasmids recorded by NCBI 注:黄色荧光标签为pJM45-1质粒 |
肺炎克雷伯菌为革兰阴性菌,无芽孢,无鞭毛,有较厚的荚膜,多数有菌毛。对人致病性较强,是重要的条件致病菌和医源性感染菌。肺炎克雷伯菌的耐药形势日益严峻,国内外不断有新的耐药基因或基因亚型被发现[8, 9, 10]。由于肺炎克雷伯菌常携带质粒、转座子、插入序列和整合子等可移动遗传元件,介导耐药基因在菌间转移,从而大大加快了耐药基因的水平传播[11]。
质粒为存在于细菌中并独立于染色体之外的能进行自我复制的遗传因子。根据质粒的功能可分成4类:F质粒(曾称致育因子,fertility facter)、R质粒(resistance plasmid)、降解性质粒和致病性质粒。但质粒重组现象也十分常见,嵌合性质粒(chimeric plasmid)或称马赛克共合体(mosaic cointegrate)不断有报道,如:pTN48[12]、pSMS35_130[13]、pCooKm[14]、pB3[15]。质粒能赋予宿主菌某些特殊机能(例如:某些化合物的代谢机能、抗菌药物灭活酶、毒力因子等),使得宿主菌在环境生存中得到生长优势。其中耐药性质粒和降解性质粒能使宿主菌在有抗菌药物或有毒化合物的环境中生存(逆境生存)。宿主菌获得质粒是为了适应环境的一种进化。
肺炎克雷伯菌JM45分离自ICU结肠癌术后脑梗塞,同时伴有呼吸衰竭以及呼吸机相关肺炎患者的血液样本,为泛耐药株,对大多数抗菌药物均表现为耐药。本研究pJM45-1质粒经全基因组测序发现了83个耐药相关编码基因,携带接合性质粒转移编码基因(见表 1和图 1中的tra/trbC locus),因此pJM45-1是可接合转移质粒(F质粒),可将耐药基因在细菌间进行水平转移,造成耐药菌的播散。pJM45-1质粒并携带多种抗菌药物耐药编码基因与重金属离子耐受编码基因,抗菌药物耐药编码基因有:①β-内酰胺类药物耐药编码基因(TEM-1、CTX-M-24、VEB-3);②氨基糖苷类药物耐药编码基因(aadA2、rmtB);③红霉素耐药编码基因(mphA);④四环素耐药编码基因(tetA);⑤氯霉素与砜霉素耐药编码基因(cmlA);⑥磺胺类药物耐药编码基因(dfrA12、sul1);⑦季胺类药物耐药编码基因(qacE△1)。pJM45-1质粒上携带大量的耐药编码基因,是该菌株呈泛耐药的主要遗传学基础。重金属离子耐受编码基因有:①汞离子耐受编码基因(mer);②砷离子耐受编码基因(ars);③铜离子耐受编码基因(pco);④银离子耐受编码基因(silA)。重金属离子是强氧化剂,为杀菌剂。细菌获得这些重金属离子耐受编码基因意味着能抵抗重金属离子杀菌剂而存活。其次,还发现了毒素编码基因(hly、KleB),是构成该菌株毒力的主要物质基础。由表 1和图 1可见,pJM45-1质粒上存在众多的转座子以及插入序列。转座子与插入序列的存在意味着在其上下游的基因可高表达,它们自带基因的启动子可致相邻的耐药编码基因高表达[16, 17, 18, 19],并且与之相邻的耐药编码基因可被转座。另外,该菌株对碳青霉烯类药物表现为高度耐药,在pJM45-1质粒上未发现任何碳青霉烯类基因,经全基因组测序发现其染色体上存在4种全新的β-内酰胺酶结构域蛋白编码基因(包括金属β-内酰胺酶结构域蛋白编码基因),是导致该菌株耐碳青霉烯类药物的原因[4]。
由pJM45-1质粒与已在NCBI登录的质粒序列分子进化分析图可见,pJM45-1质粒与源自肺炎克雷伯菌的质粒在同一簇(cluster),与源自其他肠杆菌的质粒不在一个簇。
经NCBI Blastn比对,发现pJM45-1质粒与耐药质粒R100的全长94 281个序列中的45 995个序列有99%相同;与接合性质粒F质粒的全长99 159个序列中的8322个序列有87%相同。pJM45-1质粒携带接合性质粒转移编码基因、众多的转座子以及插入序列和大量抗菌药物耐药编码基因,因此它既是接合性质粒,又是耐药性质粒,所以该质粒属于嵌合性质粒或称马赛克共合体质粒。
本研究全长达317 156 bp的pJM45-1质粒为国内外首次发现(见与已在NCBI登录的质粒序列分子进化分析),在该质粒中发现了众多的质粒转移编码基因、抗菌药物耐药编码基因、重金属离子耐受编码基因和转座子以及插入序列等耐药元件。亦是肺炎克雷伯菌JM45抗菌药物耐药的遗传学背景。未来我们将收集相近耐药性的肺炎克雷伯菌,提取质粒DNA后直接进行高通量测序。了解pJM45-1质粒传播及分子进化进程,并结合菌株的分离时间相解析菌株传播途径,为阻断细菌传播提供依据。随着耐药菌株在临床环境中的不断进化,其耐药元件势必会越来越多,耐药水平越来越高,应该引起临床抗感染治疗的足够重视。
(志谢:本研究得到国家人类基因组南方研究中心郑华军老师的鼎力支持!)| [1] | Kong HS,Zhang WL,Yang Q,et al.Mohnarin report of 2011:monltoring of bacterial resistance in east China[J].Chinese Journal of Nosocomiology,2012,22(22):4971-4976.(in Chinese) 孔海深,张伟丽,杨青,等.Mohnarin2011年度报告:华东地区细菌耐药监测[J].中华医院感染学杂志,2012,22(22):4971-4976. |
| [2] | Da Silva GJ,Mendonca N.Association between antimicrobial resistance and virulence in Escherichia coli[J].Virulence,2012,3(1):18-28. |
| [3] | Lynch JP,Clark NM,Zhanel GG.Evolution of antimicrobial resistance among Enterobacteriaceae:focus on extended spectrum beta-lactamases and carbapenemases[J].Expert Opin Pharmacother,2013,14(2):199-210. |
| [4] | Zhu JM,Jiang RJ,Weng XB,et al.Whole-genome sequence-based analysis of Klebsiella pneumoniae JM45[J].Chinese Journal of Clinical Infectious Diseases,2014,7(1):27-33.(in Chinese) 朱健铭,姜如金,翁幸鐾,等.肺炎克雷伯菌JM45株全基因组测序分析[J].中华临床感染病杂志,2014,7(1):27-33. |
| [5] | Zhu JM,Jiang RJ,Wu JL, et al.Comparative genomics analysis of aminoglycoside resistance in multi-drug resistant Klebsiella pneumoniae JM45 isolate[J]. Chinese Journal of Infectious Diseases,2013,31(6):327-330.(in Chinese) 朱健铭,姜如金,吴晋兰,等.耐药肺炎克雷伯菌JM45株氨基糖苷类药物耐药比较基因组学分析[J].中华传染病杂志,2013,31(6):327-330. |
| [6] | Zhu JM,Jiang RJ,Wu KL, et al.Analysis of quinolone resistance genes in Klebsiella pneumoniae JM45 by whole genome sequencing[J]. Chinese Journal of Nosocomiology,2014,24(10):2341-2344. (in Chinese) 朱健铭,姜如金,吴康乐,等.全基因测序法分析肺炎克雷伯菌JM45株对喹诺酮类药物耐药基因[J].中华医院感染学杂志,2014,24(10):2341-2344. |
| [7] | Clinical and Laboratory Standards Institute.Performance standards for antimicrobial susceptibility testing,Twenty-Second informational Supplement.M100-S22[S].Wayne,PA:CLSI,2013. |
| [8] | Pereira PS, de Araujo CF, Seki LM, et al. Update of the molecular epidemiology of KPC-2-producing Klebsiella pneumoniae in Brazil: spread of clonal complex 11 (ST11, ST437 and ST340) [J]. J Antimicrob Chemother,2013,68(2):312-316. |
| [9] | Hirsch EB, Tam VH. Detection and treatment options for Klebsiella pneumoniae carbapenemases (KPCs):an emerging cause of multidrug-resistant infection[J]. J Antimicrob Chemother,2010,65 (6):1119-1125. |
| [10] | Zhu JM, Jiang RJ, Mi ZH, et al. Novel KPC variant from a carbapenem-resistant strain of K. pneumoniae in a Chinese hospital[J]. Infect Control Hosp Epidemiol,2011,32(10):1050-1052. |
| [11] | Mindlin SZ,Petrova MA,Bass IA,et al.Origin,evolution,and migration of drug resistance genes[J].Genetika,2006,42(11):1495-1511. |
| [12] | Villard-Pomares T,Tenaillon O,Le Nagard H,et al.Complete nucleotide sequence of plasmid pTN48,encoding the CTX-M-14 Extended-Spectrum β-Lactamase from an Escherrichia coli O102-ST405 strain[J]. Antimicrob Agents Chemother,2011,55(3):1270-1273. |
| [13] | Fricke WF,Wright MS,Lindell AH,et al.Insights into the environmental resistance gene pool from the genome sequence of the multidrug-resistant environmental isolate Escherrichia coli SMS-3-5[J].J Bacteriol,2008,190(20):6779-6794. |
| [14] | Froehlich B,Parkhill J,Sanders M,et al.The pCoo plasmid of enterotoxigenic Escherrichia coli is a mosaic cointegrate[J]. J Bacteriol,2005,187(18):6509-6516. |
| [15] | Heuer H,Szczepanowski R,Schneiker S,et al.The complete sequences of plasmids pB2 and pB3 provide evidence for a recent ancestor of the IncP-1βgroup without any accessory genes[J].Microbiology,2004,150:3591-3599. |
| [16] | Zhu JM,Chu XL,Jiang RJ,et al.Tnp513 Transposase Gene in Multi-drug Resistant Acinetobacter baumannii[J]. Chinese Journal of Nosocomiology,2009,19(20):2678-2681. (in Chinese) 朱健铭,禇雪莲,姜如金,等.多药耐药鲍氏不动杆菌tnp513转座酶基因研究[J].中华医院感染学杂志,2009,19(20):2678-2681. |
| [17] | Figueiredo S,Poirel L,Papa A,et al.Overexpression of the naturally occurring blaOXA-51 gene in Acinetobacter baumannii mediated by novel insertion sequence ISAba9[J].Antimicrob Agents Chemother,2009,53(9):4045-4047. |
| [18] | Ryuichi Nakano R,Okamoto R,Nagano N,et al.Resistance to gram-negative organisms due to high-level expression of plasmid-encoded ampC β-lactamase blaCMY-4 promoted by insertion sequence ISEcp1[J].J Infect Chemother,2007,13:18-23. |
| [19] | David AB,Tyler S,Christianson S, et al. Complete nucleotide sequence of a 92-Kilobase plasmid harboring the CTX-M-15 extended-spectrumβ-Lactamase involved in an outbreak in long-term-care facilities in toronto, Canada[J].Antimicrob Agents Chemother,2004,48(10):3758-3764. |
 2015, Vol. 30
2015, Vol. 30


