扩展功能
文章信息
- 杜鹏程, 阚飙, 王多春
- DU Peng-cheng, KAN Biao, WANG Duo-chun
- 霍乱弧菌中VSP-Ⅰ岛的分布和变异特征研究
- Distribution and variation of VSP-Ⅰ island in Vibrio cholerae
- 疾病监测, 2015, 30(5): 353-357
- Disease Surveillance, 2015, 30(5): 353-357
- 10.3784/j.issn.1003-9961.2015.05.004
-
文章历史
- 收稿日期:2015-02-09
霍乱是一种烈性肠道传染病,在我国作为法定甲类传染病进行管理。其病原体为霍乱弧菌(Vibrio cholerae)。目前已发现霍乱弧菌有超过200个血清群,其中只有O1和O139血清群能引起霍乱暴发[1]。历史上已有7次世界性霍乱大流行,其中前6次由O1血清群古典生物型引起,目前所处的第7次大流行由O1血清群El Tor生物型引起。1992年在印度东南部发现了新的可引起霍乱暴发的O139血清群,被认为可能成为引起世界性霍乱流行的新型别,但目前表现为在东南亚地区的地区性流行,并未广泛扩散至世界其他地区造成暴发流行[2]。
霍乱弧菌的致病性和其基因组上的可移动元件密切相关,O1和O139群霍乱弧菌流行菌株的主要致病因子为CTXΦ噬菌体所携带基因编码的霍乱肠毒素(cholera toxin,CT)[3],其他元件包括毒力协同调节菌毛(toxin co-regulated pili,TCP)、霍乱弧菌致病岛(V. cholerae pathogenicity island,VPI)、霍乱第7次大流行基因岛1和2(Vibrio seventh pandemic island Ⅰ and Ⅱ,VSP-Ⅰ/Ⅱ)等[4]。其中,VSP-Ⅰ是一个整合在第7次霍乱大流行菌株染色体上的移动元件,其大小为16 kb,G+C含量为40%,低于霍乱弧菌基因组的G+C含量(47%),提示VSP-Ⅰ为外源性元件[5]。而VSP-Ⅰ在霍乱弧菌中的分布特点、序列特征和可能的功能,目前仍不清楚。
本研究通过比较基因组学方法,分析VSP-Ⅰ岛在目前已发表霍乱弧菌基因组上的分布特点和序列特征,注释VSP-Ⅰ岛上各基因可能的功能,阐述VSP-Ⅰ岛的序列变异和水平转移特点,为解释霍乱弧菌VSP-Ⅰ岛的获得提供依据,同时为研究其他可移动元件建立基础。
1 材料与方法 1.1 霍乱弧菌基因组数据的获取和分析从美国国立生物技术信息中心(national center for biotechnology information,NCBI)的GenBank数据库(ftp://ftp.ncbi.nlm.nih.gov/genbank/genomes)中,下载截至2014年底已发表的所有霍乱弧菌基因组序列,包括基因组完成图和基因组草图。对于仅发表序列未进行基因预测和功能注释的基因组使用Glimmer软件进行了基因预测。
1.2 VSP-Ⅰ岛的识别和获取以霍乱第7次世界大流行代表菌株,O1血清群El Tor生物型菌株N16961(基因组序列号:NC_002505)的VSP-Ⅰ岛作为参考序列,包含11个编码基因,序号为 VC0175~VC0185 。使用BLAST 2.2.17软件进行核酸序列比对,在下载得到的所有霍乱弧菌基因组序列上,搜索VSP-Ⅰ岛携带的11个编码基因,比对长度占基因长度50%以上、相似度在50%以上作为阳性结果,识别到3个以上基因且间距小于10 kb的区域作为VSP-Ⅰ岛。使用PERL语言脚本进行批量处理并获取基因组上的VSP-Ⅰ岛序列。
1.3 VSP-Ⅰ岛SNP位点分析及所在基因组进化分析使用MUMmer 3.23软件对VSP-Ⅰ岛及所有霍乱弧菌基因组进行单核苷酸多态性分析(single nucleotide polymorphism,SNP),对VSP-Ⅰ岛上的SNP分布进行研究;并对基因组上所有SNP位点的串联序列在Mega 5.0 软件中使用邻接法进行进化分析,bootstrap值设置为100。
1.4 VSP-Ⅰ岛同源基因分析、序列比对及功能注释使用OrthMCL 1.4 软件对所获得的VSP-Ⅰ岛进行同源基因分析。使用BLAST软件进行两两间序列比对,识别并分析插入缺失等序列结构变异。功能注释使用NCBI网站的BLAST工具(http://blast.ncbi.nlm.nih.gov/Blast.cgi),与非冗余蛋白序列库(NT)、SwissProt蛋白序列库、蛋白质资料库(PDB)、蛋白序列数据库(PIR)和蛋白研究基金会数据库(PRF)进行比对完成。
2 结果 2.1 VSP-Ⅰ岛在霍乱弧菌中的分布从GenBank数据库共获得179个霍乱弧菌基因组,包括7个基因组完成图和172个基因组草图。基因组水平进化分析显示,第7次大流行的O1群El Tor型、O139群菌株高度聚集为一簇,而O1群古典型、O1群非流行株和非O1非O139群霍乱弧菌则较为分散。基因比对结果发现,VSP-Ⅰ岛广泛存在于107株霍乱弧菌中(107/179,59.8%),其中第7次大流行中分离的O1及O139血清群菌株中(105株),均携带VSP-Ⅰ岛;在其他血清群中,2株非O1非O139群菌株VL426和ATCC14547携带VSP-Ⅰ,见图 1。
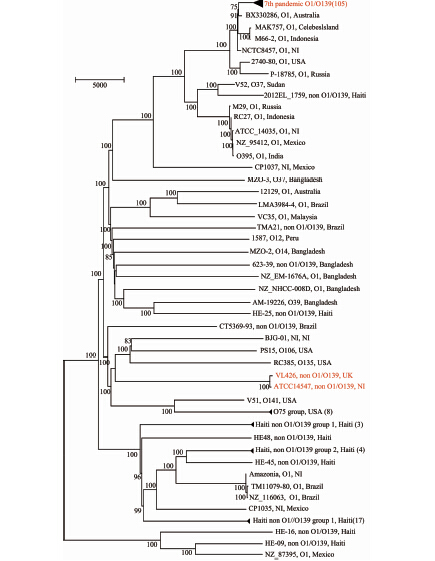
|
| 图 1 霍乱弧菌基因组进化树 Fig. 1 Phylogenetic tree of V. cholerae genomes 注:菌株名称、血清群、分离地点依次用“,”分隔,NI表示无相关信息,红色菌株携带VSP-Ⅰ,部分菌株合并为一组,括号中数据为菌株数量。 |
在霍乱第7次大流行的O1和O139群霍乱弧菌中,VSP-Ⅰ岛的序列高度保守,105株菌株中,94株的VSP-Ⅰ岛完全一致,其余11株与其相比分别存在1~14个SNP位点,其中海地2010年霍乱暴发分离株O1型HC50A2中SNP位点最多,共20个SNP差异位点,包括基因间区4个,同义突变1个,非同义突变15个。在VSP-Ⅰ岛的基因中, VC0814 的SNP差异位点最多,为7个,其次是 VC0813 ,为5个,见图 2。
同源基因分析表明,在霍乱弧菌VSP-Ⅰ岛的11个基因中,有9个共有保守基因,包括 VC0176、VC0177、VC0178、VC0179、VC0180、VC0181、VC0182、VC0184和VC0185,见图 3,存在于所有霍乱弧菌(O1、O139和非O1非O139菌株)中。但第7次大流行的菌株(O1、O139群)与非O1非O139菌株相比,在 VC0177 和 VC0178的基因间区存在2个小片段的插入缺失,在 VC0813 上存在1个小片段插入缺失,导致在非O1非O139菌株(VL426和ATCC 14547)中 VC0183 仅存在3′末端部分序列残余。 VC0183 虽在所有菌株中均存在,但在2株非O1非O139菌株中长度变化较大,只有3′末端部分序列残余,可能是由于该区域发生序列插入缺失或重组导致,见图 3。
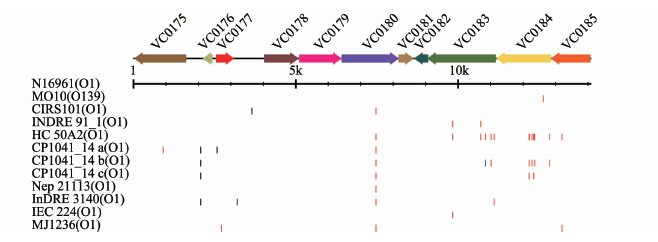
|
| 图 2 O1和O139型霍乱弧菌VSP-Ⅰ岛上SNP位点分布 Fig. 2 Distribution of SNPs in VSP-I from O1 and O139 注:黑、蓝、红色竖线依次为基因间区变异、同义突变和非同义突变。 |
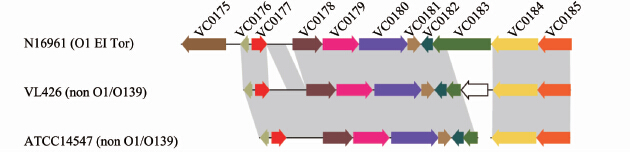
|
| 图 3 非O1非O139霍乱弧菌中VSP-Ⅰ岛与第7次大流行代表菌株的基因同源性和序列比对 Fig. 3 Orthologous genes and sequence alignments of VSP-I in non O1,non O139 strains with that in 7th pandemic strains |
在霍乱弧菌VSP-Ⅰ岛的11个基因中, VC0175 仅存在于105株第7次大流行的O1和O139群菌株中,其他10个基因均存在于所有携带VSP-Ⅰ的菌株中,见表 1。功能注释上,VSP-Ⅰ岛携带5个与该岛的转移和整合相关的基因 ( VC0181 ~ VC0185 ),其中4个基因( VC0182 ~ VC0185 ) 的编码蛋白注释为噬菌体整合酶或转座酶,功能预测为与噬菌体相关的整合酶或与移动元件有关的转座酶,提示该区域可能来源于噬菌体或其他转座元件; VC0181 编码结合转导相关蛋白,可能与该片段的插入有关。 VC0178编码马铃薯糖蛋白类似物, VC0180 编码蛋白水解酶,二者可能与毒力相关。另外, VC0175 虽然编码蛋白注释为脱氧胞苷酸脱氨酶,但其功能尚未知; VC0177 和 VC0179 的编码蛋白注释为假设蛋白,其功能也待鉴定。
| 基因 | 序列长度(bp) | 同源基因数量 | 编码蛋白注释 | 功能注释 | GenBank编号 |
| VC0175 | 1599 | 105 | 脱氧胞苷酸脱氨酶 | 未知 | AAF93351.1 |
| VC0176 | 309 | 107 | 转录调节因子 | 调控功能 | AAF93352.1 |
| VC0177 | 564 | 107 | 假设蛋白 | 未知 | AAF93353.1 |
| VC0178 | 1068 | 107 | 马铃薯糖蛋白类似物 | 可能与毒力相关 | AAF93354.1 |
| VC0179 | 1311 | 107 | 假设蛋白 | 未知 | AAF93355.1 |
| VC0180 | 1755 | 107 | 蛋白水解酶 | 可能与毒力相关 | AAF93356.1 |
| VC0181 | 471 | 107 | 结合转移相关蛋白 | 基因的插入 | AAF93357.1 |
| VC0182 | 432 | 107 | 噬菌体整合酶 | 负责噬菌体的整合 | AAF93358.1 |
| VC0183 | 2112 | 107 | 噬菌体整合酶 | 负责噬菌体的整合 | AAF93359.1 |
| VC0184 | 1689 | 107 | 整合酶 | 负责移动元件的整合 | AAF93360.1 |
| VC0185 | 1215 | 107 | 转座酶 | 促进移动元件转座 | AAF93361.1 |
霍乱弧菌是在全球范围内尤其是卫生条件较差的发展中国家,对人类健康造成重要影响的病原体之一。目前所处的霍乱第7次大流行,是由产毒(CT)的O1和O139群霍乱弧菌引起。以往认为,VSP-Ⅰ岛是霍乱第7次大流行菌株中特异的基因簇[6],本研究对GenBank 中所有的霍乱弧菌基因组序列搜索VSP-Ⅰ岛,并对VSP-Ⅰ片段在霍乱弧菌中的分布和变异进行了研究。通过在179个霍乱弧菌基因组序列中的比较分析发现,霍乱第7次大流行菌株中均携带VSP-Ⅰ岛,且高度保守。同时,笔者发现2株非O1非O139霍乱弧菌也携带VSP-Ⅰ岛,但与流行株相比,这2株菌株的VSP-Ⅰ岛部分基因长度上存在差异,可能是由于发生序列插入缺失或重组导致,非O1非O139菌株VSP-Ⅰ岛的这种变异,可能与其在水平转移过程中对不同菌株的适应性相关。非O1非O139霍乱弧菌很少产毒,只引起轻症胃肠炎或腹泻[7],因而不属于霍乱第7次大流行菌株。除本研究中发现携带VSP-Ⅰ的菌种,在之前使用PCR筛查发现,环境来源的非O1非O139霍乱弧菌中,VSP-Ⅰ岛的阳性率为5.4%,但多数菌株的VSP-Ⅰ与流行株间存在变异[8]。有文献报道在拟态弧菌中也有发现[5]。因此,作为霍乱第7次大流行菌株中的特异性基因簇,VSP-Ⅰ已转移至非O1非O139群菌株,甚至其他弧菌中,并发生了变异。
霍乱第7次大流行的O1和O139群霍乱弧菌,在基因组上具有很高的相似性,呈现出高度克隆化,其祖先菌株在进化过程中逐步获得了目前所携带的移动元件。这些元件可通过水平基因转移在细菌间进行传递,使受体菌获得新的功能和性状,是病原菌进化的重要机制之一。霍乱弧菌获得的最重要的移动元件,是携带霍乱毒素基因的噬菌体CTXΦ[3]。同时,霍乱弧菌在感染了CTXΦ后成为致病菌并大量繁殖,比非致病菌株具有更强的生存优势,并导致霍乱的发生与传播[9]。此外,在20世纪80年代后期以来分离的部分流行菌株中还发现了一个和耐药性密切相关的SXT岛[10]。这些移动元件的GC含量与霍乱基因组平均GC含量有明显差别,插入位点多为tRNA且两侧有正向重复序列,具有典型的基因组岛的结构特点。而VSP-Ⅰ岛整合位点不是tRNA,两侧无正向重复序列,但编码XerDC样重组酶,也可从基因组上脱离并环化,其机制尚不明确[11]。本研究通过比较基因组学方法,分析VSP-Ⅰ岛在目前已发表的弧菌基因组上的分布特点和序列特征,阐述弧菌VSP-Ⅰ岛的序列变异特点,为解释霍乱弧菌VSP-Ⅰ岛的获得提供依据,同时为研究其他可移动元件建立了基础。
病原学和基因组学研究表明,基因组上携带的众多可移动元件与其致病性密切相关。对VSP-Ⅰ岛上的同源基 因分布和功能分析表明,在霍乱第7 次大流行菌株携带的VSP-Ⅰ岛及部分相似度较高的序列上存在可能与毒力相关的特异基因,包括 VC0178~ VC0181 共4个基因。其中 VC0178编码马铃薯糖蛋白类似物,该类物质在植物中与抵抗寄生虫等功能相关,能够引发细胞凋亡。在动物模型和人类感染中已证明,铜绿假单胞菌中的该类物质ExoU蛋白是三型分泌系统的效应蛋白之一,与肺部损伤、败血症及细菌播散相关[12]。VSP-Ⅰ岛中广泛存在和基因重组、序列插入缺失密切相关的基因,包括转座酶、整合酶、结合转移酶等,此外也发现了一些功能尚不明确的特异片段,说明该区域存在着较为频繁的与水平基因转移密切相关的序列变异,这些变异对应的功能变化和对霍乱弧菌性状的影响还需进一步研究。
| [1] | Kaper JB, Morris JG Jr, Levine MM. Cholera[J]. Clin Microbiol Rev,1995,8(1):48-86. |
| [2] | Bhattacharya MK, Bhattacharya SK, Garg S, et al. Outbreak of Vibrio cholerae non-O1 in India and Bangladesh[J]. Lancet,1993,341(8856):1346-1347. |
| [3] | Waldor MK, Mekalanos JJ. Lysogenic conversion by a filamentous phage encoding cholera toxin[J]. Science,1996,272(5270):1910-1914. |
| [4] | Faruque SM, Chowdhury N, Kamruzzaman M, et al. Genetic diversity and virulence potential of environmental Vibrio cholerae population in a cholera-endemic area[J]. Proc Natl Acad Sci USA,2004,101(7):2123-2128. |
| [5] | Grim CJ, Choi J, Chun J, et al. Occurrence of the Vibrio cholerae seventh pandemic VSP-Ⅰ island and a new variant[J]. OMICS,2010,14(1):1-7. |
| [6] | Rahman MH, Biswas K, Hossain MA, et al. Distribution of genes for virulence and ecological fitness among diverse Vibrio cholerae population in a cholera endemic area: tracking the evolution of pathogenic strains [J]. DNA Cell Biol,2008,27(7):347-355. |
| [7] | Morris JG Jr, Picardi JL, Lisb S, et al. Non-O group 1 Vibrio cholerae: a look at the epidemiology of an occasional pathogen[J]. Epidemiol Rev, 1990, 12(1):179-191. |
| [8] | Li FJ, Du PC, Li BS, et al. Distribution of virulence-associated genes and genetic relationships in non-O1/O139 Vibrio cholerae aquatic isolates from China[J]. Appl Environ Microbiol,2014,80(16):4987-4992. |
| [9] | Karaolis DKR, Johnson JA, Bailey CC, et al. A Vibrio cholerae pathogenicity island associated with epidemic and pandemic strains[J]. Proc Natl Acad Sci USA,1998, 95(6):3134-3139. |
| [10] | Spagnoletti M, Ceccarelli D, Rieux A, et al. Acquisition and evolution of SXT-R391 integrative conjugative elements in the seventh-pandemic Vibrio cholerae lineage[J]. MBio,2014,5(4):e01356. |
| [11] | Murphy RA, Boyd EF. Three pathogenicity islands of Vibrio cholerae can excise from the chromosome and form circular intermediates[J]. J Bacteriol,2008,190(2):636-647. |
| [12] | Banerji S, Flieger A. Patatin-like proteins: a new family of lipolytic enzymes present in bacteria?[J]. Microbiology,2004,150(3):522-525. |
 2015, Vol. 30
2015, Vol. 30


