扩展功能
文章信息
- 焦素黎, 胡逢蛟, 张姝, 李永东, 倪红霞
- JIAO Su-li, HU Feng-jiao, ZHANG Shu, LI Yong-dong, NI Hong-xia
- 2003-2014年浙江省宁波市乙型流行性感冒病毒血凝素和神经氨酸酶基因变化特征分析
- Mutation of hemagglutinin and neuraminidase genes of influenza B virus isolated in Ningbo,Zhejiang,2003-2014
- 疾病监测, 2015, 30(6): 448-453
- Disease Surveillance, 2015, 30(6): 448-453
- 10.3784/j.issn.1003-9961.2015.06.005
-
文章历史
- 收稿日期:2014-12-17
乙型流行性感冒(流感)病毒较甲型流感病毒相比较没有亚型之分,但根据其抗原性和基因特征可以分为多个抗原谱系。自1983年以来逐渐演变成为以B/Yamagata/16/88和B/Victoria/2/87为代表株的两大谱系[1]。这两大谱系间的流感病毒抗原差异非常大,经常交替流行。本研究通过对宁波市2003 2014年间所分离到的乙型流感病毒的2个主要表面抗原血凝素(HA)和神经氨酸酶(NA)基因进行测序,分析近12年来宁波市乙型流感病毒的基因特性。现将结果报告如下。
1 材料与方法 1.1 标本采集2003 2014年每周从宁波市流感监测哨点医院收集流感样患者的咽拭子或含漱液标本,采样均发病3天内,体温38 ℃以上。标本立即于冷藏条件下运送到实验室进行病毒分离。
1.2 病毒分离标本经抗生素处理后接种于长成单层的MDCK细胞上,置35 ℃ 5% CO2箱培养逐日观察细胞病变。细胞病变完全后收集培养物上清,用1%豚鼠血球和标准血清进行血凝和血凝抑制(HI)试验进行型别鉴定。
1.3 核苷酸序列测定采用德国Qiagen公司的Rneasy Mini Kit试剂盒中的操作方法提取病毒RNA。反转录-聚合酶链反应(RT-PCR)的扩增采用日本TaKaRa公司的Ex Taq Premix试剂,反应条件95 ℃ 5 min;95 ℃ 60s 54 ℃ 45s 72 ℃ 2 min,35个循环;72 ℃ 10 min。核酸扩增及序列测定所用的引物序列如下:HA-F 5′-AAT ATC CAC AAA ATG AAG GCA ATA-3′;HA-R 5′-GTA ATG GTA ACA AGC AAA CAA GCA-3′;NA-F 5′-GCA CTC CTA ATT AGC CCT CAT AGA-3′;NA-R 5′-CAG AAA CAA TTA AGT CCA GTA AGG-3′。上述引物由美国Invitrogrn生物公司合成并进行双向序列测定,分别测定HA、NA 2个基因片段的全长。
1.4 序列分析采用Contig Express软件做序列拼接。用DNAstar的MegAlign 5.21 序列分析软件进行分析处理。用 Mega 5.02软件构建病毒的基因系统进化树,分析中涉及的HA及NA基因的参考序列从美国的NCBI的GenBank中下载。
2 结果 2.1 流感病毒监测结果宁波市2003 2014年9月共检测流感样病例标本16 299份,流感病毒阳性1927份,病毒分离率为11.82%。其中甲1型流感病毒 (包括季节性甲1型和新甲型H1N1型)607份(31.50%),甲3型流感病毒1050份(54.49%),乙型流感病毒270份(14.01%)。270份的乙型流感病毒中198份为Victoria系毒株,72份为Yamagata系毒株。可见虽然近2003年以来宁波市流感流行株以甲型流感占优势,但乙型流感也一直存在,见表1。
| 年份 | 样本数 | 病毒分离阳性数 | 分离率(%) | 甲3型流感病毒 | 甲1型流感病毒 | 乙型Victoria系病毒 | 乙型Yamagata系病毒 | ||||
| 阳性数 | 阳性率(%) | 阳性数 | 阳性率(%) | 阳性数 | 阳性率(%) | 阳性数 | 阳性率(%) | ||||
| 2003(1) | 368 | 76 | 20.65 | 73 | 96.05 | 0 | 0.00 | 1 | 1.32 | 2 | 2.63 |
| 2004 | 1 466 | 248 | 16.92 | 234 | 94.35 | 0 | 0.00 | 1 | 0.40 | 13 | 5.24 |
| 2005 | 1 620 | 356 | 21.98 | 290 | 81.46 | 56 | 15.73 | 5 | 1.40 | 5 | 1.40 |
| 2006 | 1 290 | 145 | 11.24 | 6 | 4.14 | 103 | 71.03 | 36 | 24.83 | 0 | 0.00 |
| 2007 | 1 223 | 117 | 9.57 | 113 | 96.58 | 1 | 0.85 | 1 | 0.85 | 2 | 1.71 |
| 2008 | 1230 | 57 | 4.63 | 18 | 31.58 | 32 | 56.14 | 4 | 7.02 | 3 | 5.26 |
| 2009 | 2 359 | 410 | 17.38 | 128 | 31.21 | 43+207(2) | 60.98 | 32 | 7.80 | 0 | 0.00 |
| 2010 | 1 812 | 183 | 10.10 | 31 | 16.93 | 58 | 31.69 | 89 | 48.63 | 5 | 2.73 |
| 2011 | 864 | 30 | 3.47 | 3 | 10.00 | 14 | 46.67 | 11 | 36.67 | 2 | 6.67 |
| 2012 | 866 | 23 | 2.66 | 5 | 21.73 | 0 | 0.00 | 18 | 78.26 | 0 | 0.00 |
| 2013 | 1 658 | 69 | 4.16 | 18 | 26.09 | 33 | 47.83 | 0 | 0.00 | 18 | 26.09 |
| 2014 | 1 543 | 213 | 14.14 | 131 | 61.50 | 60 | 28.17 | 0 | 0.00 | 22 | 10.33 |
| 合计 | 16 299 | 1927 | 11.82 | 1050 | 54.49 | 607 | 31.50 | 198 | 10.28 | 72 | 3.74 |
| 注:(1)2003年只有流行高峰的检测资料,因此数据只反应高峰期的检出率。(2)甲1型流感中2003 2009年的43份为季节性甲1流感,2009年207份起至2014年均为甲型H1N1流感。 | |||||||||||
Victoria系毒株自2003年以来(2013 2014年未分离到)与疫苗株Hongkong/330/2001相比在HA1基因片段中累计有16个位点出现过氨基酸替换,其中 2005年底及2009年2个年度较为明显,2005年底新增加了3个(48、80、129)氨基酸位点的变异,2009年又新增了4个(58、75、165、172)氨基酸位点的变异。而Yamagata系毒株与Victoria系毒株相比在HA1基因上的变异相对较快,与疫苗株Shanghai/361/2002相比有22个位点的氨基酸发生过变异,其中最明显的发生于2007年和2010年两个年份,较往年相比分别有8个(48、108、125、129、180、233、252、256)、9个(48、108、116、136、146、150、166、203、233)氨基酸位点变异,见图1、2。而Victoria系毒株的S197N位点的变异均使HA受体结合位点附近形成了一个新的糖基化位点。

|
| 图1 2003-2014年乙型流感Victoria系病毒 Fig.1 Mutation loci of HA gene of influenza B virus Victoria lineage,2003-2014 注:“.”表示与第1条序列的氨基酸相同。 |

|
| 图2 2003-2014年乙型Yamagata系病毒 Fig.2 HA gene of influenza B Yamagata virus mutation sites,2003-2014 |
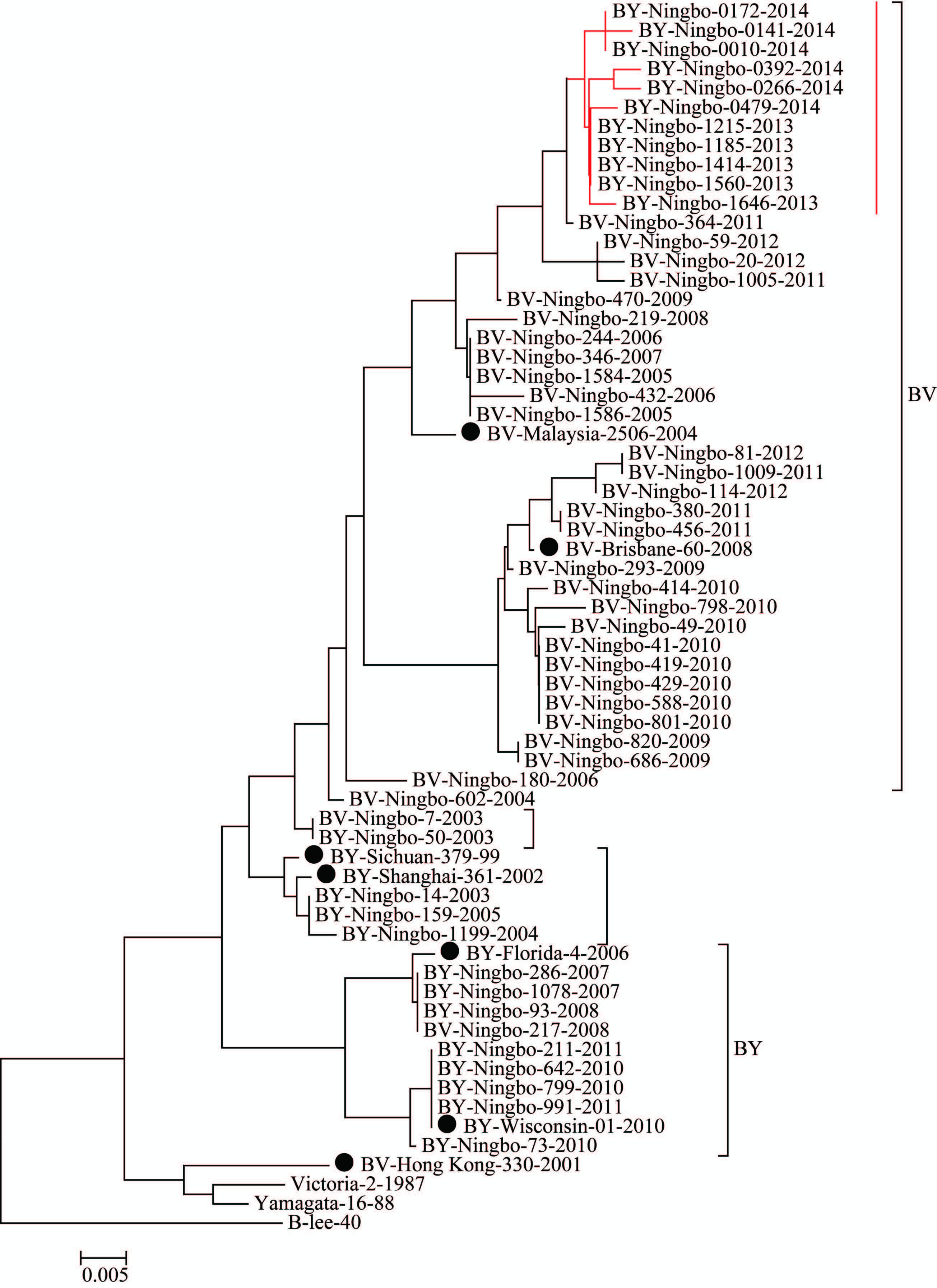
HA基因系统进化树分析结果,宁波市2003 2014年间,两大普系的乙型流感毒株各自形成3个分支。Victoria系毒株形成2003 2004年、2005 2009年、2009 2012年3个分支。其中2005 2009年流行株与WHO推荐的疫苗株B/Malaysia/2506/2004在同一分支上,2009 2012年的流行株与疫苗株B/Brisbane/60/2008于同一分支上。Yamagata系毒株形成2003 2006年、2007 2009年、2010 2014年3个分支。同样2007 2009年流行株位于B/Florida/4/2006疫苗株分支下,2010 2014年流行株位于B/Wisconsin/01/2010分支,见图3。

|
| 图3 2003-2014年宁波市乙型流血凝素基因进化树 Fig.3 Phylogenetic tree of HA gene of influenza B virus isolated in Ningbo,2003-2014 注:●为疫苗株。 |
结果与HA基因的变异在时间上相一致。Victoria系毒株的NA基因在2005年和2009年发生2次明显的变异,Yamagata系毒株的NA基因2007、2010和2013年出现3次明显的变异。其中Victoria系2005年较往年新增了5个氨基酸位点的变异,2009年至少有10个氨基酸位点的变异。Yamagata系毒株2007年出现了10个氨基酸位点的变异,2010年又有6个氨基酸位点的变异,但2013年和2014年Yamagata系毒株的NA基因与前年相比突然出现了至少20个氨基酸位点的巨大差异,而其HA基因与前年相比则只有2个氨基酸位点的差异。经NA基因的系统进化树分析结果发现,2013年和2014年 Yamagata系流行株的NA基因不在Yamagata系的进化树的主干下,而是在以疫苗株B/Malaysia/2506/2004为代表的Victoria系流行株进化分支下。与宁波市Victoria系流行株B/NingBo/364/2011的NA基因仅1个氨基酸位点的差异。即2013年和2014年Yamagata系流行株的NA基因完全替换成了Victoria系的NA基因,见图4、5。

|
| 图4 2003-2014年乙型流感NA序列变异位点(第121~466位) Fig.4 Mutation loci of influenza B virus NA sequence 2003-2004 (121~466) 注:“.”表示与第1条序列的氨基酸相同。 |
|
| 图5 2003-2014年宁波市乙型流感NA基因进化树 Fig.5 Phylogenetic tree of NA gene of influenza B virus isolated in Ningbo, 2003-2014 注:●为疫苗株。 |
乙型流感病毒也同甲型流感病毒一样,是通过基因的不断变异来改变其抗原性,从而逃避宿主特异性的免疫识别和清除,不断地在人群中造成流行[2]。2003 2014年宁波市流感监测结果显示,虽然该市流感流行的优势株以甲型为主,但是乙型流感几乎每年都会被检出,在各型别中占14.01%[3]。且Victoria系和Yamagata系两大谱系的毒株交替有时甚至同时出现,也经常发生局部的小规模暴发流行。基因分析发现,2003 2014年间乙型流感两大谱系的HA及NA基因每年均会出现一些氨基酸序列的改变,其中发生明显变异的年份依次为2005年的Victoria系、2007年的Yamagata系、2009年的Victoria系和2010年的Yamagata系变异。几乎是 两种谱系的流行株 交叉地每隔3~4年发生一次明显变异,取代另一谱系成为优势株。并且几乎每当两大谱系流行株的HA基因发生明显变异时NA基因也同时出现变异,有时NA基因的变异 速度甚至超过了HA基因[4]。
在氨基酸变异位点分析中,2003 2014年宁波市所分离到的乙型流感的Victoria系毒株在HA基因上有16个氨基酸位点发生过替换,Yamagata系毒株的HA基因上有22个氨基酸位点发生过替换。而在NA基因上至少有39个氨基酸位点发生过变异,显示NA基因比HA基因更易发生变异。而且NA基因序列的变异对流感病毒的抗原性产生重大影响。在本研究中,Yamagata系毒株在2013 2014年的流行株HA基因没有发生明显的变异,仅有2个位点的氨基酸位点的替换,但NA基因与往年的Yamagata系毒株相比发生了至少20个以上位点的差异。表明HA基因仍是往年流行的Yamagata系近似株,但NA基因与往年的Yamagata系流行株相比已经出现了明显差异。经系统进化树分析结果2013 2014年乙型流行株的HA基因在Yamagata系主干中而NA基因在Victoria系主干中,提示为Yamagata系的HA基因与Victoria系的NA发生了重配,可能是2013年底至2014年引起乙型流感高发的主要原因。
乙型流感病毒发生基因片段重配是病毒变异进化的主要机制之一[5]。宁波市2013年底至2014年初所分离到的Yamagata系HA基因与Victoria系NA基因重配株,可见由于基因重配导致的抗原转变,使其与疫苗株不相匹配,因而造成人群缺乏相应的免疫保护屏障,引起新的流行优势。该重配株是否具有更强的选择优势,还需要进一步的研究分析。乙型流感Victoria系和Yamagata系进化特点各不相同,但它们具有在人群中共存、共同进化的特征,明显增加了两者发生基因重配的机会[6]。因此在流感病毒抗原性分析中不但要加强对HA基因的检测分析,对NA基因的检测对了解流感抗原变异具有非常重要的意义。
| [1] | Rota PA, Wallis TR, Harmon MW, et al. Cocirculation of two distinct evolutionary lineages of influenza type B virus since 1983[J]. Virology, 1990,175(1):59-68. |
| [2] | Nerome R, Hiromoto Y, Sugita S, et al. Evolutionary characteristics of influenza B virus since its first isolation in 1940: dynamic circulation of deletion and insertion mechanism[J]. Arch Virol, 1998,143(8):1569-1583. |
| [3] | Hu FJ, Liu JY, Ni HX, et al. Surveillance of influenza B viruses and sequence analysis of HA1 in Ningbo city,2003-2009[J]. Disease Surveillance, 2010,25(1):33-37. (in Chinese) 胡逢蛟,刘健毅,倪红霞,等. 2003-2009年宁波市乙型流感病毒监测与HA1序列分析[J]. 疾病监测,2010,25(1):33-37. |
| [4] | Hu FJ, Li YD, Jiao SL, et al. Genetic variation of the hemagglutinin and neuraminidase of influenza B viruses isolated in Ningbo during 2010-2012[J]. Chinese Journal of Preventive Medicine, 2013,47(12):1100-1104. (in Chinese) 胡逢蛟,李永东,焦素黎,等. 2010-2012年宁波市乙型流行性感冒病毒血凝素及神经氨酸酶基因差异分析[J]. 中华预防医学杂志,2013,47(12):1100-1104. |
| [5] | McCullers JA, Wang GC, He S, et al. Reassortment and insertion-deletion are strategies for the evolution of influenza B viruses in nature[J]. J Virol, 1999,73(9):7343-7348. |
| [6] | Mao HY, Zhou M, Zhang YJ, et al. Genetic variation of the hemagglutinin and neuraminidase of influenza B viruses isolated in Zhejiang province during 1999-2010[J]. Chinese Journal of Epidemiology, 2011,32(4):376-381. (in Chinese) 茅海燕,周敏,张严峻,等. 浙江省1999-2010年乙型流感病毒血凝素和神经氨酸酶基因变化特征分析[J]. 中华流行病学杂志,2011,32(4):376-381. |
 2015, Vol. 30
2015, Vol. 30


