扩展功能
文章信息
- 朱端昊, 冯长华, 路亮, 贺凤兰, 魏芳芳, 张健, 倪贤生
- ZHU Duan-hao, FENG Chang-hua, LU Liang, HE Feng-lan, WEI Fang-fang, ZHANG Jian, NI Xian-sheng
- 人类免疫缺陷病毒HIV-1基因分型方法的初步构建
- Preliminary establishment of HIV-1 subtyping assay
- 疾病监测, 2015, 30(9): 789-791
- Disease Surveillance, 2015, 30(9): 789-791
- 10.3784/j.issn.1003-9961.2015.09.021
-
文章历史
- 收稿日期:2015-01-23
自从1985年首次报道了人类免疫缺陷病毒(HIV)的完整核酸序列以来[1],研究人员获得了大量的HIV核酸序列信息,使HIV的分子流行病学研究迅速得到发展。根据各核酸序列的分子系统发育(molecular phylogenetics)分析,在全球范围内普遍流行的HIV-1型可以分为M(Main)、 N(Non-M-Non-O)、 O(Outlier)和P 4个组(group),其中最主要的M组可以分为A、 B、 C、 D、 F、 G、 H、 J和K亚型(subtype)[2]。该分类系统并不是静态的,近年来还报道了许多广泛流行的重组亚型(circulating recombinant forms,CRFs)。了解一个地区的HIV-1基因亚型分布可以更好地预测该地区病毒的进化和传播趋势。本研究旨在为我国HIV分子流行病学研究人员提出一种合理、详尽、可行的HIV-1基因分型方法。
1 材料和方法 1.1 引物的设计在Los Alamos国家实验室的HIV核酸序列数据库中查找参考HIV核酸序列,在保守序列部分设计扩增的引物,并且确保扩增的序列位于Env基因的C2-V3区和Gag-Pol区。本实验设计的反转录-聚合酶链反应(RT-PCR)引物和nested PCR引物见表1。
| Gag区引物 | Env区引物 | |
| RT-PCR | GagF1:AAG GGG AAG TGA CAT AGC AGG(HXB2:1482-1503) GagF2:ACT TTT GGG CCA TCC ATT CC(HXB2:2592-2611) | EnvF1:TTG TAC CCC AGC TGG TTT TGC G(HXB2:6875-6896) EnvR1:GAC TCT TGC CTG GAG CTG TTT G(HXB2:7943-7964) |
| nested PCR | GagF2:AGC ATT CTG GAC ATA AGA CAA GG(HXB2:1630-1652) GagR2:CTT CCA ATT ATG TTG ACA GGT G(HXB2:2491-2512) | EnvF2:CAA TGC ACA CAT GGA ATC AAG CC(HXB2:6960-6982) EnvR2:TGG CCT GTA CCG TCA GCG TTA TT(HXB2:7885-7907) |
在南昌市男性性接触人群(MSM)监测哨点内,确证为HIV阳性的患者中选取3个样本,登记其基本信息及流行病学资料。抽取患者的抗凝静脉血5 ml,离心获得血浆-80 ℃保存备用。
1.3 血浆病毒载量的测定取1 ml 的血浆用Cobas AmpliPrep/Cobas TaqMan仪器(Roche)测量HIV病毒载量。操作步骤按仪器和试剂的说明书进行。
1.4 提取核酸并进行RT-PCR将300 μl的血浆采用Maxwell 16 viral total nucleic acid purification kit (Promega)提取HIV RNA,提取的病毒RNA采用TaKaRa公司的One-step RT PCR kit进行RT-PCR扩增。取5 μl的RNA按照试剂盒的反应说明书进行RT-PCR,其中扩增Gag区时样本的退火温度设为55 ℃,扩增Env区时样本的退火温度设为56 ℃。扩增产物直接用双蒸水稀释100倍保存于4 ℃冰箱待下一步巢式PCR用。
1.5 巢式PCR取10 μl 100倍稀释的扩增产物按照TaKaRa公司的Ex Taq酶的反应说明书配置50 μl反应体系,按94 ℃ 30 s、55 ℃(Gag)或56 ℃(Env)30 s、72 ℃ 1 min设置反应程序,共进行30次循环。取3 μl PCR产物用2%琼脂糖凝胶进行电泳,105 V,20 min。
1.6 测序及基因分型将PCR扩增之后的电泳呈明显单一条带的核酸送交Invitrogen公司进行测序,测序引物与nested PCR引物相同。获得的序列用Mega 6软件进行序列编辑、比对及遗传距离计算,构建出Env和Gag基因片段的分子进化树,通过其判断该序列的基因亚型。
2 结果 2.1 血浆HIV病毒载量3例患者的流行病学资料和HIV病毒载量见表2。3例患者都是通过男男高危性行为感染HIV,均接受过抗病毒治疗,其中MSM-1和MSM-3体内只有微量病毒(小于仪器的最低检测浓度20 copies/ml)。
| 编号 | 年龄 (岁) | 感染时间 (月) | 是否接受过 抗病毒治疗 | 病毒载量 (copies/ml) |
| MSM-1 | 23 | 21 | 是 | <20 |
| MSM-2 | 37 | 38 | 是 | 43.9 |
| MSM-3 | 36 | 90 | 是 | <20 |
提取样本核酸后进行RT-PCR,获得cDNA片段,将其直接稀释后再进行巢式PCR,所获得的目的片段进行琼脂糖凝胶电泳后所得HIV的Gag片段与Env片段见图1。
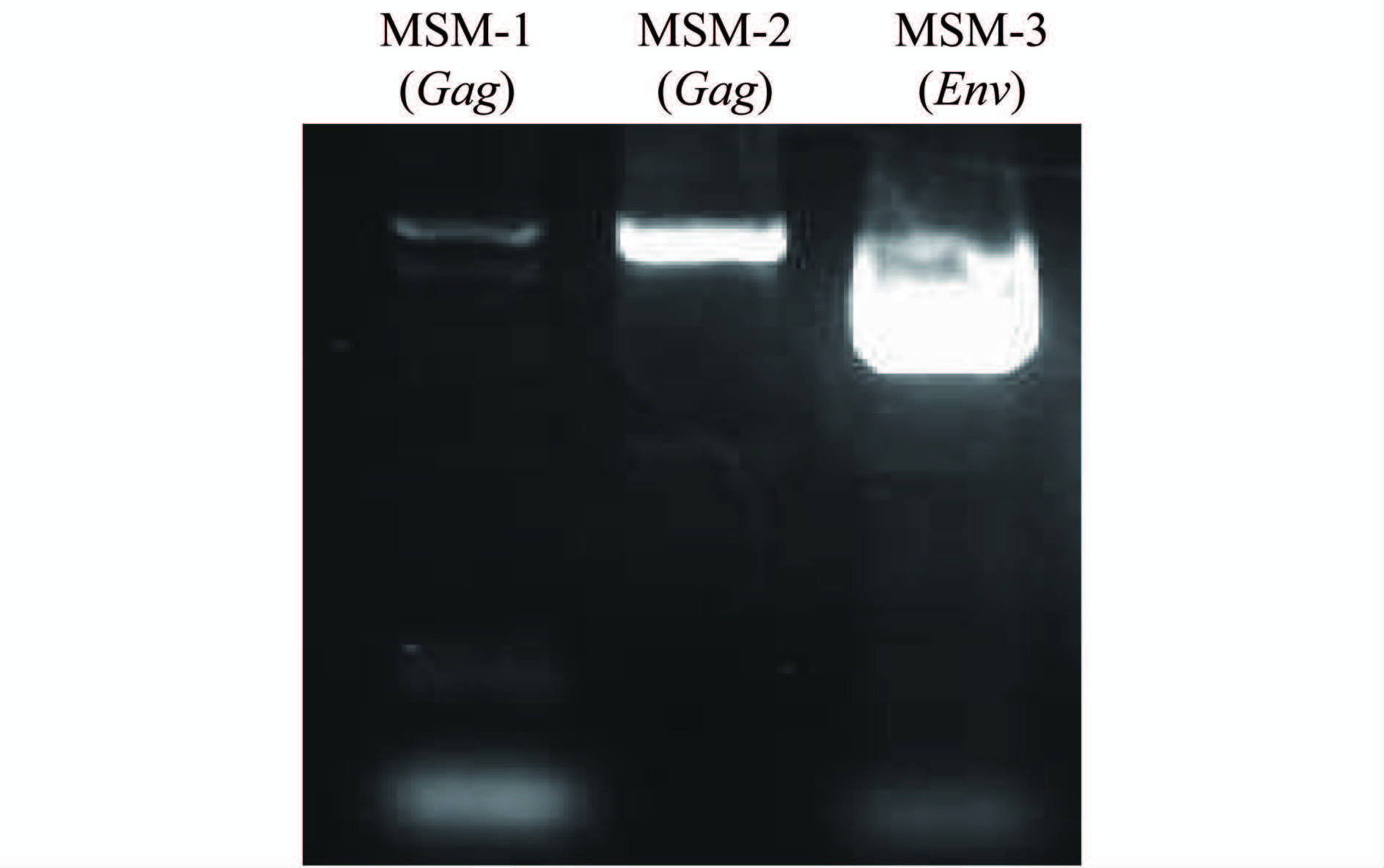
|
| 图1 MSM-1和MSM-2的Gag基因片段、MSM-3的 Env基因片段的电泳结果 Fig.1 Electrophoresis of Gag and Env region of MSM-1 and MSM-2 |
将上述扩增获得的基因片段进行测序,取得的核酸序列用Mega软件与Los Alamos实验室的HIV分型参考核酸序列进行剪接、比对[3],分别以O组HIV(GenBank序列号为AY 169804)和N组HIV(GenBank序列号为GQ 324958)为外部组(outgroup),采用最大似然性[JP2]方法生成分子进化树,见图2。根据图中的分类单元显示,MSM-1、MSM-2、MSM-3三个样本的HIV-1亚型均为CRF01_AE型。
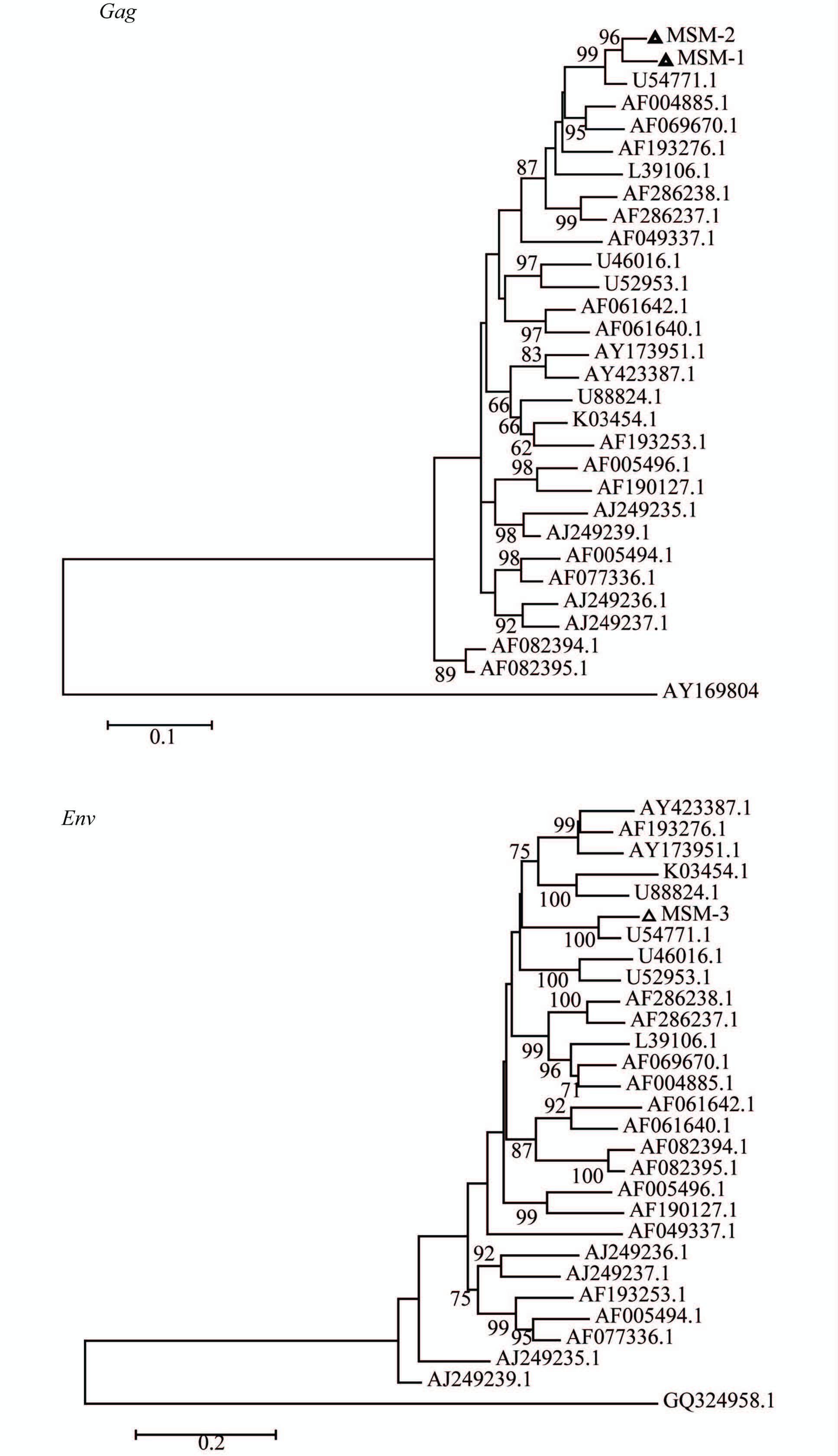
|
| 图2 以Gag和Env片段构建的分子系统发育树 Fig.2 Molecular phylogenetic trees of Gag and Env gene 注:采用最大似然性方法构建,节点上的数字表示600次bootstrap检验的百分值,仅显示大于60%的,研究样本以“△”标示。 |
HIV-1的Env基因的高度变异区V3环(V3 loop)是一个主要的病毒嗜性及抗体中和决定簇[4, 5]。笔者将MSM-3与B、01_AE亚型序列的V3环进行氨基酸比对(图3),发现其更接近于01_AE亚型,两者之间没有缺口(gaps),氨基酸匹配度为83%,相似度高达97%(相似度另外包括功能相似的氨基酸)。虽然V3环的顶端特征四肽G-P-G-R中,该样本的最后一个氨基酸由R(精氨酸)变异为Q(谷氨酰胺),但两者的功能是相似的。

|
| 图3 MSM-3 Env区V3环氨基酸比对 Fig.3 Alignment of the amino acids in v3 loop of MSM-3 Env region 注:括号里标注的是亚型,序列中的短横线代表缺口(gaps),特征四肽已用矩形框住。 |
目前我国各地的HIV分子流行病学的研究已有很多,但是大多数没有详细、可操作的完整实验流程,本研究可以为国内同行提供方法学上的一个详尽、有益的参考。在优化HIV基因分型方法中,最关键的是引物设计。目前一般是对HIV Env基因的C2-V3区进行测序,或者是Gag-Pol区。本研究将不同基因亚型的HIV基因进行比对,选定Env基因和Gag-Pol区中的保守部分作为设计引物的位点,从而增加对不同基因亚型HIV-1的扩增效率。本研究中的MSM-1和MSM-3样本的病毒载量均<20 copies/ml,但依然扩增出来明显的单一条带,说明该引物对于血浆中的HIV基因扩增灵敏度比较高。不同于以往实验中用2个基因片段来确定HIV-1亚型,本研究仅用1个基因片段,但有文献报道采用了GP41区1个小的基因片段(460 bp)依然可以获得很好的分型结果[6],笔者扩增出来的片段分别为879 bp(Gag)和882 bp (Env),而且本实验获得的分子进化树中样本与CRF01_AE有高相似度。本实验的样本来自江西省南昌市的MSM人群,在2012年的一项全国HIV-1分子流行病学调查中[7],江西省的CRF01_AE占绝大多数,与本次实验的结果相符。
图1中可见3个样本均扩增出明显的单一条带,而MSM-3的Env片段浓度更高,表明Env区段引物的特异性更好,而MSM-1和MSM-2的Gag片段的浓度对于成功测序也是足够的。本实验中MSM-3 Env基因区的V3环非常接近于CRF01_AE亚型的氨基酸序列。V3环的特征四肽G-P-G-R是抗体结合的关键位点,而MSM-3的特征四肽与CRF01_AE亚型在最后一个氨基酸位点虽然发生了变异,但两个氨基酸的功能类似,这些数据可以为进一步的结构学奠定基础。
本研究设计了一组有效的HIV-1基因分型引物,提供了一个合理、详尽的HIV-1基因分型操作步骤。
| [1] | Wain-Hobson S, Sonigo P, Danos O, et al. Nucleotide sequence of the AIDS virus, LAV[J]. Cell,1985,40(1):9-17. |
| [2] | Robertson DL, Anderson JP, Bradac JA, et al. HIV-1 nomenclature proposal[J]. Science,04/2000,288(5463):55-56. |
| [3] | Leitner T, Korber B, Daniels M, et al. HIV-1 subtype and circulating recombinant form (CRF) reference sequences[M]//HIV Sequence Compendium 2005. Los Alamos, NM: Theoretical Biology and Biophysics Group, Los Alamos National Laboratory,2005:41-47, http://www.hiv.lanl.gov. |
| [4] | Milich L, Margolin B, Swanstrom R. V3 loop of the human immunodeficiency virus type 1 Env protein: interpreting sequence variability[J]. J Virol,1993,67(9):5623-5634. |
| [5] | Zhang S, Xu GZ, Hu FJ, et al. Study on immunological status and viral subtype distribution of HIV-1 carriers in MSM, Ningbo[J]. Chinese Preventive Medicine,2010,11(4):354-357. (in Chinese) 张姝,许国章,胡逢蛟,等.宁波市男男同性恋人群中HIV-1感染者免疫状况和亚型分布研究[J]. 中华预防医学杂志,2010,11(4):354-357. |
| [6] | Yang CF,Pieniazek D, Owen SM, et al. Detection of phylogenetically diverse human immunodeficiency virus type 1 groups M and O from plasma by using highly sensitive and specific generic primers[J]. J Clin Microbiol,1999, 37(8):2581-2586. |
| [7] | He X, Xing H, Ruan YH, et al. A comprehensive mapping of HIV-1 genotypes in various risk groups and regions across china based on a nationwide molecular epidemiologic survey[J]. PLoS One,2012,7(10):e47289. |
 2015, Vol. 30
2015, Vol. 30


