扩展功能
文章信息
- 张业武, 郭青, 张春曦, 王晓风, 于萌, 苏雪梅
- ZHANG Ye-wu, GUO Qing, ZHANG Chun-xi, WANG Xiao-feng, YU Meng, SU Xue-mei
- 概率数据匹配方法在传染病报告信息匹配中的应用
- Application of probabilistic record linkage method in communicable disease reporting information matching
- 疾病监测, 2015, 30(9): 792-795
- Disease Surveillance, 2015, 30(9): 792-795
- 10.3784/j.issn.1003-9961.2015.09.022
-
文章历史
- 收稿日期:2014-08-26
不同来源数据的匹配是流行病学调查或疾病监测数据管理中比较常见的难题[1, 2, 3]。在传染病报告质量现场调查过程中,调查人员需要随机选择并抄录临床相关诊断文档或原始登记资料,如从门诊登记、住院登记、临床检验和影像检查登记等,然后将原始登记信息与传染病报告系统中现有的记录进行匹配,以发现实际报告或漏报病例。但在传染病报告实际工作过程的多个环节中,如原始登记、数据录入、审核等过程中存在着各种因素导致最终报告结果与原始记录信息之间存在一定偏差。在既往调查的资料核对过程中,往往采用人工匹配方法,工作量巨大,费时费力,或利用计算机采用传统匹配方法,导致匹配度过低。为解决这一问题,本研究采用分层多维度基于概率数据匹配方法,对调查的传染病报告卡与系统中报告进行匹配。
1 材料与方法 1.1 基本原理和方法数据匹配的基本思想是将传染病报告质量调查过程中获取的传染病病例信息,按照报告单位进行分层,逐一与大疫情系统中报告的数据进行匹配,分别计算每一配对记录之间相似性得分,若匹配相似性得分超过一定阈值(cut-off值)后,即认为匹配成功。匹配过程主要包括以下步骤:(1)对原始数据(调查数据和疫情报告数据)进行整理和规范,保证数据项名称和数据格式的统一;(2)确定比对的字段或数据项;(3)分别计算数据项和记录的总相似度;(4)选择一个合适阈值;(5)对匹配结果进行编码标记;(6)人工对计算机匹配结果进行查验,根据实际情况,对自动匹配结果的编码进行修改,形成最终匹配结果。
1.2 概率匹配赋值原则采用Fellegi-Sunter等介绍的概率数据匹配方法[1, 4],对不同数据项或字段采用不同的赋值,这种方法基本思想是记录的数据项或字段值取值范围差异越大,某个特定值出现概率越低,那么不同记录间一旦匹配成功则得分越高。举例来说,对于“姓名”和“性别”两个字段,一旦姓名匹配成功,则它的得分应远远高于性别匹配成功后的得分。为减少应用的复杂性,采用改良的Fellegi-Sunter方法[4],按照如下原则对不同数据项或字段间匹配得分直接进行赋值:
(1)对于两个记录中数据项,若任何一个记录该数据项数据缺失,则配对得分为0。
(2)若两个记录中相同数据项一致,根据一致性程度大小,给予正得分。对于不同数据项最高分取值,采取差异化原则,即数据项的取值个数越多或取值范围差异越大,该数据项得分就越高,如姓名的最高分大都高于性别;同一数据项如姓名、地址等若存在部分匹配,根据其不同属性,采用不同的函数计算其相似度,并给予相应的得分。
(3)如果两个记录中同一数据项若完全不同,则给予负得分。负得分的高低主要依据数据的可靠程度来确定,如果某个数据项的可靠程度高,那么负得分就越低。如“性别”的可靠性往往高于“职业”,若两个记录的“性别”数据项不等,则扣分多,而若两个记录的“职业”数据项不等,则扣分应该相对低。
1.3 传染病报告卡匹配项目的赋值方法和结果待匹配数据来自2014年4月在全国9省市内开展的传染病报告质量调查。本次调查过程中抽样采集了2013年度(2013年1月1日至12月31日)门诊、住院、处方登记信息。为将原始登记信息与大疫情报告数据进行匹配,选择了姓名、性别、年龄、职业、县区地址编码、详细地址、疾病诊断、发病日期和诊断日期,作为匹配项,进行多维度的匹配。采用Fellegi-Sunter改良方法对不同项目匹配得分按照如下方法进行赋值,见表1。
| 数据项 | 完全匹配得分 | 部分匹配得分 | 部分匹配举例 | 完全不匹配得分 |
| 姓名 | 20 | 两个字匹配10一个字匹配-3 | 两个字匹配如:“李则平”与“李钊平”、“王玉芬”与“王玉芳”;一个字匹配如:“肖玉”与“尚玉”,“徐耀”与“徐熠” | -5 |
| 性别 | 2 | - | -2 | |
| 年龄 | 相差2岁范围内得3分 | 3/ | 如27岁与25 岁匹配得分为0.7543岁与56岁匹配得分为0.2 | 0 |
| 职业 | 1 | - | 0 | |
| 地址编码 | 2 | - | 0 | |
| 详细地址 | 2 | 2*(1-complev(strip(ADDRESS_1), strip(ADDRESS_2)) /max(length(strip(ADDRESS_1)), length(strip(ADDRESS_2)))) | 如“公正乡枯娄那琴屯”与“公正乡枯娄村那琴屯”匹配得分为1.78“大水坑张布掌”与“大水坑镇张布良村”匹配得分为1.25 | 0 |
| 疾病诊断编码 | 10 | - | -5 | |
| 发病日期 | 8 | 相差两个月以内则得5分,否则为2/int(abs(onset_1-onset_2)/30) | 如2013/3/17与2013/1/26匹配得分为52013/1/10与2013/3/29匹配得分为1 | - |
| 诊断日期 | 8 | 相差两个月以内则得5分,否则为2/int(abs(diagnosis_1-diagnosis_2)/30) | 同上 | - |
对于详细地址数据项,采用Levenshtein编辑距离(Levenshtein edit distance)计算方法,即计算两个字串之间,由一个转成另一个所需的最少编辑操作次数,来衡量两个字符串之间的相似程度[5]。为保证不同配对间的可比性,将编辑距离除以最长字符串的长度获得相对编辑距离。
1.4 运算过程本研究算法过程简要如下:
(1) 准备好匹配(电子报告卡)和待匹配(原始登记)数据集。数据集包含姓名、性别、年龄、职业、所在县区编码、家庭住址、疾病名称、发病日期、诊断日期等变量,两个数据集的变量名称和格式一致。
(2) 将原始数据集按机构进行分层。
(3) 分别从电子报告卡数据集和原始登记数据集中选择第一家机构的数据,分别为该机构的电子报告卡数据集和原始登记数据集。
(4) 将不同来源的记录按笛卡尔乘积方式进行逐一配对。即电子报告卡数据集第1条记录,与原始登记数据集所有记录进行匹配,然后第2条记录与原始登记数据集所有记录进行匹配,直至所有记录完成匹配。假设电子报告卡有m条记录,原始登记有n条记录,最终生成的匹配数据集有m×n条数据记录。
(5) 按表1介绍的赋值方法,计算每条匹配记录中各匹配项目分项得分,以及各分项目之和,即总得分。
(6) 根据原始登记数据集生成最终匹配结果数据集。按笛卡尔乘积方式,每条原始登记记录,可与m条电子报告卡逐一匹配生成m条匹配。每个匹配在步骤5中都有一个得分,选择得分最高的那条匹配结果保留在最终匹配结果数据集中。这样每个机构最终匹配结果数据集记录数与原始登记数据集记录数相同。
(7) 继续从两个原始数据集中,选择第二家机构数据,重复步骤4、5、6。如此重复,直至完成所有机构原始记录的匹配计算和选择。
(8) 根据cut-off值对匹配结果进行分类,并与人工结果进行比较。
1.5 结果评估为验证模糊配对算法的有效性,将机器配对结果进行人工审核,将最终人工审核结果作为金标准,计算模糊配对算法的灵敏度、特异度和总一致率等相关指标;为选择最佳阈值,连续选择不同的阈值分别对机器配对总得分结果进行分类,配对总得分大于等于某个阈值时,认为机器配对成功,而小于该阈值则认为不匹配。随后将分类结果与人工配对结果进行比较,计算灵敏度、特异度和约登指数,选择约登指数最大情况下的阈值作为最终阈值,评价机器匹配效果。将最终配对结果与配对得分进行比较,绘制相应的受试者工作特征曲线(receiver operating characteristic curve,ROC)。所有的统计分析工作均在SAS软件中完成。
2 结果 2.1 基本情况2014年传染病报告质量调查过程中,共采集2013年度9个省市115所医疗机构2153条门诊、住院和处方原始记录,这些机构2013年共报告97 271张传染病报告卡。根据机构进行分层按笛卡尔乘积进行配对,计算每个配对得分,按照得分高低进行降序排列,原始记录作为主记录,电子报告卡数据作为从记录,将与主记录对应配对得分最高的第一条从记录保留在配对记录中,作为人工配对审核依据。配对总得分和分项得分情况见表2。
| 变量 | n | 均值 | 最小值 | 最大值 | 标准差 |
| 总得分 | 2153 | 42.32 | -2.25 | 52.00 | 11.44 |
| 姓名 | 2153 | 16.50 | -5.00 | 20.00 | 7.35 |
| 性别 | 2153 | 1.91 | -2.00 | 2.00 | 0.58 |
| 年龄 | 2153 | 2.42 | 0.00 | 3.00 | 1.11 |
| 职业 | 2153 | 0.55 | 0.00 | 1.00 | 0.50 |
| 所在县区 | 2153 | 1.58 | 0.00 | 2.00 | 0.81 |
| 家庭住址 | 2153 | 0.83 | 0.00 | 2.00 | 0.69 |
| 疾病名称 | 2153 | 9.31 | -5.00 | 10.00 | 3.08 |
| 发病日期 | 2153 | 3.99 | 0.00 | 6.00 | 2.63 |
| 诊断日期 | 2153 | 5.46 | 0.00 | 6.00 | 1.20 |
最终以25分作为阈值,即配对总得分≥25分,认为机器配对成功,而<25分则认为不匹配。在约登指数最大情况下,机器配对与人工配对结果进行一致性分析结果见表3。
| 机器匹配 | 人工审核 | 总计 | |
| 已报告 | 未上报 | ||
| 总分≥25分 | 1897 | 12 | 1909 |
| 总分<25分 | 20 | 234 | 244 |
| 合计 | 1917 | 236 | 2153 |
计算结果显示,机器自动匹配的灵敏度为98.96%(1987/1917),95%置信区间为(98.39%~99.36%);特异度为94.92%(234/236),95%置信区间为(91.29%~97.35%)。总一致率=98.51%[(1897+234)/2153],95%置信区间为(97.91%~98.98%)。Kappa值为0.9250,95%置信区间为(0.8992~0.9507)。
2.3 总得分的ROC以人工审核后匹配结果作为金标准,绘制匹配总得分的ROC,并计算ROC下面积(Area under the curve,AUC),见图1。
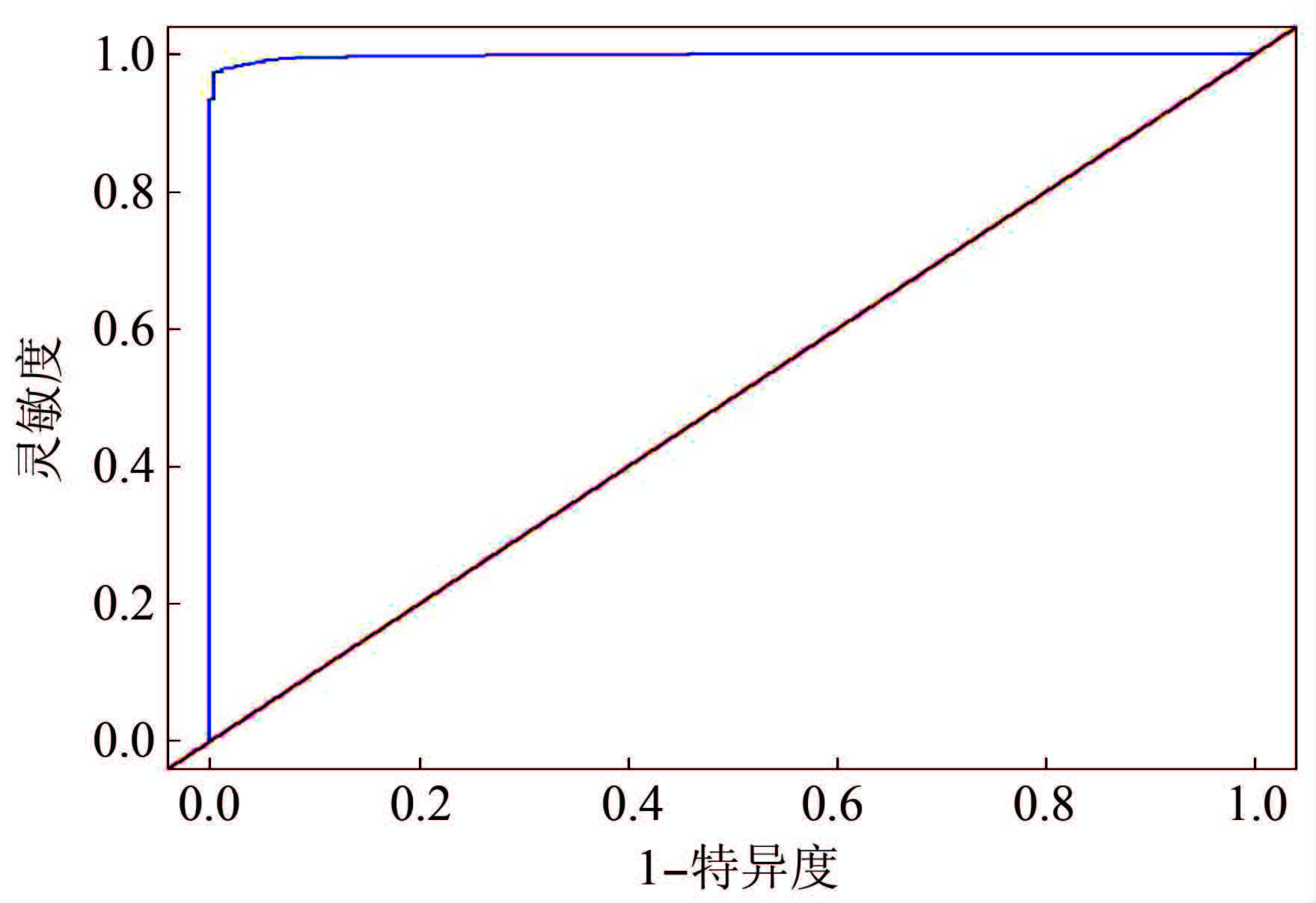
|
| 图1 匹配总得分的ROC Fig.1 ROC curve for overall scores |
计算结果显示,AUC为0.9979。AUC可用于检查指标的判别能力。一般来说,在AUC>0.5的情况下,AUC越接近于1,说明指标判别效果越好。AUC在 0.5~0.7时有较低准确性,AUC在0.7~0.9时有一定准确性,AUC>0.9时有较高准确性。因此,采用多维度概率模糊匹配方法获得的指标在判断不同来源传染病报告记录是否匹配具有较高的准确性。
3 讨论本研究利用信息匹配原理,采用改良的Fellegi-Sunter简易赋值方法[4],对不同来源信息的传染病登记和报告信息进行匹配。匹配结果评价表明,该方法具有较高的灵敏度、特异度和总一致率。在既往的传染病报告质量调查过程中,如何对不同来源的记录信息进行匹配是项挑战性工作。对于同一传染病病例,不同来源原始信息和最终报告信息,存在一定程度的不一致性现象,如信息空缺、错误和不完整等。常见的情况包括姓名拼写错误、疾病诊断名称不规范、日期(包括出生日期、诊断时间、发病时间)偏差和地址信息填写不完整等。在人工匹配过程中,往往采取少量关键信息进行判断,很难做到对多个非关键信息或对部分匹配信息的利用,从而导致不同人员之间匹配结果存在较大差异。
Fellegi-Sunter方法综合利用记录中的多个属性信息进行匹配,并基于匹配项取值概率,给出了不同匹配项匹配得分的计算方法(表1)。该方法假设各匹配项取值概率在记录匹配的条件下相互独立。但既往的研究发现,即使记录的匹配项不满足条件独立的假设要求,该模型判断结果的准确性仍保持较高[1]。另外,笔者在构建不同数据匹配项权重系数时,并没有严格按照Fellegi-Sunter建议的方法来计算各数据匹配项的权重系数,仅保证实际权重系数大小排列与理论推导结果在方向保持一致,按照经验确定适当的规则进行赋值,也获得很高的一致性,同时说明了这种方法具有很强的稳健性。
本研究采用较为简易的数据匹配项赋值方法,并利用SAS软件编写了相应的算法宏程序,成功解决了现场调查的原始数据与网络报告系统的数据匹配问题,匹配结果与实际情况具有较高的一致性,显著提高了工作效率,也为今后开展类似工作提供简易的分析工具。
| [1] | Fellegi IP, Sunter AB. A theory for record linkage[J]. J Am Stat Assoc,1969,64(328):1183-1210. |
| [2] | Harron K, Wade A, Gilbert R, et al. Evaluating bias due to data linkage error in electronic healthcare records[J]. BMC Med Res Methodol,2014,14:36. |
| [3] | Méray N, Reitsma JB, Ravelli ACJ, et al. Probabilistic record linkage is a valid and transparent tool to combine databases without a patient identification number[J]. J Clin Epidemiol,2007,60(9):883-891. |
| [4] | Glenn Wright MPA, Permanente K, Oakland CA. Probabilistic record linkage in sas®[M/OL]. (2011-10-12)[2014-06-21]. http://www.wuss.org/proceedings11/Papers_Wright_G_76128.pdf. |
| [5] | Ristad ES, Yianilos PN. Learning string-edit distance[J]. IEEE Trans Pattern Anal Mach Intell,1998,20(5):522-532. |
 2015, Vol. 30
2015, Vol. 30


