扩展功能
文章信息
- 杜小莉, 周海健
- DU Xiao-li, ZHOU Hai-jian
- 肺炎克雷伯菌脉冲场凝胶电泳分型能力评价
- Evaluation of performance of pulsed field gel electrophoresis in subtyping of Klebsiella pneumoniae
- 疾病监测, 2015, 30(11): 969-975
- Disease Surveillance, 2015, 30(11): 969-975
- 10.3784/j.issn.1003-9961.2015.11.018
-
文章历史
- 收稿日期: 2015-06-19
肺炎克雷伯菌(Klebsiella pneumoniae)是临床分离及医院感染的重要致病菌之一,引起的医院感染率近年居高不下,且多耐药性菌株的不断增加常导致临床抗菌药物治疗的失败和病程迁延[1, 2]。细菌分型有助于肺炎克雷伯菌院内感染的早期发现、耐药菌株溯源和传播途径甄别[3]。近年来随着微生物基因组测序技术的飞速发展和数据的迅速积累,以核酸序列差异分析为主的分子分型技术逐步建立并且成熟,在菌株进化研究、流行病学溯源调查等方面均显示了很好的适用性。其中脉冲场凝胶电泳(pulsed-field gel electrophoresis,PFGE)技术是目前细菌性传染病流行病学调查领域被最广泛使用的一种细菌分子分型技术。多位点序列分型(multilocus sequence typing,MLST)适合用于长期、大范围的分子流行病学调查和菌群变迁研究,但由于其分辨力不高,用于暴发调查的能力有限。
在之前的研究中,笔者系统地优化和建立了肺炎克雷伯菌PFGE分型方法,确定了合适的内切酶和电泳参数[4]。本研究进一步使用更多的菌株对该方法的分型力(typeablity)、可重复性(reproducibility)、分辨力(discriminatory power)、流行病学一致性(epidemiololgic concordance)及其与MLST的分型一致性进行评价,并建立PFGE分型数据库供肺炎克雷伯菌感染性疾病的预防控制和院内感染调查使用。
1 材料与方法 1.1 实验菌株随机选取实验室保存的于20062014年从27个省(市)分离的220株肺炎克雷伯菌。所有菌株经生化鉴定为肺炎克雷伯菌。菌株包括:20132015年全国50家医院分离的碳青霉烯耐药临床分离株56株、2011年某医院在一起院内感染暴发期间分离的34株、2013年北京市分离的没有流行病学关联的腹泻患者粪便分离株37株、20102011年沈阳市没有流行病学关联的健康人体检粪便标本分离株11株、2005 2006年(期间未发现暴发)四川省某医院临床患者分离株20株、2014年(期间未发现暴发)江西省某医院临床患者分离株7株、2014年(期间未发现暴发)北京市某医院临床患者分离株7株、20132014年石家庄市食物分离株48株。
本研究使用全部220株评价PFGE方法的分型力;随机挑选18株评价PFGE方法的可重复性;分别挑选51株分离自不同医院的无直接流行病关联的碳青霉烯耐药临床分离株、37株无直接流行病关联的腹泻患者肠道分离株、48株无直接流行病关联的食品分离株评价PFGE方法的分辨力;使用已经明确的一次暴发期间分离的34株菌(具有详细的流行病学信息、耐药基因检测和MLST分型结果)评价PFGE分型的流行病学一致性;分别使用一次暴发期间分离的34株菌和无直接流行病学关联的64株菌(已有MLST分型结果)评价PFGE和MLST分型的一致性。沙门菌Braenderup血清型菌株H9812作为PFGE分子质量标准[5]。
1.2 PFGE实验方法使用本实验室建立的肺炎克雷伯菌PFGE分型方案[4],简述如下:用bioMérieux DENSIMAT比浊仪调整细菌悬液浓度,使浊度为3.8~4.0。加入1% Seakem Gold:1% SDS胶,混匀制备胶块。使用含蛋白酶K的细胞裂解液(CLB)消化2 h。纯水清洗胶块2次;TE(10 mmol/L Tris:1 mmol/L EDTA,pH值8.0)清洗胶块4次。使用45 U XbaⅠ(TaKaRa,中国大连)内切酶进行酶切,在37 ℃孵育2 h。在CHEF-DRⅢ(Bio-Rad Laboratories,美国)电泳仪中进行脉冲场电泳。电泳参数为6~36 s,18.5 h。电泳结束后,使用GelRed核酸染料(Biotium,美国)染色。在读胶仪中成像,并转换成TIFF图像格式保存。
1.3 数据分析PFGE图像录入BioNumerics(Version 5.1,Applied maths,Inc.)软件包进行处理,识别图像条带,经统一的分子质量标准进行校准,标定条带位置,必要时进行手工校正,<20.5 kb的条带忽略不计。每两个图像之间的相似性系数用Dice系数(Dice coefficients,SD)表示[6],SD=2nxy/(nx+ny),其中nx是菌株x的总条带数,ny是菌株y的总条带数,nxy是菌株x和菌株y共有的条带数。SD值反映不同菌株PFGE图像之间的相似性程度,范围在0~1之间,0代表完全不一样,1代表完全相同。出现不同条带即判定为不同的型别,对每1个型别都进行命名。根据每两个图像之间的相似性系数,用非加权配对算术平均法(unweighted pair group average method,UPGMA)进行聚类,构建聚类树。
使用Simpson差异指数(D)来评价不同分型方法的分辨力[7]。Simpson差异指数的计算公式为 D=1-{∑ [nj (nj -1)]}/ [N (N-1)],其中nj为属于第j个型别的菌株数,N为特定群体中的总菌株数。D值介于0~1之间,D值越趋于1表示该方法分辨力越好。
2 结果 2.1 分型力评价共对220株肺炎克雷伯菌进行PFGE分型,其中216株菌获得的PFGE图谱条带清晰,酶切电泳后可得到25~1000 kb的电泳片段,不同的菌株条带数不同,从16条到22条不等。其余4株菌通过酶切电泳后图谱出现降解现象,通过在电泳液中添加硫脲进行重复实验同样出现降解现象,不能满足数据分析要求,判断为不可分型菌株。故本研究中PFGE对肺炎克雷伯菌的分型力为98.18%(216/220)。
2.2 可重复性评价随机挑选18株肺炎克雷伯菌以相同的实验条件,由不同的实验人员在不同实验室的两台脉冲场凝胶电泳仪上分别进行两次实验,结果显示18株菌的两次实验均获得相同的图谱,表明PFGE方法对肺炎克雷伯菌分型具有很好的重复性(图1)。

|
| 图 1 18株肺炎克雷伯菌的PFGE 分型的可重复性分析 Fig. 1 Reproducibility of PFGE subtyping of 18 K. pneumoniae isolates |
本研究通过3组菌对PFGE的分辨力进行评价。第1组为51株分离自不同医院的无直接流行病关联的碳青霉烯耐药临床分离株,除1株菌株重复两次实验均降解外,其余50株分为43个不同的PFGE型,分型D值为0.9910(图2A、表1);第2组为37株无直接流行病关联的腹泻病人粪便分离株,除3株菌株重复两次实验均降解外,其余34株分为32个PFGE型,分型D值为0.9964(图2B、表1);第3组为48株无直接流行病关联的食品分离株,48株分为48个PFGE型,分型D值为1.0000(图2C、表1)。

|
| 图 2 注:A. 50株无直接流行病关联的碳青霉烯耐药临床株PFGE聚类结果; B. 34株无直接流行病关联的腹泻病人粪便分离株PFGE聚类结果,菌株均于2013年分离自北京市; C. 48株无直接流行病关联的食品分离株PFGE聚类结果。 Fig. 2 Discriminatory power of PFGE in subtyping of 18 K. pneumoniae isolates. |
| 测试菌株分组 | 菌株 数量 | PFGE 型别数 | 分型 D值 |
| 无直接流行病关联的碳青霉烯耐药 临床株 | 50 | 43 | 0.9910 |
| 无直接流行病关联的腹泻患者 粪便分离株 | 34 | 32 | 0.9964 |
| 无直接流行病关联的食品分离株 | 48 | 48 | 1.0000 |
挑选既往发生的一次院内感染暴发期间分离的34株肺炎克雷伯菌(均分离自患者)进行分型,结果显示34株菌分为11个不同的PFGE带型(图3)。根据TENOVER法则[8],其中21株具有相同的PFGE带型,确定为本起暴发的优势带型;6株的PFGE带型与优势带型相差1~3条带,判断为与优势带型菌株高度相关菌株;其余7株与优势带型相差条带数多于6条,判断为与优势带型菌株不相关菌株。结合流行病学信息、耐药基因检测和MLST分型资料,21株优势带型及其6株高度相关菌株均分离自病区1和病区4,携带有KPC-2基因,且MLST型别均为ST11;7株PFGE判断为不相关的菌株,均不携带KPC-2基因,MLST型别均为非ST11,其中分离自病区2的3株菌(2012TR-QT-50、2012TR-QT-51、2012TR-QT-55)又具有相同的MLST型别。在分离时间上,3株分离自病区1的不相关菌株,分离时间均在优势PFGE带型菌株出现之前。结合流行病学信息、耐药基因检测和MLST分型结果,显示PFGE分型能够很好地区分同一暴发期间分离的肺炎克雷伯菌与暴发的相关程度。

|
| 图 3 34株分离株同一起院内感染暴发事件期间的肺炎克雷伯菌PFGE分型聚类结果 Fig. 3 PFGE clustering of 34 K. pneumoniae isolates from an outbreak |
对34株一次院内感染暴发期间分离的肺炎克雷伯菌同时进行PFGE和MLST分型(图3)。结果显示,34株菌通过PFGE,分为11个PFGE型;通过MLST,分为6个MLST型。其中携带KPC-2基因的菌株,PFGE带型相同或者相似,聚成一簇;MLST分型均为ST11型。不携带KPC-2基因的菌株均不是ST11型,与携带KPC-2基因的菌株PFGE差异明显,聚类关系远。其中3株分离自病区2的不携带KPC-2基因的菌株,PFGE带型和MLST型别均相同。
对64株无直接流行病学关联的肺炎克雷伯菌同时进行PFGE和MLST分型,比较两种方法的分型一致性(图4)。结果显示,PFGE和MLST分别将64株菌株分为59个和31个型别。在聚类关系上,两种分型方法的聚类树结构相似度仅为18.1%。MLST型别相同的菌株通过PFGE图谱不一定聚在一起,如20株ST11型菌株中有19株成簇聚集,另外1株单独一个分支;ST23的5株菌株中,有4株PFGE图谱相似,聚类成簇,另外1株(JX2014K49)单独一个分支;ST37型的2株菌株(NA1715、BJYY1871)在PFGE聚类树上处于很远的两个分支。
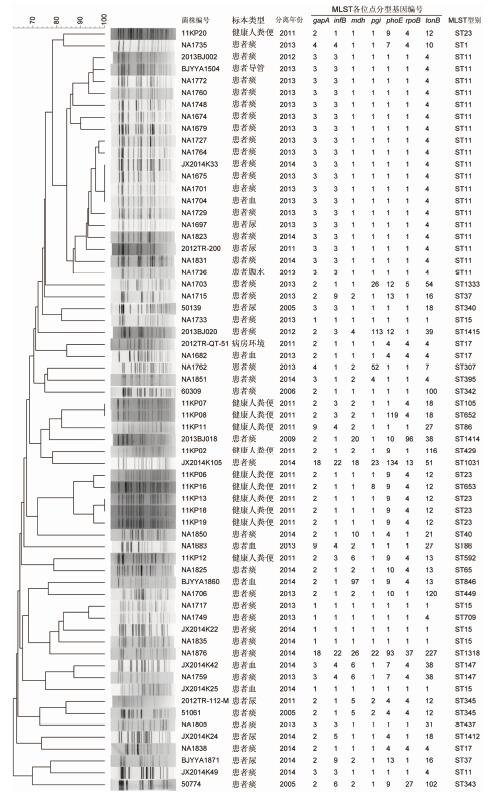
|
| 图 4 64株无直接流行病学关联的肺炎克雷伯菌PFGE分型聚类结果和MLST分型信息 Fig. 4 PFGE clustering and MLST results of 64 K. pneumoniae isolates without epidemiologic link |
在细菌分型的实际应用中,PFGE显示出比其他分型方法更强的分辨率和流行病学调查能力。而且,由于PFGE是选用全基因组DNA在统一设定内切酶和实验条件下进行的,产生的结果可重复性好,因而不同实验室的PFGE结果具有可比性,这是PFGE分型方法优于其他众多分子分型方法之处。PFGE在肺炎克雷伯菌的分型中被广泛使用,但目前没有报道对现有方法进行系统的评价,而且不同实验室使用的内切酶和电泳参数不一致,导致不同实验室间的结果不能进行比较。对分型方法分型能力的评价指标一般包括分型力、可重复性、分辨力和流行病学一致性等方面[9]。
本研究表明PFGE对肺炎克雷伯菌分型具有很好的分型力和可重复性。PFGE的分辨力主要取决于使用的内切酶和电泳参数。在之前的研究中,笔者确定了XbaⅠ为首选内切酶,电泳参数为6~36 s,18.5 h。在本研究中,PFGE对于选择的无流行病关联的碳青霉烯耐药临床菌、无流行病关联的腹泻患者肠道分离菌、无流行病关联食品分离菌3组肺炎克雷伯菌的分型D值均>0.99,表明对于这些组别菌的分型,PFGE产生Ⅰ类错误的概率小于1%。在分析一次院内感染暴发期间分离的菌株时,PFGE能够很好地区分暴发相关和不相关菌株,分型结果与菌株的病房分布、分离时间和其他实验检测的结果相符。综上所述,PFGE不但具有很好的分辨力,而且具有很好的流行病学一致性,能够识别相同暴发和不同暴发以及暴发期间分离的非相关菌株。
在实际应用中仅使用一种分型方法所得出的结论会存在一定的片面性,使用多种方法进行协同分型往往能得到更有用的结果。目前使用较多的是将PFGE和MLST协同分型。MLST数据可靠,而且有公共数据库供全球的实验室使用。在本研究中将肺炎克雷伯菌的PFGE和MLST两种分型结果进行比较时,针对一次暴发菌株的分析,两种分型方法展现了很好的分型一致性;但是针对不同来源(流行病学上无直接关联)的菌株分型时,两种方法的一致性较差,相同MLST型别的菌株在PFGE聚类树上距离远。这种不一致性可能与两种方法的分型原理不同有关。PFGE是通过检测菌株基因组之间大片段的插入、缺失、重组、酶切位点的点突变、质粒的丢失和获得等引起的差异,而MLST检测的只是管家基因的序列差异。PFGE检测了多种基因变异事件,无法确认图谱反映出来的差异是由哪种基因变异事件引起的,而不同基因变异事件的理论发生时间是不一样的,故PFGE不适合用于研究细菌的种群结构和遗传进化关系,只适合用于比较菌株之间的差异性,应用于监测、暴发甄别和溯源。在本研究中,PFGE和MLST针对暴发菌株分型的一致性和针对不同来源菌株分型的不一致性证明了这一点。MLST能够揭示细菌的种群结构,但是其分辨力低于PFGE,许多不同来源的肺炎克雷伯菌均属于相同的MLST型,而这些属于相同MLST型的菌株具有不同的PFGE型。所以不能单独用MLST分析肺炎克雷伯菌之间的流行病学关联,在暴发鉴别和溯源过程中必须在PFGE分型结果的基础上开展MLST研究。
近年,肺炎克雷伯菌引起的院内感染和传播流行在国内外都时有发生[10, 11, 12, 13]。建立分子分型公共数据库有利于不同实验室之间的数据交流,从而观察到相同地区的不同医院、不同地区流行菌株的异同以及菌株在传播流行过程中发生的分子水平的变异,从而掌握菌株的流行规律,为控制疾病传播提供实验室数据。在本研究中,笔者依据肺炎克雷伯菌相关疾病的监测需求,对PFGE用于肺炎克雷伯菌分型研究的能力进行了评价。目前国际上还没有通用的肺炎克雷伯菌分子分型数据库,国内不同实验室的数据由于使用的内切酶和电泳参数不一致,无法放到同一个数据库中进行比对和分析。本评价确定目前使用的PFGE方法对肺炎克雷伯菌具有很好的分型能力,为建立全国性的网络化的肺炎克雷伯菌分子溯源数据库奠定了方法学基础。数据库的建立将增强我国肺炎克雷伯菌感染防控能力。随着工作的开展,在数据扩充的同时,供全国各类实验室查询、比对图谱,为分析肺炎克雷伯菌相关的疾病暴发与流行原因、散发病例分离菌株之间的关系及病例菌株与环境菌株之间的关系提供实验室数据支持。
| [1] | Shon AS, Bajwa RP, Russo TA. Hypervirulent (hypermucoviscous) Klebsiella pneumoniae:a new and dangerous breed[J]. Virulence,2013,4(2):107-118. |
| [2] | Gürntke S, Kohler C, Steinmetz I, et al. Molecular epidemiology of extended-spectrum beta-lactamase (ESBL)-positive Klebsiella pneumoniae from bloodstream infections and risk factors for mortality[J]. J Infect Chemother,2014,20(12):817-819. |
| [3] | Pereira GH, Garcia DO, Mostardeiro M, et al. Outbreak of carbapenem-resistant Klebsiella pneumoniae:two-year epidemiologic follow-up in a tertiary hospital[J]. Mem Inst Oswaldo Cruz,2013,108(1):113-115. |
| [4] | Han H, Zhou H, Li H, et al. Optimization of pulse-field gel electrophoresis for subtyping of Klebsiella pneumoniae[J]. Int J Environ Res Public Health,2013,10(7):2720-2731. |
| [5] | Hunter SB, Vauterin P, Lambert-Fair MA, et al. Establishment of a universal size standard strain for use with the PulseNet standardized pulsed-field gel electrophoresis protocols:converting the national databases to the new size standard[J]. J Clin Microbiol,2005,43(3):1045-1050. |
| [6] | Dice LR. Measures of the amount of ecologic association between species[J]. Ecology,1945,26(3):297-302. |
| [7] | Hunter PR, Gaston MA. Numerical index of the discriminatory ability of typing systems:an application of Simpsons index of diversity[J]. J Clin Microbiol,1988,26(11):2465-2466. |
| [8] | Tenover FC, Arbeit RD, Goering RV, et al. Interpreting chromosomal DNA restriction patterns produced by pulsed-field gel electrophoresis:criteria for bacterial strain typing[J]. J Clin Microbiol,1995,33(9):2233-2239. |
| [9] | Wang LL, Xu JG. Application of pulsed field gel electrophoresis (PFGE) in molecular typing[J]. Disease Surveillance,2006,21(5):276-279.(in Chinese),王丽丽,徐建国.脉冲场凝胶电泳技术(PFGE)在分子分型中的应用现状[J].疾病监测,2006,21(5):276-279. |
| [10] | Li B, Xu XH, Zhao ZC, et al. High prevalence of metallo-β-lactamase among carbapenem-resistant Klebsiella pneumoniae in a teaching hospital in China[J]. Can J Microbiol,2014,60(10):691-695. |
| [11] | Sun K, Chen X, Li C, et al. Clonal dissemination of multilocus sequence type 11 Klebsiella pneumoniae carbapenemase-producing K. pneumoniae in a Chinese teaching hospital[J]. APMIS,2015,123(2):123-127. |
| [12] | Zaman UZ, Aldrees M, AL Johani SM, et al. Multi-drug carbapenem-resistant Klebsiella pneumoniae infection carrying the OXA-48 gene and showing variations in outer membrane protein 36 causing an outbreak in a tertiary care hospital in Riyadh, Saudi Arabia[J]. Int J Infect Dis,2014,28:186-192. |
| [13] | Zweigner J, Gastmeier P, Kola A, et al. A carbapenem-resistant Klebsiella pneumoniae outbreak following bronchoscopy[J]. Am J Infect Control,2014,42(8):936-937. |
 2015, Vol. 30
2015, Vol. 30


