扩展功能
文章信息
- 方斌, 刘琳琳, 李翔, 叶国军, 余晓, 宋毅
- FANG Bin, LIU Lin-lin, LI Xiang, YE Guo-jun, YU Xiao, SONG Yi
- 2010-2015年湖北省乙型流感病毒流行和进化分析
- Epidemiological and evolutionary characteristics of influenza B viruses in Hubei, 2010-2015
- 疾病监测, 2016, 31(7): 554-560
- Disease Surveillance, 2016, 31(7): 554-560
- 10.3784/j.issn.1003-9961.2016.07.006
-
文章历史
- 收稿日期:2016-02-26
乙型流感病毒和甲型流感病毒都会引起人呼吸道感染,在全球均会造成高发病率和高死亡率[1-3]。因宿主有限,乙型流感病毒重配通常只发生在同时期不同人源的乙型流感病毒间,而甲型流感病毒重配则发生在众多不同宿主的甲型流感病毒间[4-5]。目前全球流行着两种乙型流感病毒系:Victoria系(源自B/Victoria/02/87)和Yamagata系(源自B/Yamagata/16/88)[4, 6],它们在交叉流行和共同流行时期会发生系内重配和系间重配[7-8]。
聚合酶链反应(polymerase chain reaction,PCR)和血凝抑制试验(hemagglutination inhibition test,HI)是监测乙型流感病毒流行情况的主要技术,进化树分析可以揭示病毒系内或系间重配的进化情况[4]。本研究将20102015年湖北省乙型流感病毒每周流行情况与其对应血凝素(hemagglutinin,HA)和神经氨酸酶(neuraminidase,NA)蛋白进化树分布情况进行协同分析,以了解湖北省乙型流感病毒系内、系间重配的流行情况,并分析其基因特性、氨基酸突变和三维结构变化,为流感科学防控工作提供依据。
1 材料与方法 1.1 乙型流感病毒测序株和流行病学数据来源乙型流感病毒测序株来源于20102015年湖北省流感监测网络流感样病毒分离株。20102015年湖北省流感样本核酸荧光PCR检测数、阳性数以及Victoria系、Yamagata系病毒分离周分布数和总数来源于中国流感监测信息系统(http://10.249.1.170/UVSSERVER2.0/)。
1.2 毒株RNA提取,HA和NA基因反转录,PCR扩增及测序取各测序毒株100 μl,采用BIO ROBOT EZ1核酸提取仪EZ1 virus mini kit v2.0试剂盒提取RNA。HA和NA基因测序引物序列由国家流感中心提供。采用Access RT-PCR System反转录和扩增。PCR产物凝胶电泳阳性样送北京华大基因公司进行序列测定。
1.3 HA和NA基因进化树分析序列采用DNAStar 7.1.0软件拼接,并上传至全球流感共享数据库(global initiative on sharing all influenza data,GISAID) EpiFlu数据系统(http://platform.gisaid.org/epi3/frontend),从该系统下载20102015年湖北省29株乙型流感病毒HA和NA氨基酸序列,毒株均来自湖北省流感监测网络,其中5株为湖北省疾病预防控制中心测序,24株为国家流感中心测序。同时下载流感病毒进化树分支参考株对应氨基酸序列[9-10]。HA和NA进化分支参考株参照20092015年间欧洲疾病预防控制中心(http://ecdc.europa.eu/) 的流感季度概况报告,Yamagata系1~3分支参考株分别为B/Florida/4/2006、B/Brisbane/3/2007和B/Wisconsin/1/2010;Victoria系1A、1B、3~5分支参考株分别为B/Brisbane/60/2008、B/Sydney/508/2010、B/Uruguay/12/2008、B/Malaysia/2506/2004和B/Cambodia/30/2011,其余亚分支参考株见图 2。HA和NA进化树采用Mega 5.2.2软件绘制,建树方法为邻接法(neighbor-joining,NJ),Bootstrap=1000。
1.4 氨基酸突变位点分析HA抗原表位、NA耐药位点和其他氨基酸突变位点采用Mega 5.2 Sequence Data Explorer分析,氨基酸突变位点通过病毒与其对应进化分支的参考株序列比对获得。
1.5 三维建模分析通过SWISS-MODEL搜索NA氨基酸序列的三维结构模板并模拟其三维结构[11-14]。神经氨酸酶活性位点、神经氨酸酶非活性位点和差异氨基酸位点的结构均通过PyMOL 1.4.1软件编辑并标出[15]。
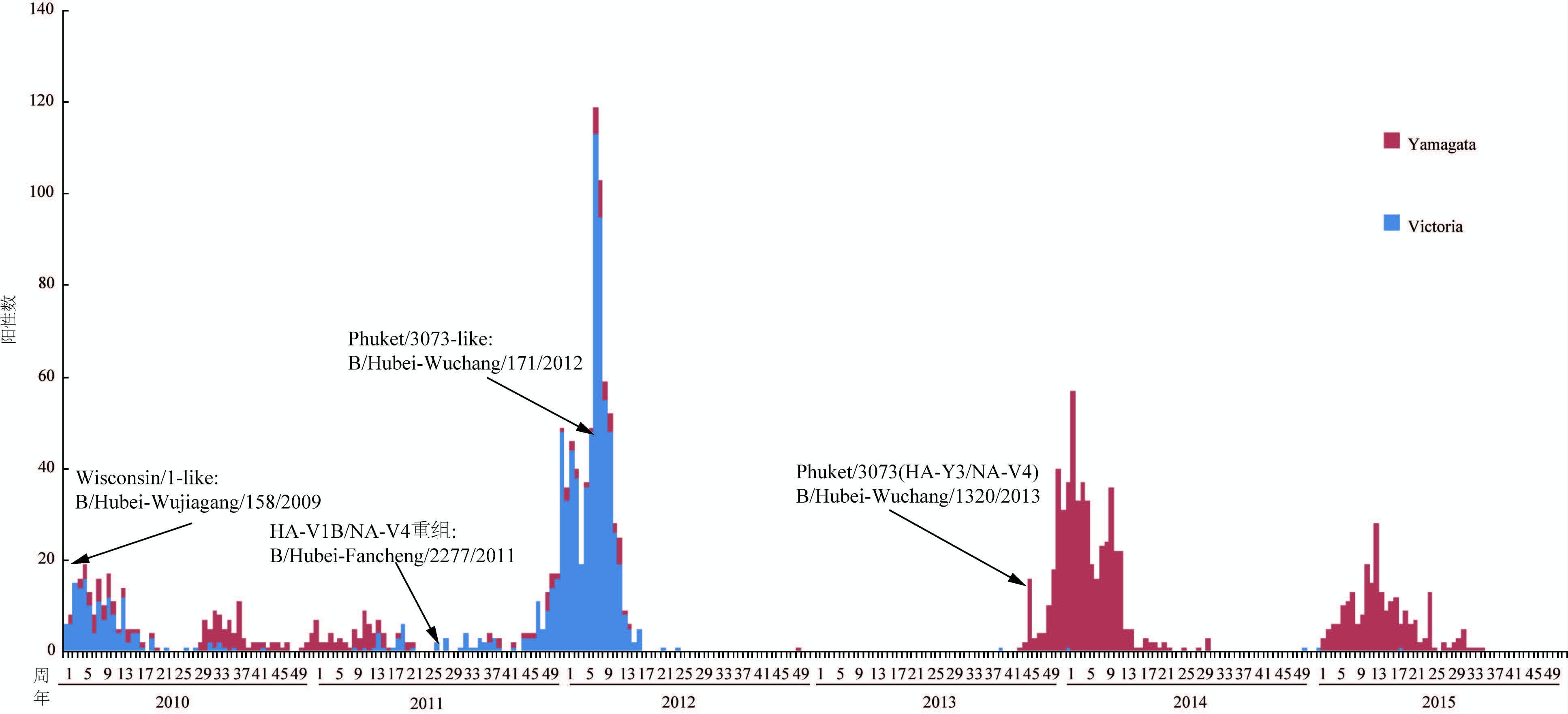
2 结果 2.1 流行情况2010-2015年湖北省流感监测网络共对74 523份样本进行乙型流感核酸荧光PCR检测,阳性3492份,分离907株Victoria系病毒和1141株Yamagata系病毒。Victoria和Yamagata系病毒流行高峰都集中在冬季峰的12月,但20122013年间无冬季峰,主要流行峰为2010年、2012年、2014和2015年的12月(图 1)。
|
| 图 1 2010-2015年湖北省乙型流感病毒分离周分布和进化分支首例株分布 Figure 1 Distribution of isolated influenza B viruses and first reassortant virus in Hubei,2010-2015 |
| |
首例湖北省HA-Y3/NA-V4系间重配株B/Hubei-Wuchang/1320/2013(2013年11月8日采样)出现在2013年Yamagata系的冬季峰,且2013年43周后主要流行Yamagata系乙型流感病毒,偶尔2~3例Victoria系乙型流感病毒。首例湖北省HA-V1B/NA-V4系内重配B/Hubei-Fancheng/2277/2011(2011年7月3日采样)则处在2010年Victoria系流行年向2011年Yamagata系流行年的过渡期。首例湖北省Wisconsin/1类病毒和Phuket/3073类病毒分别为B/Hubei-Wujiagang/158/2009(2009年3月4日采样)和B/Hubei-Wuchang/171/2012(2012年2月12日采样)。
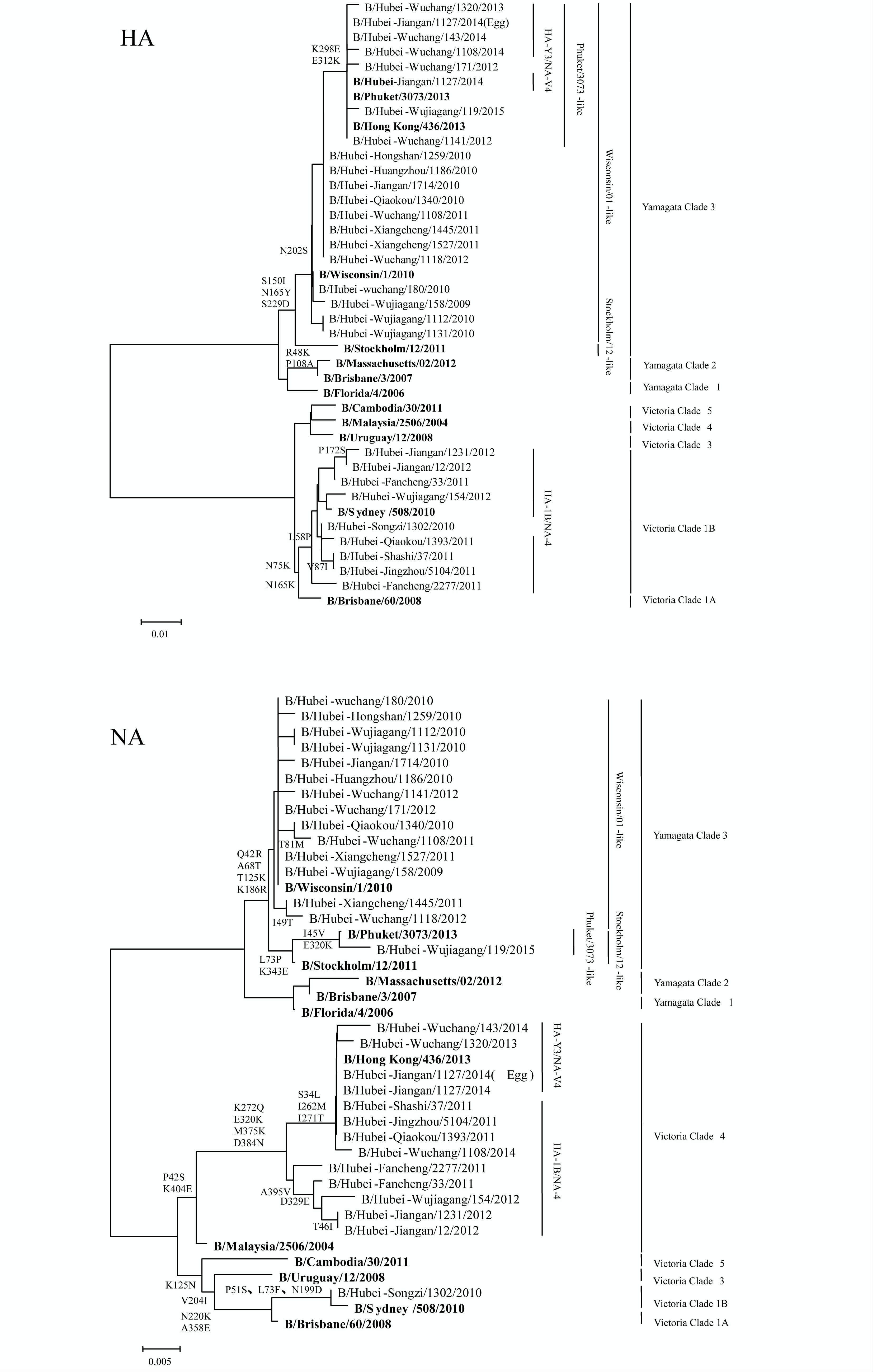
2.2 HA和NA氨基酸进化树分析湖北省5株乙型流感病毒属于Yamagata系HA蛋白3分支和Victoria系NA蛋白4分支的系间重配(HA-Y3/NA-V4系间重配),8株属于Victoria系HA蛋白1B分支和NA蛋白4分支的系内重配(HA-V1B/NA-V4系内重配)。
湖北省乙型流感病毒进化树分布为HA进化树中20株Yamagata 3分支的Wisconsin/01类病毒包含8株Phuket/3073类病毒,而Phuket/3073类病毒包含5株HA-Y3/NA-V4系间重配病毒,9株Victoria 1B分支病毒包含8株HA-V1B/NA-V4系内重配病毒;NA进化树中14株Yamagata 3分支病毒为Wisconsin/01类病毒,另1株Yamagata 3分支病毒为Phuket/3073类病毒且属于Stockholm/12类病毒而非Wisconsin/01类病毒,Oong等[8]也曾报道过Phuket/3073类病毒在HA和NA进化树中的不同从属 。仅有1株Victoria 1B分支病毒,其余13株Victoria 4分支病毒包含8株HA-V1B/NA-V4系内重配病毒和5株HA-Y3/NA-V4系间重配病毒(图 2)。湖北省乙型病毒Victoria系主要集中在1B,这与我国同期乙型病毒的基因型分布一致[16],其中20112012年的湖北省Victoria系病毒均为HA-V1B/NA-V4系内重配。
|
| 图 2 2010-2015年湖北省乙型流感病毒HA和NA进化树 Figure 2 Phylogenetic tree of HA and NA genes of influenza B viruses in Hubei,2010-2015 |
| |
将湖北省乙型流感病毒与对应HA和NA进化分支中参考株氨基酸序列进行比对分析获得其氨基酸突变位点,并将突变位点与携带该突变位点毒株数和从属该进化分支毒株数列于表 1。它们在HA抗原表位120-loop (116-137)、150-loop (141-150)、160-loop (162-167)和190-helix (194-202)处均存在突变位点[17-20],主要集中在Victoria 1B分支和Yamagata 3分支的Wisconsin/01类病毒株,其中120-loop上的突变位点尤为突出,Victoria系和Yamagata系均有,44.4%(4/9)的Victoria 1B分支病毒拥有N129S突变,80.0%(16/20)的Yamagata系Wisconsin/01类病毒拥有N116K突变,这一结果与120-loop是4个抗原表位中突变最为频繁的表位一致[21-22],其他突变表位和数量分布见表 1。
| 蛋白 | Victoriae | Yamagata 3 | |||
| Victoria 1Bf | Victoria 4 | Wisconsin/01-like | Phuket/3073-like | ||
| HA | P58S(1g/9h) | N43S(1/20) | N43S(1/9) | ||
| T73M(1/9) | N116K(16/20)(a) | L172Q(1/9) | |||
| V87I(2/9) | A146T(2/20)(b) | V219I(1/9) | |||
| N129S(4/9) (a) | L172Q(1/20) | R278K(1/9) | |||
| K162R(1/9) (c) | T198N(1/20) | ||||
| N171D(4/9) | V229I(1/20) | ||||
| P172S(2/9) | R278K(1/20) | ||||
| S197N(8/9) d | K298E(8/20) | ||||
| K330E(1/9) | |||||
| NA | L27S(1/1) | I6T(1/13) | F12L(2/14) | R186K(1/1) | |
| S34L(8/13) | I45V(1/14) | D197N(1/1) | |||
| I36V(1/13) | I49T(2/14) | ||||
| P41S(2/13) | M50I(1/14) | ||||
| T46I(2/13) | N59K(1/14) | ||||
| D53N(1/13) | L73P(1/14) | ||||
| I262M(8/13) | L74P(1/14) | ||||
| I271T(8/13) | P78Q(1/14) | ||||
| K272Q(13/13) | T81M(2/14) | ||||
| E320K(13/13) | A218E(1/14) | ||||
| D329E(4/13) | E320K(1/14) | ||||
| D342G(12/13) | N340D(1/14) | ||||
| D342S(1/13) | K343E(1/14) | ||||
| K373E(13/13) | |||||
| G362A(1/13) | |||||
| M375K(13/13) | |||||
| D384N(13/13) | |||||
| A395V(5/13) | |||||
| A395T(1/13) | |||||
| M403I(1/13) | |||||
| I455V(1/13) | |||||
| 注:a、b、c、d分别表示HA抗原表位120-loop、150-loop、160-loop和190-helix处的突变位点; e表示基因组分支及类毒株型; f为氨基酸突变位点根据对应参考株序列比对(Victoria-1B参考株为B/Sydney/508/2010,Victoria 4参考株为B/Malaysia/2506/2004,Yamagata 3的Wisconsin/01类毒株参考株为B/Wisconsin/1/2010,Yamagata 3的Phuket/3073类毒株参考株为B/Phuket/3073/2013); g为从属对应进化分支参考株且携带该突变位点的毒株数; h为从属对应进化分支参考株的毒株数。 | |||||
湖北省乙型流感病毒NA氨基酸突变位点数明显高于HA(表 1),而NA进化树中Victoria 4分支的毒株包含8株HA-V1B/NA-V4系内重配病毒和5株HA-Y3/NA-V4系间重配病毒,因此该分支的NA氨基酸突变位点数多的原因可能是系间和系内病毒的NA基因重配引起。对神经氨酸酶活性位点和神经氨酸酶非活性位点的分析[23-24],仅发现B/Hubei-Wujiagang/119/2015拥有神经氨酸酶活性位点D197N。
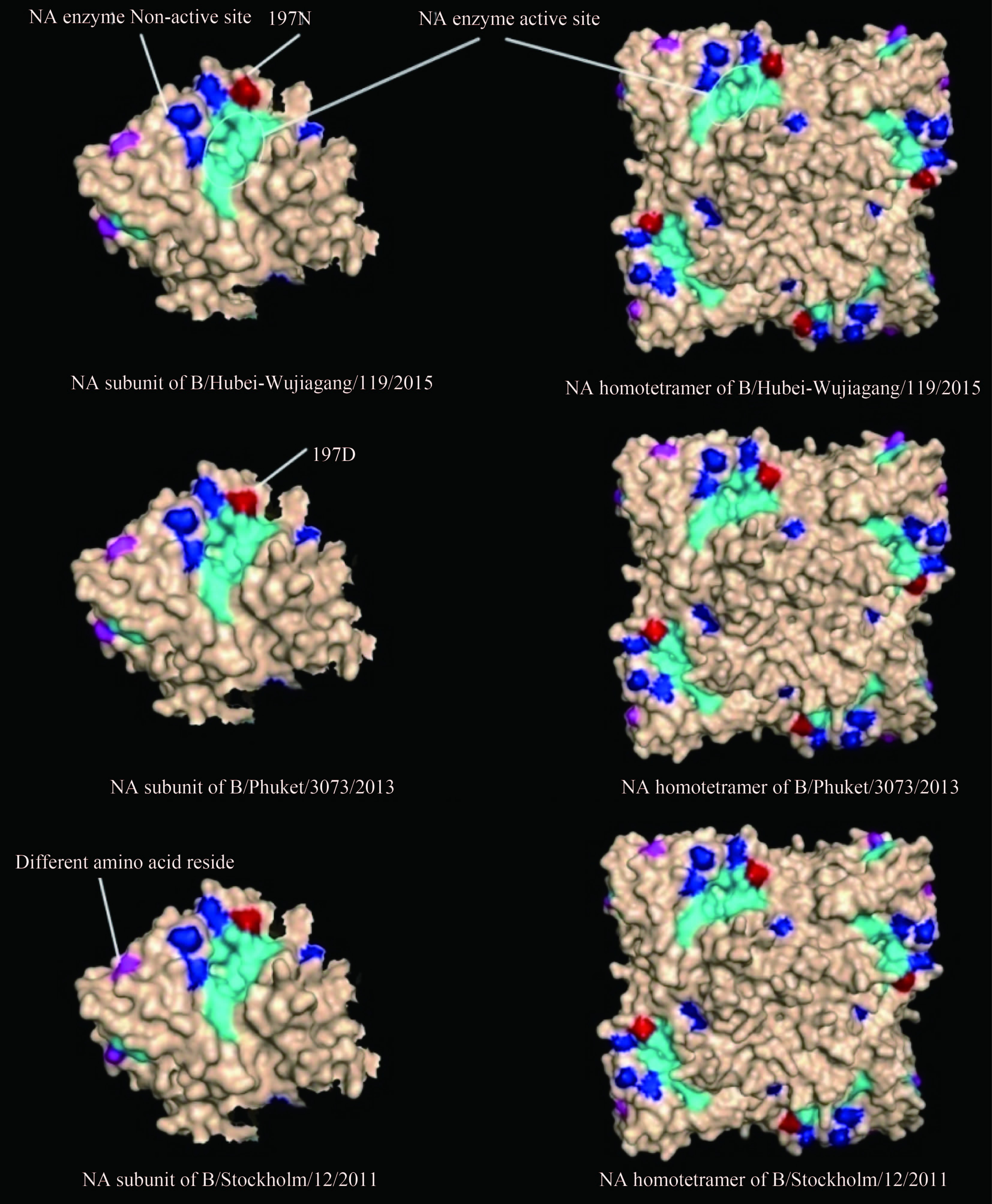
2.4 耐药株三维建模分析携带D197N耐药位点的B/Hubei-Wujiagang/119/2015在NA进化树中亲缘性与B/Phuket/3073/2013最近,B/Stockholm/12/2011次之。三者在神经氨酸酶活性位点197和其他氨基酸位点存在差异,为了分析这些差异对NA蛋白神经氨酸酶活性区域的影响,通过SWISS-MODEL搜索并获得NA蛋白共同建模模板(PDB ID: 4 cpl),利用共同模板对3个毒株分别建模后采用PyMOL软件编辑NA蛋白的神经氨酸酶活性位点、神经氨酸酶非活性位点和差异氨基酸位点区域[11-15]。
三维建模图表明神经氨酸酶活性位点在NA蛋白凸起面一侧构成可结合底物口袋状的酶活性中心,神经氨酸酶非活性位点环绕经氨酸酶活性位点,或距离很远,但是3株病毒的差异氨基酸位点离氨酸酶活性位点很远,且存在结构差异。197N与另外2株病毒的197D的结构明显不同,使紧密的三突起峰分散,197位点并非神经氨酸酶结合底物核心区域(图 3)。
|
| 图 3 湖北省乙型流感病毒NA蛋白单体、四聚体模拟图 Figure 3 Simulated subunit and homotetramer of NA protein of influenza B viruses in Hubei |
| |
甲型和乙型流感病毒都采用抗原改变的方式逃逸宿主免疫压力[25],基因重配是流感病毒抗原改变的方式之一,乙型流感病毒与甲型流感病毒一样也会出现不同种病毒间的基因重配[5],自从B/Yamagata/16/88和B/Victoria/2/87代表株发现以来[4],Victoria系和Yamagata系病毒就存在系内重配和系间重配[5, 7-8],20102015年湖北省流感监测网络的乙型流感病毒同样存在HA-V1B/NA-V4系内重配和HA-Y3/NA-V4系间重配,分离数周分布图表明系内重配和系间重配的病毒均会造成该流感病毒的大流行,这与陈继明等[26]关于乙型流感病毒变异和乙型流感流行的关系论述一致。
笔者还发现HA-Y3/NA-V4系间重配参考病毒株B/Hong Kong/436/2013[9-10] (2013年12月)采集日期晚于首例湖北省系间重配株B/Hubei-Wuchang/1320/2013(2013年11月8日),这表明湖北省的系间重配株发现时间早于香港。为追溯系间重配株更早的发现时间,在GISAID数据库中对B/Hong Kong/436/2013病毒株的NA氨基酸序列搜索并获得500份97%以上序列匹配度的毒株,然后对这些毒株分别进行HA和NA进化树分析,发现92株属于HA-Y3/NA-V4系间重配株,这些毒株均包含了病毒采集时间,其中最早的系间重配株和采集时间可追溯到日本札幌B/SAPPORO/1/2013(2013年2月4日),这也表明湖北省系间重配株源于省外。虽然HA-Y3/NA-V4系间重配株首先在日本发现,东南亚、美国等地都存在,但大部分HA-Y3/NA-V4系间重配株存在于中国,这也与2014年中国69%的Yamagata系病毒属于HA-Y3/NA-V4系间重配株一致[10]。
湖北省乙型流感病毒在4个HA抗原表位区均有突变位点,120-loop是突变最为频繁的区域[21],其中129位点的突变可引起抗原变化[27],116位点则是Victoria系病毒的独特表位[28] ,N129S和N116K在湖北省乙型流感病毒中都有发现。150-loop呈长突出环结构,占据150-loop显著表面结构的突变位点有:141、147、148、149和150 [18, 29-30],但湖北省两株乙型流感病毒携带的却是A146T,其抗原特性有待进一步研究。160-loop是乙型流感病毒HA蛋白唯一插入和删除的区域[5, 18, 31],笔者发现163N插入位点[18, 29],162、164和165氨基酸突变位点虽然在160-loop上多有报道[18, 29-30],但湖北省只发现K162R突变。190-helix外表面的195、196、197、199和200位点具有很强的抗原性[18],湖北省8株乙型流感病毒均携带S197N突变。Victoria和Yamagata系病毒在NA上的系间和系内重组可能使得NA氨基酸突变位点数多于HA。
黄维娟等[32]在HA-Y3/NA-V4系间重配株B/Hunan-Lingling/350/2013的NA蛋白上发现D197N突变。湖北省B/Hubei-Wujiagang/119/2015并非重配株,但同样携带D197N,D197N会破坏197位点与R150位点的相互作用,降低关键抑制结合位点的稳定性,形成潜在的耐药作用[33],但是D197N的三维结构表明该位点并非直接与底物或抑制物接触,其突变株对奥司他韦的敏感性存在差异(奥司他韦IC50升高存在2 ~10倍差别),暗示NA进化时其他位点可能影响D197N对奥司他韦敏感性的作用[32, 34]。
本文通过对湖北省乙型流感病毒流行病学和基因进况进行分析,探讨了湖北省乙型流感重配株的时间分布和溯源情况,同时也深入分析了乙型流感的氨基酸突变情况,为流感防控工作提供了科学的理论支持。
| [1] | Centers for Disease Control and Prevention. Influenza-associated pediatric deaths-United States, September 2010-August 2011[J]. MMWR Morb Mortal Wkly Rep, 2011, 60 (36) : 1233–1238 . |
| [2] | Yamashita M, Krystal M, Fitch WM, et al. Influenza B virus evolution:co-circulating lineages and comparison of evolutionary pattern with those of influenza A and C viruses[J]. Virology, 1988, 163 (1) : 112–122 . |
| [3] | Glezen WP, Schmier JK, Kuehn CM, et al. The burden of influenza B:a structured literature review[J]. Am J Public Health, 2013, 103 (3) : e43–e51 . |
| [4] | Rota PA, Wallis TR, Harmon MW, et al. Cocirculation of two distinct evolutionary lineages of influenza type B virus since 1983[J]. Virology, 1990, 175 (1) : 59–68 . |
| [5] | McCullers JA, Wang GC, He SQ, et al. Reassortment and insertion-deletion are strategies for the evolution of influenza B viruses in nature[J]. J Virol, 1999, 73 (9) : 7343–7348 . |
| [6] | Shaw MW, Xu XY, Li Y, et al. Reappearance and global spread of variants of influenza B/Victoria/2/87 lineage viruses in the 2000-2001 and 2001-2002 seasons[J]. Virology, 2002, 303 (1) : 1–8 . |
| [7] | European Centre for Disease Prevention and Control. Influenza virus characterisation, summary Europe, September 2015[R]. Stockholm:ECDC,2015. |
| [8] | Oong XY, Ng KT, Lam TTY, et al. Epidemiological and evolutionary dynamics of influenza B viruses in Malaysia,2012-2014[J]. PLoS One, 2015, 10 (8) : e0136254. |
| [9] | WHO Influenza Centre London. Report prepared for the WHO annual consultation on the composition of influenza vaccine for the Southern Hemisphere 2015[R]. London, United Kingdom:National Institute for Medical Research,2014. |
| [10] | Huang WJ. Information and analysis of global and Chinese influenza surveillance[A]//Collected papers of annual conference of national influenza and avian influenza between 2013 and 2014[C]. 2014. (in Chinese) 黄维娟. 全球及我国流感监测概况及分析[A]//2013-2014年度全国流感、禽流感监测和防控工作年会论文汇编[C]. 2014. |
| [11] | Biasini M, Bienert S, Waterhouse A, et al. SWISS-MODEL:modelling protein tertiary and quaternary structure using evolutionary information[J]. Nucleic Acids Res, 2014, 42 (W1) : W252–W258 . |
| [12] | Arnold K, Bordoli L, Kopp J, et al. The SWISS-MODEL workspace:a web-based environment for protein structure homology modelling[J]. Bioinformatics, 2006, 22 (2) : 195–201 . |
| [13] | Kiefer F, Arnold K, Künzli M, et al. The SWISS-MODEL repository and associated resources[J]. Nucleic Acids Res, 2009, 37 (S1) : D387–392 . |
| [14] | Guex N, Peitsch MC, Schwede T. Automated comparative protein structure modeling with SWISS-MODEL and Swiss-PdbViewer:a historical perspective[J]. Electrophoresis, 2009, 30 (S1) : S162–S173 . |
| [15] | Delano WL. The PyMOL molecular graphics system[M]. San Carlos, CA: De Lano Scientific LLC, 2002 . |
| [16] | Li XY, Cheng YH, Tan MJ, et al. Antigenic and genetic characteristics of influenza B virus in the mainland of China,2012-2013[J]. Disease Surveillance, 2014, 29 (1) : 14–20 .(in Chinese) 李希妍, 成艳辉, 谭敏菊, 等. 2012-2013流感监测年度中国大陆B型流感病毒抗原性及基因特性分析[J]. 疾病监测,2014,29 (1) :14–20. |
| [17] | Wang QH, Tian X, Chen XR, et al. Structural basis for receptor specificity of influenza B virus hemagglutinin[J]. Proc Natl Acad Sci USA, 2007, 104 (43) : 16874–16879 . |
| [18] | Wang QH, Cheng F, Lu MY, et al. Crystal structure of unliganded influenza B virus hemagglutinin[J]. J Virol, 2008, 82 (6) : 3011–3020 . |
| [19] | Shen J, Kirk BD, Ma J, et al. Diversifying selective pressure on influenza B virus hemagglutinin[J]. J Med Virol, 2009, 81 (1) : 114–124 . |
| [20] | Ni FY, Kondrashkina E, Wang QH. Structural basis for the divergent evolution of influenza B virus hemagglutinin[J]. Virology, 2013, 446 (1/2) : 112–122 . |
| [21] | Verhoeyen M, van Rompuy L, Jou WM, et al. Complete nucleotide sequence of the influenza B/Singapore/222/79 virus hemagglutinin gene and comparison with the B/Lee/40 hemagglutinin[J]. Nucleic Acids Res, 1983, 11 (14) : 4703–4712 . |
| [22] | Fang QX, Gao Y, Chen MF, et al. Molecular epidemiology and evolution of influenza A and B viruses during winter 2013-2014 in Beijing, China[J]. Arch Virol, 2015, 160 (4) : 1083–1095 . |
| [23] | Nguyen HT, Fry AM, Gubareva LV. Neuraminidase inhibitor resistance in influenza viruses and laboratory testing methods[J]. Antivir Ther, 2012, 17 (1 Pt B) : 159–173 . |
| [24] | Colman PM, Hoyne PA, Lawrence MC. Sequence and structure alignment of paramyxovirus hemagglutinin-neuraminidase with influenza virus neuraminidase[J]. J Virol, 1993, 67 (6) : 2972–2980 . |
| [25] | Webster RG, Laver WG, Air GM, et al. Molecular mechanisms of variation in influenza viruses[J]. Nature, 1982, 296 (5853) : 115–121 . |
| [26] | Chen JM, Guo YJ, Wu KY, et al. The origin and evolution of two genetic lineages of influenza B virus[J]. Chinese Journal of Virology, 2001, 17 (4) : 322–327 .(in Chinese) 陈继明, 郭元吉, 吴昆昱, 等. 乙型流行性感冒病毒两大谱系的起源及其演变特征[J]. 病毒学报,2001,17 (4) :322–327. |
| [27] | Lugovtsev VY, Vodeiko GM, Strupczewski CM, et al. Generation of the influenza B viruses with improved growth phenotype by substitution of specific amino acids of hemagglutinin[J]. Virology, 2007, 365 (2) : 315–323 . |
| [28] | Nakagawa N, Suzuoki J, Kubota R, et al. Discovery of the neutralizing epitope common to influenza B virus victoria group isolates in Japan[J]. J Clin Microbiol, 2006, 44 (4) : 1564–1566 . |
| [29] | Webster RG, Berton MT. Analysis of antigenic drift in the haemagglutinin molecule of influenza B virus with monoclonal antibodies[J]. J Gen Virol, 1981, 54 (Pt 2) : 243–251 . |
| [30] | Nakagawa N, Kubota R, Nakagawa T, et al. Neutralizing epitopes specific for influenza B virus Yamagata group strains are in the ‘loop’[J]. J Gen Virol, 2003, 84 (Pt 4) : 769–773 . |
| [31] | Nerome R, Hiromoto Y, Sugita S, et al. Evolutionary characteristics of influenza B virus since its first isolation in 1940:dynamic circulation of deletion and insertion mechanism[J]. Arch Virol, 1998, 143 (8) : 1569–1583 . |
| [32] | Huang WJ, Li XY, Tan MJ, et al. Susceptibility of influenza B viruses to neuraminidase inhibitors isolated during 2013-2014 influenza season in Mainland China[J]. Chinese Journal of Virology, 2015, 31 (2) : 152–156 .(in Chinese) 黄维娟, 李希妍, 谭敏菊, 等. 2013-2014年流感流行季节中国大陆B型流感病毒对NAI的敏感性[J]. 病毒学报,2015,31 (2) :152–156. |
| [33] | Oakley AJ, Barrett S, Peat TS, et al. Structural and functional basis of resistance to neuraminidase inhibitors of influenza B viruses[J]. J Med Chem, 2010, 53 (17) : 6421–6431 . |
| [34] | Meijer A, Rebelo-de-Andrade H, Correia V, et al. Global update on the susceptibility of human influenza viruses to neuraminidase inhibitors,2012-2013[J]. Antiviral Res, 2014 : 31–41 . |
 2016, Vol. 31
2016, Vol. 31


