扩展功能
文章信息
- 许美燕, 蔡茂荣, 胡慧玲, 林淑银
- XU Mei-yan, CAI Mao-rong, HU Hui-ling, LIN Shu-yin
- 2013-2014年漳州市手足口病的病原学特征及分子流行病学研究
- Etiology and molecular epidemiology of hand, foot and mouth disease in Zhangzhou, 2013-2014
- 疾病监测, 2016, 31(10): 824-830
- Disease Surveillance, 2016, 31(10): 824-830
- 10.3784/j.issn.1003-9961.2016.10.007
-
文章历史
- 收稿日期:2016-03-30
手足口病(hand foot mouth disease,HFMD)是由多种人肠道病毒引起的一种儿童常见传染病,是我国法定报告管理的丙类传染病。大多数患者症状轻微,以发热和手、足、口腔等部位出现皮疹或疱疹为主要症状。少数患者可出现无菌性脑膜炎、脑炎、急性弛缓性麻痹、神经源性肺水肿和心肌炎等,个别重症患儿病情进展快,可导致死亡。
引起HFMD的病原组成较为复杂,包括人肠道病毒71型(EV71),柯萨奇病毒A组(Cox A2~12、Cox A16和Cox A20)和B组(Cox B1~6)及部分埃可病毒(E3、E4、E5、E6、E9、E11、E18、E25和E30)[1]。目前HFMD常规病原学监测只要求检测肠道病毒通用型、EV71和Cox A16,对引发HFMD的非EV71和非Cox A16的肠道病毒未做进一步的型别鉴定。而近年来的研究报告显示,非EV71及非Cox A16的肠道病毒在HFMD病原谱中的比例日渐增加,为全面了解本地区HFMD的病原构成情况,本研究通过实时荧光定量反转录聚合酶链反应(real-time reverse transcription polymerase chain reaction,Real-time RT-PCR)、反转录聚合酶链反应(reverse transcription polymerase chain reaction,RT-PCR)扩增肠道病毒VP1基因并测序,确定病原体,并通过构建系统进化树对病原体进行分子流行病学分析。
1 材料与方法 1.1 标本采集按照卫生部发布的《手足口病预防控制指南》(2009年版)执行。根据该指南的病原学监测要求,以县(区)为单位,每月至少采集5例首次就诊的HFMD普通病例标本。当月县(区)病例总数少于5例时,全部采样。漳州市共11个县(区),每个县(区)1个监测点医院,市区另设1个监测点医院,共12个监测点医院。采集发病3 d内的患者咽拭子或肛拭子标本。标本采集后,立即置3~5 ml采样液中。2013-2014年共采集12个监测点医院临床诊断HFMD疑似病例的标本1421份。
1.2 病毒RNA的提取采用QIAGEN公司的QIAamp Viral RNA Mini Kit试剂盒进行病毒RNA提取。
1.3 Real-time RT-PCR采用江苏硕世肠道病毒通用型、EV71、Cox A16荧光PCR检测试剂盒对标本RNA进行Real-time RT-PCR检测。该反应在ABI7500荧光定量PCR仪上进行。
1.4 基因扩增及序列测定 1.4.1 EV71和Cox A16 VP1区基因全长扩增从不同县(区)的EV71和Cox A16核酸阳性标本中,每年各随机抽取5份,采用QIAGEN one step RT-PCR试剂盒进行VP1区基因全长扩增。引物序列为:EV71-VP1-A:5'-AAG TCG CGA GAG CTG TCT TC-3',EV71-VP1-S:5'-GCA GCC CAA AAG AAC TTC AC-3';Cox A16-VP1-S:5'-ATT GGT GCT CCC ACT ACA GC-3',Cox A16-VP1-A:5'-GCT GTC CTC CCA CAC AAG AT-3'。RT-PCR反应条件:50 ℃ 30 min;95 ℃ 15 min;94 ℃ 30 s,50 ℃ 40 s,72 ℃ 80 s,35个循环;72 ℃ 10 min。扩增产物约为1100 bp,包括VP1区全长基因。该反应在Eppendorf Mastercycler pro PCR仪上进行。
1.4.2 非EV71及非Cox A16的肠道病毒分型从不同县(区)的肠道病毒通用阳性而EV71和Cox A16均阴性的标本中,每年随机抽取20份,采用QIAGEN one step RT-PCR试剂盒进行VP1区基因5'端核苷酸扩增。用于扩增VP1区的相关引物名称为[2-4]:222/292,HEV-A的特异性引物486/488,HEV-B的特异性引物490/492,AN88/AN89。RT-PCR反应条件:50 ℃ 30 min;95 ℃ 15 min;94 ℃ 30 s,46 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 10 min。
1.4.3 Cox A6和Cox A10 VP1区基因全长扩增根据非EV71及非Cox A16的肠道病毒分型结果,随机抽取Cox A6和Cox A10各4份,使用半巢式RT-PCR进行VP1区基因3'端核苷酸扩增。使用引物[2, 5]:第1步RT-PCR反应使用引物EUC2/EUG3(EUG3为根据EUG3a、EUG3b和EUG3c序列合成的简并引物);第2步RT-PCR反应以第1步RT-PCR的产物为模板,使用引物EUG3/489。将Cox A6和Cox A10的VP1区两部分基因:486/488测序结果即VP1区基因5'端,EUG3/489测序结果即VP1区基因3'端,分别进行拼接后获得VP1区基因全长。
1.4.4 扩增产物的纯化及测序扩增产物经1%琼脂糖凝胶电泳后,用QIA quick Gel extraction Kit回收纯化目的条带。纯化后的扩增产物按TaKaRa pMDTM 18-T Vector Cloning Kit说明连接至pMD18-T载体,转化至DH5α感受态细胞,重组克隆经RV-M和M13-47通用引物鉴定。用TIANGEN公司质粒提取试剂盒提取重组阳性克隆质粒送invitrogen公司测序。
1.4.5 肠道病毒病原体的确定及VP1区基因全长序列分析所有的VP1基因序列使用HEV在线分型工具(www.rivm.nl/mpf/enterovirus/typingtool)进行型别鉴定。Cox A6和Cox A10的测序结果采用DNAStar 5.00软件中的Seqman程序进行拼接组装成完整的VP1基因序列。全部核苷酸和氨基酸的序列分析和同源性比较应用DNAStar软件中的MegAlign完成,系统进化树构建应用Mega 4.0软件完成。所有参考序列均来自GenBank。
2 结果 2.1 Real-time RT-PCR检测结果2013-2014年共对1421份疑似HFMD标本进行肠道病毒通用型、EV71和Cox A16的核酸检测,结果见表 1。不同年份之间不同型别检测结果差异有统计学意义(χ2 =160.04,P=0.00)。
| 年份 | 标本数 | 肠道病毒通用型 核酸检测 |
EV71 | Cox A16 | EV71和Cox A16 混合感染 |
其他肠道病毒阳性 | |||||||||
| 阳性数 | 阳性率 (%) |
阳性数 | 阳性率 (%) |
阳性数 | 阳性率 (%) |
阳性数 | 阳性率 (%) |
阳性数 | 阳性率 (%) |
||||||
| 2013 | 622 | 480 | 77.17 | 91 | 14.63 | 72 | 11.58 | 0 | 0.00 | 317 | 50.96 | ||||
| 2014 | 799 | 583 | 72.97 | 131 | 16.40 | 268 | 33.54 | 8 | 1.00 | 176 | 22.03 | ||||
| 合计 | 1421 | 1063 | 74.81 | 222 | 15.62 | 340 | 23.93 | 8 | 0.56 | 493 | 34.69 | ||||
从不同县(区)的肠道病毒通用型阳性而EV71和Cox A16均阴性的标本中,每年随机抽取20份,共40份标本进行分型鉴定,成功鉴定38份,2份未能分型,结果为:Cox A6占65.79%(25/38),Cox A10占21.05%(8/38),Cox A5占2.63%(1/38),Cox A4占7.89%(3/38),Cox A9占2.63%(1/38)。
2.3 EV71和Cox A16 VP1区基因全长序列同源性比较和系统进化分析2013-2014年对EV71和Cox A16标本各10份进行VP1区基因全长扩增和测序,获得覆盖VP1区基因全长的1100 bp序列后,利用Clustal X 1.83软件进行VP1区基因序列位置的确定,用BioEdit 7.0软件对序列进行剪切,结果均获得891 bp的VP1区全长核苷酸序列。系统进化树显示(图 1):漳州市的10个EV71 VP1区基因全长序列均集中于C4基因亚型的C4a进化分支;漳州市的10个Cox A16 VP1区基因全长序列均属于B1基因亚型,在进化树中分别归属于两支,其中9份标本与H573F-SD-CHN-2008、EV218-GD-CHN-2014等中国内地分离株构成亲缘关系较近的一支,均属于B1基因亚型的B1b分支,ZZ14-420-FJ-CHN-2014标本与江苏南京2013年的分离株DC13-012-NJ-CHN-2013亲缘关系较近,属于B1基因亚型的B1a分支。

|
| 图 1 基于EV71和Cox A16的完整VP1区核苷酸序列(891 bp)构建的系统进化树 Figure 1 Phylogenetic trees based on entire VP1 nucleotide sequences (891 bp) of EV71 and Cox A16 注:(a)●漳州市2013-2014 EV71阳性病毒,○EV71原型株BrCr,■外群Cox A16 G10;(b)●漳州市2013-2014 Cox A16阳性病毒,○Cox A16原型株Cox A16 G10,■外群EV71 BrCr。 |
| |
本研究从Cox A6阳性标本中随机抽取4份标本进行完整VP1区基因的扩增测序,获得的Cox A6完整VP1区基因序列全长均为915 bp,与Cox A6的原型株Gdula相比,未发现核苷酸的缺失和插入。将漳州市的4个Cox A6 VP1区基因全长序列与GenBank检索的29株Cox A6型病毒代表株构建基于完整VP1区基因的系统进化树(图 2),并进行核苷酸和氨基酸的同源性比较。漳州市4份CoxA6标本之间核苷酸和氨基酸同源性分别为94.1%~98.0%和97.4%~98.7%;与原型株Gdula的核苷酸同源性和氨基酸同源性分别为82.3%~82.7%和94.1%~95.1%。由于目前对Cox A6的基因分型没有明确的标准或共识,本研究参照VP1区核苷酸同源性高于85%的毒株为同一基因型,同源性高于91%的毒株为同一基因亚型这一标准[6],结合进化树分析,将Cox A6分为4个基因型:A、B、C、D。D基因型又分为D1~D3基因亚型,Cox A6同一基因型内核苷酸序列同源性均 > 85.0%(A、B基因均为单一菌株,无法比较;C内部为90.8%,D内部为89.3%~100.0%),同一基因亚型内核苷酸序列同源性均 > 91.0%(D1内部为94.1%~100.0%,D2内部为95.8%~97.2%,D3内部为93.4%~96.0%),详见表 2。漳州市的4个Cox A6 VP1区基因全长序列均分布在D1基因亚型中,进化树显示,漳州市4个Cox A6标本与原型株Gdula、国内地区两株山东分离株(92022-SD-CHN-1992,96188-SD-CHN-1996)及印度株(N-313-IND)等年代较为久远的分离株遗传距离较远,亲缘关系远,与国内近年来流行的分离株的遗传距离近,亲缘关系近。
| Cox A6基因 (亚)型 |
A | B | C | D | D1 | D2 | D3 |
| A | |||||||
| B | 85.0 | ||||||
| C | 83.0~83.4 | 82.6~83.3 | 90.8(1) | ||||
| D | 80.9~83.5 | 82.2~84.2 | 83.9~86.4 | ||||
| 89.3~100.0(1) | |||||||
| D1 | 94.1~100.0(1) | 89.3~92.2 | 90.2~95.8 | ||||
| D2 | 95.8~97.2(1) | 89.7~93.6 | |||||
| D3 | 93.4~96.0(1) | ||||||
| 注:(1)为同基因(亚)型内毒株间VP1区核苷酸序列的同源性;其他为不同基因(亚)型毒株间VP1区核苷酸序列的同源性。 | |||||||
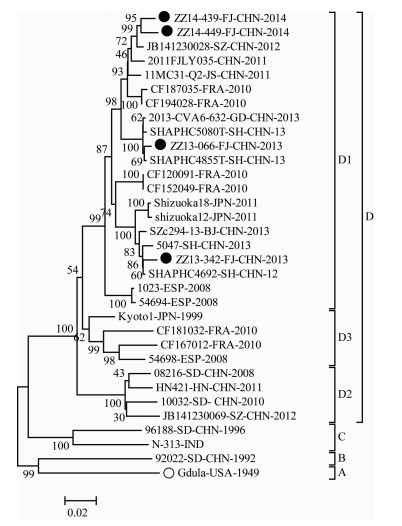
|
| 图 2 基于Cox A6的完整VP1区核苷酸序列(915 bp)构建的系统进化树 Figure 2 Phylogenetic tree based on entire VP1 nucleotide sequence (915 bp) of Cox A6 strains 注:●为漳州市2013-2014年Cox A6阳性病毒;○为Cox A6原型株Gdula。 |
| |
同样,本研究从Cox A10阳性标本中随机抽取4份标本进行完整VP1区基因的扩增测序,获得的Cox A10完整VP1区基因序列全长均为894 bp。将漳州市的4个Cox A10 VP1区基因全长序列与GenBank检索的28株Cox A10型病毒代表株构建基于完整VP1区基因的系统进化树(图 3),并进行核苷酸和氨基酸的同源性比较。漳州市的4份Cox A10标本之间核苷酸和氨基酸同源性分别为95.6%~99.7%和97.3%~99.7%;与原型株Kowalik的核苷酸和氨基酸同源性分别为76.7%~77.2%和91.6%~92.6%。目前对Cox A10的基因分型没有明确的标准或共识,本研究综合VP1区核苷酸同源性及遗传进化树分析的结果,将Cox A10分为4个基因型(A、B、C、D)。D基因型内部的核苷酸序列同源性为94.0%~100.0%,亲缘关系近,不划分基因亚型,但至少可以看出有L1~L4共4个独立的分支;Cox A10同一基因型内核苷酸序列同源性均>85.0%(A基因均为单一菌株,无法比较;B基因内部为96.6%~99.1%,C内部为95.5%~96.8%,D内部为94.0%~100.0%),详见表 3。原型株Kowalik单独分布在A基因型,B基因型主要由西班牙、法国等国家的分离株组成,中国山东、深圳、河北和江苏等地区所有的Cox A10分离株分布在C和D基因型中。漳州市的4个Cox A10 VP1区基因全长序列均分布在D基因型中,与原型株Kowalik的遗传距离较远,亲缘关系远,而与国内地区如山东WD0048-SD-CHN-2009、深圳SHZH12-JB141230348-CHN-2012、江苏M10F74-JS-CHN-2010等分离株的亲缘关系近。

|
| 图 3 基于Cox A10的完整VP1区核苷酸序列(894 bp)构建的系统进化树 Figure 3 Phylogenetic tree based on entire VP1 nucleotide sequence (894 bp) of Cox A10 strains 注:●为漳州市2013-2014年Cox A10阳性病毒;○为Cox A 10原型株Kowalik。 |
| |
| Cox A10基因型 | A | B | C | D |
| A | ||||
| B | 75.1~76.5 | 96.6~99.1(1) | ||
| C | 75.7~76.1 | 78.9~81.2 | 95.5~96.8(1) | |
| D | 76.3~77.4 | 79.0~81.3 | 81.2~84.2 | 94.0~100.0(1) |
| 注:(1)为同基因型内毒株间VP1区核苷酸序列的同源性;其他为不同基因型毒株间VP1区核苷酸序列的同源性。 | ||||
在国内外早期HFMD病原学研究中,EV71和Cox A16是其主要病原体,但近年来随着对病原学研究的深入,认为除了EV71和Cox A16外,其他肠道病毒型别在HFMD病原谱中也占有重要位置。HFMD疫情监测数据显示,在2008-2010年间,引起福建省HFMD的主要病原体为EV71和Cox A16,2008年和2009年其他肠道病毒引起福建省HFMD病例的比例只有1.0%,2010年占2.8%,但是近几年其他肠道病毒的构成比例明显上升,2011-2015年分别为26.5%、32.2%、65.7%、41.8%和59.3%。2012-2014年江苏省太仓市其他型别肠道病毒逐年出现的构成比分别为17.6%、66.7%和42.5%[7],广西地区HFMD轻症病例中,2008年其他型别肠道病毒构成比占52.7%,而2013年则增加至61.8%[8]。本研究对漳州市的2013-2014年1421份疑似HFMD标本进行肠道病毒通用型、EV71和Cox A16的核酸检测,发现2013年HFMD病原体主要为非EV71及非Cox A16的肠道病毒,阳性率高达50.96%,而2014年HFMD病原体主要为Cox A16(阳性率为33.54%),其次为非EV71及非Cox A16的肠道病毒(阳性率为22.03%)。说明在漳州市其他的肠道病毒型别在HFMD病原谱中同样占有重要位置。进一步的分型鉴定发现,非EV71及非Cox A16的肠道病毒中,以Cox A6(65.79%,25/38)和Cox A10(21.05%,8/38)为主,偶见Cox A5、Cox A4和Cox A9。陈炜等[9-10]对福建省2011-2014年其他肠道病毒HFMD标本进行分离鉴定,共鉴定出Cox A2、Cox A4~6、Cox A8~10、Cox A12、Cox A21,Cox B1~5、E3、E6、E7、E9、E16、E25、E30和EV68 22种肠道病毒,其中以Cox A6和Cox A10为优势毒株,其构成比分别为48.9%和25.3%,该结果与2009年河北、2013年广州、2012年北京、2010年上海等地区的HFMD病原谱的构成情况相似[11-14],Cox A6、Cox A10成为HFMD重要的病原之一。由此可见,HFMD的病原组成已呈现多样化和复杂性的特点,在其他肠道病毒中,Cox A6和Cox A10也占较大比例。
EV71系统进化树结果显示,漳州市2013-2014年的10个EV71 VP1区基因全长序列均集中于C4基因亚型的C4a进化分支,与2004年以来中国大陆EV71流行的基因型完全一致。Cox A16系统进化树结果显示,漳州市2013-2014年的10个Cox A16 VP1区基因全长序列均属于B1基因亚型,在进化树中分别归属于B1a和B1b两支,说明目前B1a和B1b这两条进化分支在漳州市共同流行,与近年来国内有关Cox A16分子进化方面研究结果一致。对Cox A6和Cox A10进行进一步基因分型目前并没有明确的标准或共识,本研究参照VP1区核苷酸同源性高于85%的毒株为同一基因型,同源性高于91%的毒株为同一基因亚型这一标准[6],结合进化树分析,将Cox A6和Cox A10都分为A、B、C、D 4个基因型,基因分型结果与Tan等[15]和Tian等[16]对于Cox A6、Cox A10的基因分型结果基本一致。漳州市2013-2014年的4个Cox A6 VP1区基因全长序列和4个Cox A10 VP1区基因全长序列均与原型株遗传距离较远,亲缘关系远,与国内近年来流行的分离株的遗传距离近,亲缘关系近,可见,漳州市的Cox A6和Cox A10毒株在进化上并不是独立的,而是与国内地区(如江苏、深圳等)的毒株共进化共循环的。
作者贡献:许美燕:项目设计和论文撰写
蔡茂荣、胡慧玲、林淑银:论文撰写
| [1] | Lai FF.Epidemiological and etiological characteristic analysis of hand-foot-and mouth disease in Chongqing, 2010-2013[D]. Chongqing:Chongqing Medical University, 2014:8.(in Chinese) 赖方方.2010-2013年重庆地区手足口病流行病学与住院病例病原学特征分析[D].重庆:重庆医科大学, 2014:8. http://cdmd.cnki.com.cn/Article/CDMD-10631-1014409499.htm |
| [2] | Oberste MS, Maher K, Williams AJ, et al. Species-specific RT-PCR amplification of human enteroviruses:a tool for rapid species identification of uncharacterized enteroviruses[J]. J Gen Virol , 2006, 87 (1) : 119–128. DOI:10.1099/vir.0.81179-0 |
| [3] | Oberste MS, Nix WA, Maher K, et al. Improved molecular identification of enteroviruses by RT-PCR and amplicon sequencing[J]. J Clin Virol , 2003, 26 (3) : 375–377. DOI:10.1016/S1386-6532(03)00004-0 |
| [4] | Nix WA, Oberste MS, Pallansch MA. Sensitive, seminested PCR amplification of VP1 sequences for direct identification of all enterovirus serotypes from original clinical specimens[J]. J Clin Microbiol , 2006, 44 (8) : 2698–2704. DOI:10.1128/JCM.00542-06 |
| [5] | Caro V, Guillot S, Delpeyroux F, et al. Molecular strategy for 'serotyping' of human enteroviruses[J]. J Gen Virol , 2001, 82 (1) : 79–91. DOI:10.1099/0022-1317-82-1-79 |
| [6] | Dong XN, Ying J, Chen YH. Molecular epidemiology and evolution of worldwide enterovirus 71 strains isolated from 1970 to 2004[J]. Chinese Science Bulletin , 2007, 52 (9) : 1021–1027. (in Chinese) 董晓楠, 应剑, 陈应华. 1970-2004年全球肠道病毒71型分离株的分子流行病学分析[J]. 科学通报 , 2007, 52 (9) : 1021–1027. |
| [7] | Cai YB, Ji H. Study on the epidemiological and etiological characteristics of hand-foot-mouth disease in Taicang, 2012-2014[J]. Jiangsu Journal of Preventive Medicine , 2016, 27 (2) : 198–200. (in Chinese) 蔡永彬, 嵇红. 2012-2014年太仓市手足口病流行病学和病原学特征[J]. 江苏预防医学 , 2016, 27 (2) : 198–200. |
| [8] | Yang YP, Chen MM, Ju Y, et al. Epidemiology and pathogen distribution of hand-foot-mouth disease in Guangxi, 2008-2014[J]. Journal of Applied Preventive Medicine , 2015, 21 (6) : 365–368. (in Chinese) 阳益萍, 陈敏玫, 居昱, 等. 2008-2014年广西手足口病流行病学及病原分布特征[J]. 应用预防医学 , 2015, 21 (6) : 365–368. |
| [9] | Chen W, Weng YW, He WX, et al. Molecular epidemiology of HFMD-associated pathogen coxsackievirus A6 in Fujian province, 2011-2013[J]. Chinese Journal of Virology , 2014, 30 (6) : 624–629. (in Chinese) 陈炜, 翁育伟, 何文祥, 等. 2011-2013年福建省手足口病相关病原柯萨奇病毒A组6型的分子流行病学研究[J]. 病毒学报 , 2014, 30 (6) : 624–629. |
| [10] | Chen W, Weng YW, He WX, et al. Molecular epidemiology of hand-foot-mouth disease associated pathogen coxsackievirus A10 identified in Fujian province, 2011-2014[J]. Chinese Journal of Epidemiology , 2016, 37 (4) : 563–567. (in Chinese) 陈炜, 翁育伟, 何文祥, 等. 福建省2011-2014年手足口病相关病原柯萨奇病毒A组10型的分子流行病学研究[J]. 中华流行病学杂志 , 2016, 37 (4) : 563–567. |
| [11] | Yu QL, Shen YX, Han CZ, et al. An Etiological study on the hand, foot and mouth disease in 2009 of Hebei province[J]. Modern Preventive Medicine , 2012, 39 (8) : 2026–2031. (in Chinese) 于秋丽, 申悦霞, 韩彩芝, 等. 河北省2009年手足口病病原学研究[J]. 现代预防医学 , 2012, 39 (8) : 2026–2031. |
| [12] | Zhang XL, Yu HJ, Yu Y, et al. The pathogenic spectrum and molecular epidemiology analysis of 440 hand, foot and mouth disease cases in Shanghai in 2010[J]. Journal of Microbes and Infections , 2011, 6 (4) : 214–224. (in Chinese) 张晓玲, 俞慧菊, 余曜, 等. 2010年上海部分地区440例足口病病例的病原谱及分子流行病学分析[J]. 微生物与感染 , 2011, 6 (4) : 214–224. |
| [13] | Wang HB, Lei N, Jiang XH, et al. Genetype of non-EV71, non-CoxA16 enteroviruses caused hand, foot and mouth disease in Chaoyang of Beijing[J]. Modern Preventive Medicine , 2014, 41 (9) : 1699–1703. (in Chinese) 王海滨, 雷娜, 姜晓红, 等. 北京市朝阳区手足口病非EV71、非CoxA16肠道病毒的基因型[J]. 现代预防医学 , 2014, 41 (9) : 1699–1703. |
| [14] | Zhou LL, Wei QD, Zhang LR, et al. Study on the etiology of hand, foot and mouth disease in Zhuhai, Guangdong in 2013[J]. Chinese Journal of Health Laboratory Technology , 2015, 25 (10) : 1596–1599. (in Chinese) 周兰兰, 魏泉德, 张丽荣, 等. 2013年广东省珠海市手足口病病原学研究[J]. 中国卫生检验杂志 , 2015, 25 (10) : 1596–1599. |
| [15] | Tan XJ, Li L, Zhang BM, et al. Molecular epidemiology of coxsackievirus A6 associated with outbreaks of hand, foot and mouth disease in Tianjin, China, in 2013[J]. Arch Virol , 2015, 160 (4) : 1097–1104. DOI:10.1007/s00705-015-2340-3 |
| [16] | Tian HF, Zhang Y, Sun Q, et al. Prevalence of multiple enteroviruses associated with hand, foot and mouth disease in Shijiazhuang city, Hebei province, China:Outbreaks of coxsackieviruses A10 and B3[J]. PLoS One , 2014, 9 (1) : e84233. DOI:10.1371/journal.pone.0084233 |
 2016, Vol. 31
2016, Vol. 31


