扩展功能
文章信息
- 邱小彤, 郝琴, 白雪梅
- QIU Xiao-tong, HAO Qin, BAI Xue-mei
- 猪链球菌血清未分型菌株的荚膜多糖基因簇分析
- Genetic analyzing capsular polysaccharide synthesis gene loci of the non-serotypeable Streptococcus suis
- 疾病监测, 2016, 31(11): 925-931
- Disease Surveillance, 2016, 31(11): 925-931
- 10.3784/j.issn.1003-9961.2016.11.009
-
文章历史
- 收稿日期:2016-08-03
2. 南开大学泰达生物技术研究院 分子微生物学与技术教育部重点实验室, 天津 300457
2. Key Laboratory of Molecular Microbiology and Technology, Ministry of Education, TEDA Institute of Biological Sciences and Biotechnology, Nankai University, Tianjin 300457, China
猪链球菌(Streptococcus suis)可引起猪脑膜炎、败血症、心内膜炎、关节炎等疾病,同时猪链球菌也是一种重要的人兽共患病病原体[1]。人感染猪链球菌可引起脑膜炎、中毒性休克样综合征等,严重者会导致死亡。人与病、死猪密切接触是人感染猪链球菌的主要原因。2005年7月我国四川省暴发人感染猪链球菌疫情,共造成215人感染,39人死亡[2-4]。
猪链球菌是具有荚膜的一种革兰阳性球菌,其荚膜富含多糖类物质,称为荚膜多糖(capsular polysaccharide,CPS)。荚膜多糖是猪链球菌的重要致病因子,在猪链球菌抗吞噬的过程中起关键作用[5],由基因组中的荚膜多糖基因簇(cps cluster)编码。血清型是猪链球菌的一种重要的流行病学特征,对猪链球菌感染具有重要的诊断价值。CPS抗原性的变异是猪链球菌血清分型的分子基础。根据CPS抗原性不同,猪链球菌可分为33个血清型(1~31、33与1/2型)。凝集实验和协同凝集实验是鉴定猪链球菌血清型的常规方法,但该方法需要制备昂贵的诊断血清,费时费力,结果判读受主观因素影响较大,并且随着研究的深入,无法用传统的血清凝集方法分型的菌株逐渐增多[6]。
在基因组时代,随着高通量测序技术的普及,猪链球菌33种血清型标准菌株的基因组草图完成测序。Okura等[7]系统地分析了所有血清型猪链球菌的cps基因簇,根据cps基因簇在基因组中的位置不同,猪链球菌全部血清型标准菌株的cps基因簇位置可分为5个pattern(patternⅠ-a、patternⅠ-b、patternⅡ、patternⅢ、patternⅣ),且大部分血清型属于patternⅠ-a、patternⅠ-b,即cps基因簇5′端上游为orfZ-orfX 3个保守基因,3′端下游为aroA(patternⅠ-a)或glf(patternⅠ-b)。序列比对结果证实猪链球菌CPS的合成为Wzx/Wzy决定途径,wzy基因是决定血清型别的关键基因,同时也是血清型特异基因,其在型内保守,型间差异大(同源性低于70%),因此,wzy基因成为区分不同cps型别猪链球菌的良好靶基因。近年来,多个以wzy为靶基因的高通量分子血清分型技术相继出现[8-12],这些方法灵敏度高,特异性好,逐步取代了传统的血清分型技术。分子血清分型技术及测序技术的应用极大地促进了血清未分型菌株的研究。2014年,Pan等[13]在患脑膜炎的病猪体内分离到新血清型的猪链球菌,并将该血清型命名为Chz型。2015年,又有8种新的cps型别(NCL1~NCL8)被发现[14],cps型别在表型上是否属于新的血清型尚有待证实,但它们的cps基因簇与已知血清型的cps基因簇差异非常大:除了拥有完全不同于已知血清型的型特异的wzy基因外,还包含73个新的基因,其编码蛋白主要集中于包括糖基转移酶、氨基转移酶及乙酰基转移酶等与单糖结构重复单位的形成及修饰相关的转移酶家族。这些基因与CPS抗原的形成密切相关。序列分析提示这些NCL型别极有可能代表新的血清表型。
近期,英国研究人员利用Illumina测序技术获得了41株血清未分型猪链球菌的基因组序列[15]。本研究利用该数据,截取出这些菌株的cps基因簇,并与33种血清型及NCL1~NCL8、Chz型标准菌株的cps序列进行比对,以期从基因水平上探究这些猪链球菌菌株血清不可分型的原因及cps基因簇序列的变异规律。
1 材料与方法 1.1 数据来源本研究数据来源为GenBank数据库于2016年3月公布的41株猪链球菌血清未分型菌株的全基因组序列。菌株相关信息见表 1。
| 菌株编号 | 类型 | 国家 | 宿主 | GenBank登记号 | cps型别 |
| LSS8 | 未知 | 英国 | 猪 | ERS132370 | 血清16型 |
| LSS9 | 非临床株 | 英国 | 猪 | ERS132371 | 血清10型 |
| LSS10 | 未知 | 英国 | 猪 | ERS132372 | NCL4型 |
| LSS11 | 非临床株 | 英国 | 猪 | ERS132373 | 血清31型 |
| LSS18 | 非临床株 | 英国 | 猪 | ERS132380 | 血清16型 |
| LSS23 | 非临床株 | 英国 | 猪 | ERS132385 | 血清24型 |
| LSS30 | 非临床株 | 英国 | 猪 | ERS132392 | 血清31型 |
| LSS31 | 非临床株 | 英国 | 猪 | ERS132393 | 血清24型 |
| LSS32 | 非临床株 | 英国 | 猪 | ERS132394 | 血清24型 |
| LSS34 | 非临床株 | 英国 | 猪 | ERS132396 | 血清8型 |
| LSS36 | 未知 | 英国 | 猪 | ERS132398 | NCL7型 |
| LSS37 | 非临床株 | 英国 | 猪 | ERS132399 | 血清24型 |
| LSS39 | 非临床株 | 英国 | 猪 | ERS132401 | 血清31型 |
| LSS40 | 非临床株 | 英国 | 猪 | ERS132402 | 血清6型 |
| LSS41 | 非临床株 | 英国 | 猪 | ERS132403 | 血清21型 |
| LSS42 | 非临床株 | 英国 | 猪 | ERS132404 | 血清16型 |
| LSS44 | 非临床株 | 英国 | 猪 | ERS132406 | 血清15型 |
| LSS47 | 非临床株 | 英国 | 猪 | ERS132409 | 血清16型 |
| LSS48 | 非临床株 | 英国 | 猪 | ERS132410 | 血清31型 |
| LSS52 | 未知 | 英国 | 猪 | ERS132414 | 血清19型 |
| LSS53 | 非临床株 | 英国 | 猪 | ERS132415 | 血清21型 |
| LSS55 | 非临床株 | 英国 | 猪 | ERS132417 | 血清9型 |
| LSS58 | 非临床株 | 英国 | 猪 | ERS132420 | 血清31型 |
| LSS59 | 非临床株 | 英国 | 猪 | ERS132421 | 血清10型 |
| LSS64 | 非临床株 | 英国 | 猪 | ERS132426 | 血清21型 |
| LSS65 | 非临床株 | 英国 | 猪 | ERS132427 | 血清10型 |
| LSS66 | 未知 | 英国 | 猪 | ERS132428 | 血清9型 |
| LSS67 | 非临床株 | 英国 | 猪 | ERS132429 | 血清8型 |
| LSS68 | 非临床株 | 英国 | 猪 | ERS132430 | 血清8型 |
| LSS70 | 非临床株 | 英国 | 猪 | ERS132432 | 血清9型 |
| LSS75 | 非临床株 | 英国 | 猪 | ERS132437 | 血清31型 |
| LSS76 | 非临床株 | 英国 | 猪 | ERS132438 | 血清8型 |
| LSS79 | 非临床株 | 英国 | 猪 | ERS132441 | 血清15型 |
| LSS80 | 非临床株 | 英国 | 猪 | ERS132442 | 血清31型 |
| LSS81 | 非临床株 | 英国 | 猪 | ERS132443 | 血清15型 |
| LSS82 | 非临床株 | 英国 | 猪 | ERS132444 | 血清16型 |
| LSS88 | 非临床株 | 英国 | 猪 | ERS132450 | NCL3型 |
| LSS89 | 非临床株 | 英国 | 猪 | ERS132451 | 血清8型 |
| LSS90 | 非临床株 | 英国 | 猪 | ERS132452 | 血清31型 |
| LSS92 | 非临床株 | 英国 | 猪 | ERS132454 | 血清9型 |
| SS985 | 未知 | 英国 | 猪 | ERS132525 | NCL6型 |
首先查找cps基因簇的cpsA保守基因,以确定cps基因簇的起始位置,并通过cpsA上游的开放阅读框(open reading frame,ORF)确定该cps基因簇在基因组中的位置;然后查找cps基因簇3′端下游的保守基因(glf或aroA),提取从cpsA至glf或aroA之间的序列,即为该菌株的cps基因簇序列。
1.3 cps基因簇中各基因的分析及注释使用Artemis 16.0.0软件,检索GenBank (www.ncbi.nlm.nih.gov/GenBank),Clusters of Orthologous Groups (COG,www.ncbi.nlm.nih.gov/COG/)以及 Pfam (pfam.sanger.ac.uk)protein motif 数据库确定CDS并注释,参照之前的研究确定该基因编码产物所属同源群(homology group,HG)[7, 14]。cps基因的命名按照以往对血清2型cps基因的命名规则[16],按照5′~3′的基因顺序从A~Z编序,超出部分以A1~Z1编序。然后,根据wzy序列比对结果确定该菌株的cps型别:若某菌株的wzy序列与已知某血清型标准菌株的wzy序列比对结果的覆盖率及同源性均>50%,则认为该菌株属于此cps型别。Blastn程序和Mega 5.1软件用于比对基因的核苷酸序列,以发现基因内部的点突变。
1.4 数据的可视化处理使用Vector NTI 11.5软件及Adobe Illustrator CS4软件呈现cps基因簇的基因构成与组织方式;采用Artemis comparison tool(ACT)对cps基因簇的同源区进行分析和可视化处理[17],以呈现cps基因簇的序列相似性,直观反映cps基因簇之间的差异。
2 结果 2.1 cps型别分布41株血清未分型猪链球菌中,共有37株菌的cps型别属于33种已知血清型。其中,血清31型最多,共8株;其次为血清8型、16型,各5株;血清9型、24型,各4株;血清10型、15型、21型,各3株;血清6型、血清19型,各1株。有4株属于新cps型别,其中NCL3型、NCL4型、NCL6型、NCL7型各1株,见表 1。
2.2 已知血清型cps基因簇的描述及变异 2.2.1 血清9型携带血清9型cps基因簇的英国菌株LSS66、LSS70、LSS92与血清9型标准菌株22083(GenBank登记号:BR001006)相比,3′端的cps9O~cps9S(HG124~HG128)5个基因及整合酶基因 int9-1和转座酶基因tnp9-1发生了缺失,见图 1A。菌株LSS55与22083的cps基因簇同源性超过99%。
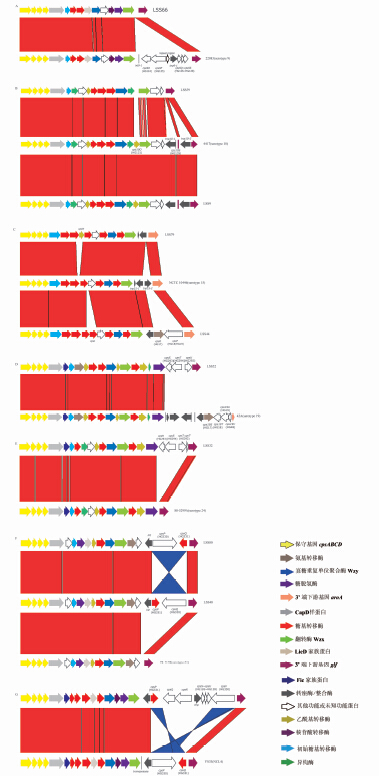
|
| 图 1 血清未分型菌株与其相应型别标准菌株的cps基因簇的比较 Figure 1 The comparison of cps loci of non-serotypeable strains and corresponding strains |
| |
携带血清10型cps基因簇的英国菌株LSS59、LSS65与血清10型标准菌株4417(GenBank登记号:BR001007)相比,cps10O(HG131)、cps10S(HG135)以及转座酶基因 tnp10-1、tnp10-2发生了缺失;菌株LSS9仅cps10S(HG135)缺失,见图 1B。
2.2.3 血清15型携带血清15型cps基因簇的英国菌株LSS79、LSS81与血清15型标准菌株NCTC 10446(GenBank登记号:AB737823)相比,缺失了转座酶基因tnp15-2,并在cps基因簇中部cps15G(HG33)和cps15H(HG77)之间插入一个乙酰基转移酶基因,此基因之前尚未被报道在猪链球菌cps基因簇中出现;菌株LSS44与标准菌株相比,3′区域缺失了转座酶基因tnp15-1、tnp15-2,插入了编码HG17~HG19的基因,并在cps基因簇中部cps15H(HG77)和cps15I(HG78)之间插入一个糖基转移酶基因,此基因之前也尚未报道在猪链球菌cps基因簇中出现,见表 2和图 1C。
| 菌株编号(1) | cps型别 | 基因 名称 | 假定产物 | 产物所属 同源群 | 来源 | 相似蛋白,所属菌种(GenBank登记号),覆盖率/同源性(%) |
| LSS79 | 血清15型 | cpsH | 乙酰基转移酶 | 未定 | 无 | 麦芽糖O-乙酰基转移酶,罗伊乳杆菌(CUR43586.1),96/69 |
| LSS44 | 血清15型 | cpsI | 糖基转移酶 | 未定 | 无 | alpha-1,2-岩藻糖转移酶,变黑普氏菌(WP_004362670.1),100/42 |
| cpsO | 天冬氨酸转移酶 | HG17 | 血清4、5、7、17、18、19、23型 | |||
| cpsP | 假定蛋白 | HG18、HG19 | 血清4、5、7、17、18、19、23型 | |||
| LSS52 LSS32 | 血清19型 血清24型 | cpsS(2) | 假定蛋白 | HG293 | NCL1-2、NCL1-6、NCL7、NCL8、Chz | |
| cpsT(2) | 假定蛋白 | HG294 | NCL1-2、NCL1-6、NCL7、NCL8、Chz | |||
| cpsU(2) | 假定蛋白 | HG292 | NCL1、NCL2-1、NCL2-2 | |||
| LSS80 LSS48 | 血清31型 | cpsP(3) | 假定蛋白 | HG330 | NCL4、NCL5 | |
| cpsQ(3) | 糖基转移酶 | HG331 | NCL4、NCL5 | |||
| LSS10 | NCL4 | cpsQ | 假定蛋白 | 未定 | 无 | 假定蛋白,马肠链球菌(WP_039693845.1),100/99 |
| cpsR | N-6 DNA甲基化酶 | 未定 | 无 | N-6 DNA甲基化酶,伊氏李斯特菌(WP_038406899.1),99/77 | ||
| cpsS | 假定蛋白 | HG126 | 血清9型 | |||
| cpsT | 假定蛋白 | HG127 | 血清9型 | |||
| cpsU | 假定蛋白 | HG128 | 血清9型 | |||
| 注:(1)表中仅列出代表菌株编号;(2)以菌株LSS52为准,菌株LSS32中基因名称分别为cpsR、cpsS、cpsT (cpsT′);(3)以菌株LSS80为准,菌株LSS48中基因名称分别为cpsQ 、cpsP。 | ||||||
携带血清19型cps基因簇的英国菌株LSS52与血清19型标准菌株42A(GenBank登记号:BR001009)相比,42A cps基因簇3′端的cps19S~cps19V(HG17~HG19、HG48)及全部的整合酶基因和转座酶基因被编码HG293、HG294、HG292的3个基因替换,见表 2和图 1D。
2.2.5 血清24型携带血清24型cps基因簇的英国菌株LSS32与血清24型标准菌株88-5299A(GenBank登记号:AB737829)相比,在cps基因簇3′端cps24Q(HG7)后插入编码HG293、HG294、HG292的3个基因,其中编码HG292的基因发生了部分重复,见表 2和图 1E。菌株LSS23、LSS31、LSS37与88-5299A的cps基因簇同源性为96%。
2.2.6 血清31型携带血清31型cps基因簇的英国菌株LSS39、LSS48、LSS58、LSS75、LSS80、LSS90与血清31型标准菌株92-4172(GenBank登记号:AB737835)相比,在cps基因簇3′端cps31O(HG277)后插入编码HG330、HG331的2个基因及1个整合酶基因int,其中,LSS48、LSS58、LSS75、LSS90的插入基因在cps基因簇内部又发生了倒转,见表 2和图 1F;菌株LSS11的 cpsE(HG5)基因发生了移码突变。菌株LSS30与92-4172的cps基因簇同源性超过99%。
除此之外,携带血清6型cps基因簇的英国菌株LSS40与血清6型标准菌株2524(GenBank登记号:AB737818),携带血清8型cps基因簇的英国菌株LSS34、LSS67、LSS68、LSS76、LSS89与血清8型标准菌株14636(GenBank登记号:BR001005),携带血清16型cps基因簇的英国菌株LSS8、LSS18、LSS42、LSS47、LSS82与血清16型标准菌株2726(GenBank登记号:BR001008),携带血清21型cps基因簇的英国菌株LSS41、LSS53、LSS64与血清21型标准菌株14A(GenBank登记号:AB737827)的cps基因簇相比,同源性均超过99%。
2.3 新cps型别cps基因簇的描述及变异4种新cps型别存在于这些菌株中。NCL3型菌株LSS88属于NCL3-1亚型[14],其与标准菌株YS27(GenBank登记号:KM972262)的cps基因簇同源性超过99%;NCL4型菌株LSS10与标准菌株YS33(GenBank登记号:KM972263)的cps基因簇覆盖率为77%,同源性为99%,LSS10与YS33相比,转座酶基因transposase发生了缺失,cpsP(HG330)和cpsQ(HG331)两个基因发生了倒转,且在这2个基因之间插入了2个未知功能的基因、1个转座酶基因tnp和编码HG126~HG128的3个基因,其中的2个未知功能基因此前未报道在猪链球菌cps基因簇中存在,见表 2和图 1G; NCL6型菌株SS985与标准菌株YS43(GenBank登记号:KM972270)的cps基因簇同源性超过99%;NCL7型菌株LSS36属于NCL7-1亚型[14],其与标准菌株YS57(GenBank登记号:KM972274)的cps基因簇同源性超过99%。
2.4 cps基因簇在基因组中的位置变化除了cps基因簇内部的变化外,部分菌株的cps基因簇在基因组中的位置也与之前的研究有所不同。在Okura等[7]的研究中,血清10型标准菌株4417和血清19型标准菌株42A均属于patternⅠ-a,但在本研究中,携带血清10型cps基因簇的英国菌株LSS9、LSS59、LSS65和携带血清19型cps基因簇的英国菌株LSS52均属于patternⅠ-b。除上述变化外,本研究中其他携带已知血清型cps基因簇菌株的cps基因簇在基因组中的位置均与Okura等[7]的研究一致;4株NCL型别的菌株与之前研究一致,均属于patternⅠ-b[14]。
3 讨论血清分型是病原菌流行病学研究的重要手段。猪链球菌的33种血清型是20世纪80年代被发现和命名的,随着研究的深入,全球范围内报道的猪链球菌血清未分型菌株也逐渐增多[6],对猪链球菌的流行病学研究造成了一定的困难。猪链球菌血清未分型菌株是由于其荚膜多糖抗原变异或荚膜缺失,导致与33种诊断血清中的2种及以上出现凝集反应(多凝)或与33种诊断血清都不凝集(不凝)的猪链球菌菌株。当前,高通量测序技术的发展极大地促进了猪链球菌cps基因簇序列的比较研究,为血清未分型菌株的cps基因簇序列研究提供了技术支撑。本研究中,笔者将GenBank公布的41株猪链球菌血清未分型菌株的cps基因簇从全基因组中提取出来并与其相应血清型标准菌株的cps序列进行比对分析,以期从基因水平上探究这些猪链球菌菌株血清不可分型的原因及其cps基因簇序列变异规律。
通过序列比对分析发现,绝大多数菌株(37/41,90.2%)的cps型别属于已知血清型,其中18株菌株与其相应血清型标准菌株的cps基因簇相比有较大程度的变异,主要体现在部分基因的插入、缺失、倒转、替换和移码突变。这些菌株cps基因簇的5′端相对保守,基因缺失主要发生在3′区域,发生缺失的基因编码产物主要有Ⅰ型限制修饰DNA的特异性结构域蛋白(HG124)、N-6 DNA甲基化酶(HG125)、假定蛋白(HG126~HG128、HG135)、乙酰基转移酶(HG131)。基因的插入可发生于基因簇的中部和(或)后部,这些插入的基因有的之前尚未报道在猪链球菌cps基因簇中出现,有的在其他血清型猪链球菌标准菌株的cps基因簇中存在(表 2),例如,携带血清15型cps基因簇的菌株LSS44的cps基因簇3′端插入了编码HG17~HG19的基因,这些基因在血清4、5、7、17、18、19、23型标准菌株的cps基因簇3′端正向或反向存在;携带血清31型cps基因簇的菌株LSS39、LSS48、LSS58、LSS75、LSS80、LSS90在cps基因簇3′端插入编码HG330和HG331的2个基因,这2个基因在NCL4和NCL5型标准菌株的cps基因簇3′端存在,而在33种血清型标准菌株的cps基因簇中均不存在[7, 14]。基因的插入、缺失和替换大多伴随着整合酶基因和转座酶基因的插入和缺失,这主要与整合酶和转座酶的功能有关。之前的研究表明,猪链球菌cps基因的替换和移码突变容易造成新的血清凝集表型[9, 18-20]。此外已有文献报道,在其他菌种中,基因簇外的某些基因也对抗原多糖有修饰和转录调控作用[21],可能是导致本研究中19株与其对应血清型标准菌株的cps基因簇序列高度同源的猪链球菌的荚膜多糖抗原发生变异,从而不可分型的机制之一。
本研究中,发现4株菌株携带新的cps基因簇,属于NCL型别。这些cps基因簇有各自型特异的wzx与wzy基因以及糖基/氨基/乙酰基转移酶基因,从而产生了新的荚膜抗原型别。此外,与中国属于相同NCL型别的分离株相比,本研究中的英国菌株的cps基因簇中有较多基因替换,提示中、英两国猪链球菌所处的微生态菌群环境存在差异。
猪链球菌cps基因簇可在种属间或跨种属水平转移。在水平转移过程中,会伴随出现cps中部分基因的交换、重组及序列变异,在cps基因簇中普遍存在的转座酶基因增加了上述基因事件出现的概率。不同地区、宿主来源的菌株处于不同的进化环境以及不同的选择压力下,导致cps基因簇序列的多样性及其在基因组中位置的变异。与此同时,猪链球菌cps基因簇外的某些基因也可能参与对荚膜多糖的修饰。这些机制最终导致猪链球菌荚膜多糖新的抗原型别不断涌现。这种抗原型别的多样性增强了猪链球菌的环境适应能力并有助于猪链球菌逃避机体的免疫清除,促进了猪链球菌种群的播散。综上所述,本研究表明,猪链球菌cps基因簇的序列变异、进化持续存在,传统的血清分型技术无法鉴别这些变化,亟需引入更灵敏的分子血清鉴定技术开展监测工作。
| [1] | Wertheim HFL, Nghia HDT, Taylor W, et al. Streptococcus suis:an emerging human pathogen[J]. Clin Infect Dis , 2009, 48 (5) : 617–625. DOI:10.1086/597768 |
| [2] | Yang WZ, Yu HJ, Jing HQ, et al. An outbreak of human Streptococcus suis serotype 2 infections presenting with toxic shock syndrome in Sichuan, China[J]. Chinese Journal of Epidemiology , 2006, 27 (3) : 185–191. (in Chinese) 杨维中, 余宏杰, 景怀琦, 等. 四川省一起伴中毒性休克综合征的人感染猪链球菌2型暴发[J]. 中华流行病学杂志 , 2006, 27 (3) : 185–191. |
| [3] | Ye CY, Zhu XP, Jing HQ, et al. Streptococcus suis sequence type 7 outbreak, Sichuan, China[J]. Emerg Infect Dis , 2006, 12 (8) : 1203–1208. DOI:10.3201/eid1708.060232 |
| [4] | Yu HJ, Jing HQ, Chen ZH, et al. Human Streptococcus suis outbreak, Sichuan, China[J]. Emerg Infect Dis , 2006, 12 (6) : 914–920. DOI:10.3201/eid1206.051194 |
| [5] | Segura M, Gottschalk M, Olivier M. Encapsulated Streptococcus suis inhibits activation of signaling pathways involved in phagocytosis[J]. Infect Immun , 2004, 72 (9) : 5322–5330. DOI:10.1128/IAI.72.9.5322-5330.2004 |
| [6] | Goyette-Desjardins G, Auger JP, Xu JG, et al. Streptococcus suis, an important pig pathogen and emerging zoonotic agent-an update on the worldwide distribution based on serotyping and sequence typing[J]. Emerg Microbes Infect , 2014, 3 (6) : e45. DOI:10.1038/emi.2014.45 |
| [7] | Okura M, Takamatsu D, Maruyama F, et al. Genetic analysis of capsular polysaccharide synthesis gene clusters from all serotypes of Streptococcus suis:potential mechanisms for generation of capsular variation[J]. Appl Environ Microbiol , 2013, 79 (8) : 2796–2806. DOI:10.1128/AEM.03742-12 |
| [8] | Bai XM, Liu ZJ, Ji SB, et al. Simultaneous detection of 33Streptococcus suis serotypes using the luminex xTAG® assayTM[J]. J Microbiol Methods , 2015, 117 : 95–99. DOI:10.1016/j.mimet.2015.07.018 |
| [9] | Liu ZJ, Zheng H, Gottschalk M, et al. Development of multiplex PCR assays for the identification of the 33 serotypes of Streptococcus suis[J]. PLoS One , 2013, 8 (8) : e72070. DOI:10.1371/journal.pone.0072070 |
| [10] | Okura M, Lachance C, Osaki M, et al. Development of a two-step multiplex PCR assay for typing of capsular polysaccharide synthesis gene clusters of Streptococcus suis[J]. J Clin Microbiol , 2014, 52 (5) : 1714–1719. DOI:10.1128/JCM.03411-13 |
| [11] | Liu ZJ, Bai XM, Ji SB, et al. Development of a multiplex PCR assay to identify seven new capsular gene loci of Streptococcus suis[J]. Chinese Journal of Zoonoses , 2014, 30 (4) : 337–346. (in Chinese) 刘志杰, 白雪梅, 纪少博, 等. 鉴定7种新型猪链球菌荚膜多糖基因型多重PCR方法的建立[J]. 中国人兽共患病学报 , 2014, 30 (4) : 337–346. |
| [12] | Kerdsin A, Akeda Y, Hatrongjit R, et al. Streptococcus suis serotyping by a new multiplex PCR[J]. J Med Microbiol , 2014, 63 (6) : 824–830. |
| [13] | Pan ZH, Ma JL, Dong WY, et al. Novel variant serotype of Streptococcus suis isolated from piglets with meningitis[J]. Appl Environ Microbiol , 2015, 81 (3) : 976–985. DOI:10.1128/AEM.02962-14 |
| [14] | Zheng H, Ji SB, Liu ZJ, et al. Eight novel capsular polysaccharide synthesis gene loci identified in nontypeable Streptococcus suis Isolates[J]. Appl Environ Microbiol , 2015, 81 (12) : 4111–4119. DOI:10.1128/AEM.00315-15 |
| [15] | Weinert LA, Chaudhuri RR, Wang JH, et al. Genomic signatures of human and animal disease in the zoonotic pathogen Streptococcus suis[J]. Nat Commun , 2015, 6 : 6740. DOI:10.1038/ncomms7740 |
| [16] | Smith HE, de Vries R, van't Slot R, et al. The cps locus of Streptococcus suis serotype 2:genetic determinant for the synthesis of sialic acid[J]. Microb Pathog , 2000, 29 (2) : 127–134. DOI:10.1006/mpat.2000.0372 |
| [17] | Carver TJ, Rutherford KM, Berriman M, et al. ACT:the Artemis comparison tool[J]. Bioinformatics , 2005, 21 (16) : 3422–3423. DOI:10.1093/bioinformatics/bti553 |
| [18] | Morona JK, Morona R, Paton JC. Comparative genetics of capsular polysaccharide biosynthesis in Streptococcus pneumoniae types belonging to serogroup 19[J]. J Bacteriol , 1999, 181 (17) : 5355–5364. |
| [19] | Yun KW, Cho EY, Choi EH, et al. Capsular polysaccharide gene diversity of pneumococcal serotypes 6A, 6B, 6C, and 6D[J]. Int J Med Microbiol , 2014, 304 (8) : 1109–1117. DOI:10.1016/j.ijmm.2014.08.004 |
| [20] | Lakkitjaroen N, Takamatsu D, Okura M, et al. Capsule loss or death:the position of mutations among capsule genes sways the destiny of Streptococcus suis[J]. FEMS Microbiol Lett , 2014, 354 (1) : 46–54. DOI:10.1111/fml.2014.354.issue-1 |
| [21] | Knirel YA, Wang JP, Luo X, et al. Genetic and structural identification of an O-acyltransferase gene (oacC) responsible for the 3/4-O-acetylation on rhamnoseⅢin Shigella flexneri serotype 6[J]. BMC Microbiol , 2014, 14 : 266. DOI:10.1186/s12866-014-0266-7 |
 2016, Vol. 31
2016, Vol. 31


