扩展功能
文章信息
- 刘丽云, 汪永禄, 金东, LANRuiTing, 原方芳, 叶长芸, 徐建国
- LIU Li-yun, WANG Yong-lu, JIN Dong, LAN Rui-ting, YUAN Fang-fang, YE Chang-yun, XU Jian-guo
- 安徽省马鞍山市分离的枸橼酸杆菌的分子分型
- Genetic diversity and molecular typing of Citrobacter in Maanshan, Anhui
- 疾病监测, 2016, 31(12): 995-1000
- Disease Surveillance, 2016, 31(12): 995-1000
- 10.3784/j.issn.1003-9961.2016.12.006
-
文章历史
- 收稿日期:2016-07-15
2. 浙江大学感染性疾病诊治协同创新中心, 浙江 杭州 310003;
3. 安徽省马鞍山市疾病预防与控制中心, 安徽 马鞍山 243000;
4. 澳大利亚悉尼市新南威尔士大学生物技术与生物分子学院, 澳大利亚 悉尼 南威尔士 00098G;
5. 北京科技大学化学与生物工程学院, 北京 100083
2. Collaborative Innovation Center for Diagnosis and Treatment of Infectious Diseases, Hangzhou 310003, Zhejiang, China;
3. Maanshan Prefecture Center for Disease Control and Prevention, Maanshan 243000, Anhui, China;
4. School of Biotechnology and Biomolecular Sciences, University of New South Wales, Sydney 00098 G, New South Wales, Australia;
5. School of Chemistry and Biological Engineering, Beijing University of Science and Technology, Beijing 100083, China
枸橼酸杆菌是革兰阴性杆菌,属肠杆菌科,通常寄居在土壤、水、食物、人类和动物肠道中[1-2],容易获得耐药基因而成为院内感染的常见耐药菌之一[3]。枸橼酸杆菌由于污染食物而引起散在感染和暴发[4-8],在发展中国家,是构成食源性疾病的主要原因之一,往往通过食物、水、人的手指污染的粪便传播[9]。此外,食品加工者不良的个人卫生习惯也是污染这些微生物的原因[10]。枸橼酸杆菌可以从通心面、炖番茄沙拉、鱼肉、槟榔汤、花生汤、红油、辣椒、豆子、木薯、山药、车前草和豆腐等中分离到[10]。水果由于不进行加工直接食用而成为消费者高风险食物[11]。Kouamá等[12]报道从阿比让地区5个不同市场出售的attieke食品中分离到包括枸橼酸杆菌在内的病原菌。Tassew等[13]报道,2009年4-8月在埃塞俄比亚金马市的24个酒店、5个肉铺和1个屠宰场165份食品样本中共分离到15份枸橼酸杆菌(9%)。
弗氏枸橼酸杆菌、布氏枸橼酸杆菌和杨氏枸橼酸杆菌,作为枸橼酸杆菌属的3个种,通常定植在人类和动物的肠道中[14]。弗氏枸橼酸杆菌是引起感染最常见的枸橼酸杆菌种[14-15]。布氏枸橼酸杆菌和杨氏枸橼酸杆菌很少引起感染[14-15]。弗氏枸橼酸杆菌的一些分离株通过获得毒力因子而引起人类食物中毒或腹泻[16],偶见黏附性弗氏枸橼酸杆菌[17]。有研究报道鉴定出1株强细胞毒性和强黏附性的弗氏枸橼酸杆菌,并且发现引起不同地区人群腹泻的弗氏枸橼酸杆菌不同[16]。布氏枸橼酸杆菌引起医院获得性感染和尿道感染而成为机会致病菌[17]。已经报道由于感染布氏枸橼酸杆菌而在腹膜透析患者中引起急性腹膜炎[18],在碎牛肉和猪肉产品中也分离到布氏枸橼酸杆菌[19-20]。
本研究对从安徽省马鞍山市的腹泻患者粪便、食品和食品加工者肛拭中分离到的弗氏枸橼酸杆菌、布氏枸橼酸杆菌和杨氏枸橼酸杆菌,通过脉冲场凝胶电泳(pulsed field gel electrophoresis,PFGE)和多位点序列分型(multi-locus sequence typing,MLST)进行分子分型,分析枸橼酸杆菌的传播特征。
1 材料和方法 1.1 枸橼酸杆菌分离株2007-2011年在安徽省马鞍山市进行的调查中,分离到62株枸橼酸杆菌,包括弗氏枸橼酸杆菌、布氏枸橼酸杆菌和杨氏枸橼酸杆菌。其中18株杨氏枸橼酸杆菌和2株弗氏枸橼酸杆菌分离自腹泻患者粪便标本。42株分离株(包括23株杨氏枸橼酸杆菌、11株弗氏枸橼酸杆菌和8株布氏枸橼酸杆菌)分离自食品(鸡肉、猪肉、鱼肉和蔬菜)和食品加工者肛拭。所有分离株都通过API 20E试纸条(bioMérieux,La Balme les Grottes,France)进行枸橼酸杆菌菌种的鉴定。所有分离株储存在甘油中,于-80 ℃保存。枸橼酸杆菌常规培养在37 ℃ LB液体或固体培养基中进行。
1.2 PFGE分析用于PFGE的枸橼酸杆菌基因组DNA的制备,使用的方法和条件是根据大肠埃希菌O157:H7菌的方法修改[17]。具体方法:琼脂糖胶块用45 U XbaⅠ酶切2 h,1%琼脂糖凝胶在CHEF DR Ⅲ系统(Bio-Rad,California)进行电泳。脉冲时间为2.2~54.2 s,电泳时间设置为18~19 h。PFGE图谱导入BioNumerics (Applied Math,Belgium)进行进一步分析和手动校准, < 20.5 kb的条带不进行分析。计算机分析完的所有带型需要人工校对。计算机和人眼无法区分的带型视为相同的带型。对每个PFGE带型进行统一的命名:CITX01.CNnnnn,CIT代表枸橼酸杆菌,X01代表XbaⅠ,CN代表中国,nnnn为序列号。
1.3 MLST方法按照MLST网站(http://shigatox.net/ecmlst/cgi-bin/da)提供的管家基因序列,选取7个管家基因(aspC、clpX、fadD、mdh、arcA、dnaG和lysP)进行PCR扩增。MLST的引物按照文献设计的引物,由生物工程(上海)股份有限公司合成[16]。1%的琼脂糖胶进行PCR产物的分离纯化。双向的双脱氧测序法进行DNA序列测定[生物工程(上海)股份有限公司]。7个管家基因相对差异度(表 1)算法按照参考文献[16, 21]完成。
| 基因名称 | 等位基因数 | 多态性(%) | 平均变异范围(%) |
| aspC | 43 | 106(21) | 7.45(0.19~13.84) |
| clpX | 46 | 83(15) | 5.03(0.18~14.74) |
| fadD | 53 | 120(25) | 8.25(0.21~16.12) |
| mdh | 45 | 83(15) | 5.44(0.18~10.20) |
| arcA | 33 | 48(11) | 4.68(0.23~23.68) |
| dnaG | 42 | 57(13) | 4.10(0.23~7.66) |
| lysP | 45 | 82(17) | 6.59(0.21~12.16) |
利用DNAStar v7.1中的SeqMan软件对PCR产物序列与数据库中的管家基因标准序列进行拼接和校正,校正后的7个管家基因序列同时上传至Citrobacter MLST数据库,确定菌株的等位基因型和序列类型。基于7个管家基因的串联序列,利用Mega 6.0软件Neighbor-joining法构建系统进化树,与以前报道的6个ST型(ST1~ST6)进行亲缘性比对,探究该62株枸橼酸杆菌的种属关系。沙门菌LT2作为外群。
对枸橼酸杆菌的ST型进行eBURST聚类分析,根据MLST单位点差异(SLV)或双位点差异(DLVs),ST序列等位基因至少有4个以上等位基因相似的参数原则进行eBURST聚类分析,可以将不同的ST型归为特定的ST序列群。ST基因型中SLV和DLVs最多的ST型可作为ST序列群的核心基因型,并用该核心基因型的ST型别号来命名ST序列群。
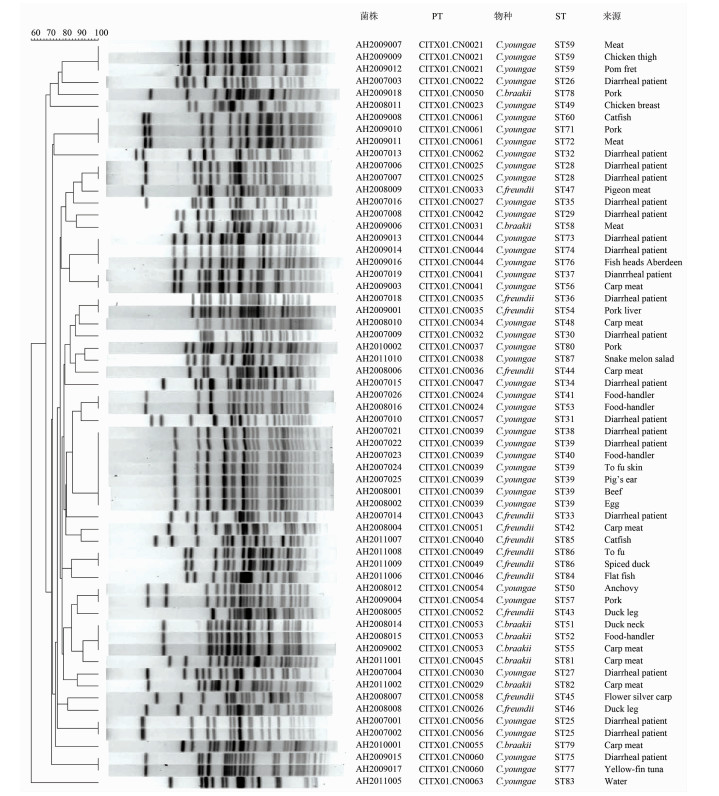
2 结果 2.1 枸橼酸杆菌PFGE分型及流行病学追溯为了对枸橼酸杆菌进行分子分型和流行病学追溯,使用PFGE方法分析62株枸橼酸杆菌,根据相似度评价值从53.9%到100.0%,将其分为43个不同的PFGE带型(PTs)(图 1)。CITX01.CN0039是优势PT,包括7株杨氏枸橼酸杆菌,其中5株是在2007年从1种食品、3例腹泻患者的粪便和1名食品加工者肛拭分离得到,另外2株是2008年从食品中分离得到。2007-2009年期间,从腹泻患者粪便分离的20株枸橼酸杆菌分为16个PTs,包括18株杨氏枸橼酸杆菌和2株弗氏枸橼酸杆菌。对于杨氏枸橼酸杆菌,有4个PTs,分别包括2株从腹泻患者粪便分离的菌株。从食品中分离到的枸橼酸杆菌分为6个PTs (4个是杨氏枸橼酸杆菌,另外2个分别是布氏枸橼酸杆菌和弗氏枸橼酸杆菌)。有4个PTs的菌株来源包括食品和腹泻患者粪便的分离株,它们是:CITX01.CN0044(3个杨氏枸橼酸杆菌分离株),CITX01.CN0041(2个杨氏枸橼酸杆菌分离株),CITX01.CN0060(2个杨氏枸橼酸杆菌分离株)和CITX01.CN0035 (2个弗氏枸橼酸杆菌分离株)。
|
| 图 1 62株枸橼酸杆菌的PFGE带型结果 Figure 1 Dendrogram of the 62 Citrobacter isolates based on PFGE patterns |
| |
用7个管家基因(aspC、clpX、fadD、mdh、arcA、dnaG和lysP)对62株枸橼酸杆菌进行MLST分析[16]。7个管家基因相对差异度见表 1。arcA是变异最大的基因,平均变异范围为4.68(0.23~23.68)。dnaG是最保守的基因,平均变异范围为4.10(0.23~7.66)。虽然fadD有最高的平均相对变异度8.25%,但最大变异度(16.12%)低于arcA。
62株枸橼酸杆菌分离株分成53个ST型别(STs)。其中41株杨氏枸橼酸杆菌分成32个STs,13个弗氏枸橼酸杆菌分成12个STs和8株布氏枸橼酸杆菌分成8个STs。ST39是优势ST型别,包括从4个食品样本和1个腹泻患者粪便分离的菌株;ST25和ST28分别分离自2例腹泻患者的粪便;ST59包括从不同的食品样本分离的菌株。
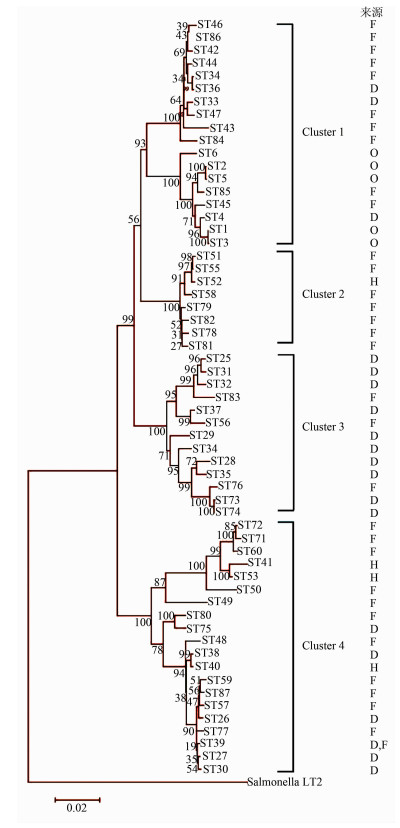
利用Mega 6.0软件Neighbor-joining法构建系统进化树(图 2),按照亲缘关系的远近,将该系统进化树分成4个集群(cluster)。集群1包括所有的弗氏枸橼酸杆菌,集群2包括所有的布氏枸橼酸杆菌,集群3和4包括所有的杨氏枸橼酸杆菌。
|
| 图 2 枸橼酸杆菌MLST数据之间建立的系统发育关系 Figure 2 Relationships among the Citrobacter isolates indicated by MLST data 注:邻接树显示STs。包括每个ST型别,来源F、D、H和O分别代表分离自食品、腹泻患者、食品加工者、动物和昆虫。 |
| |
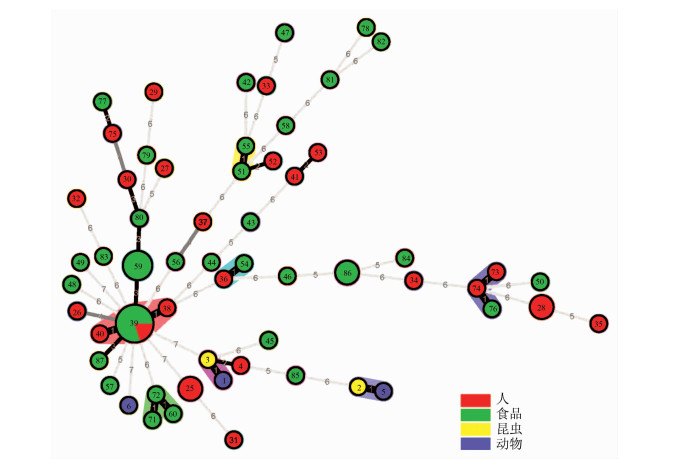
用eBURST V3软件对本次研究的59个STs进行了聚类分析,由图 3可见,主要包括7个ST序列群(ST序列群2、3、36、39、51、72和74)。每个ST序列群包括至少2个由于1个邻近等位基因的差别导致的不同ST型别。通过最小生成树来描述基于等位基因相关性的STs。本次研究菌株组成的5个ST序列群中,ST序列群39包括从腹泻患者粪便、食品加工者肛拭和食品中分离的枸橼酸杆菌组成的STs。ST序列群72和55包括从食品中分离的枸橼酸杆菌组成STs。ST序列群36和74包括从腹泻患者粪便和食品中分离的枸橼酸杆菌组成的STs。
|
| 图 3 基于MLST数据的枸橼酸杆菌的遗传关系 Figure 3 Genetic relationships of the Citrobacter isolates based on MLST 注:基于59个STs构造最小生成树。每个圆圈代表 1个ST。用不同颜色标注的阴影区代表不同的ST复合体。圆圈的大小与分离株的数量成正比。根据菌株来源将圆圈标有不同的颜色。 |
| |
用Bionumerics 7.5软件进行聚类分析,将62株枸橼酸杆菌分为43个不同的PTs。MLST分型方法将62株枸橼酸杆菌分离株分成53个ST型别(STs)。相同的STs (ST25、ST28、ST59和ST86)分别具有相同的PTs (CITX01.CN0056、CITX01.CN0025、CITX01.CN0021和CITX01.CN0049)。相同的PTs对应多个STs,其中4个PTs对应的STs分别有1个等位基因的差异。PFGE分型方法不能将枸橼酸杆菌的3个种分开。但是,MLST方法通过聚类分析可以将枸橼酸杆菌的3个种分开。
3 讨论弗氏枸橼酸杆菌是枸橼酸杆菌感染中最常见的种[14-15],通常与肠胃炎暴发和食源性疾病暴发相关[4-10]。1983年,美国疾病预防控制中心报道了哥伦比亚地区一起由弗氏枸橼酸杆菌污染食物引起的暴发腹泻事件[22]。1980年和1994年,在印度和德国分别报道了由于弗氏枸橼酸杆菌污染婴儿奶粉,而引起的新生儿监护室的婴儿集体腹泻[23-24]。Ibenyassine等[25]发现,在蔬菜中弗氏枸橼酸杆菌的检出率可达18.7%。
既往研究发现,包括12株从腹泻患者粪便、14株动物粪便和其他来源的共26株菌株分成6个STs (ST1~ST6)[16]。本研究弗氏枸橼酸杆菌分离株没有上述的ST型别,说明弗氏枸橼酸杆菌在不同的地区有较高的差异性。研究数据表明,相同的STs存在于从腹泻患者粪便、动物粪便和其他来源分离到的分离株[16]。在意大利医院暴发的产VIM的弗氏枸橼酸杆菌属于ST7,仅1个等位基因相同,但与中国分离株ST2和ST5进化关系较近[26]。
由全球分离株组成的枸橼酸杆菌MLST数据库包括28个STs,但均与我国分离菌株的ST型别不同,提示枸橼酸杆菌在不同国家之间变异度较高。
布氏枸橼酸杆菌通常存在水、土壤、食物、动物和人类的肠道中[19]。布氏枸橼酸杆菌是机会致病菌,已经在腹膜透析患者中分离到笔者从食物中分离到的8株布氏枸橼酸杆菌[18],但是在腹泻患者中没有分离到,需要进一步研究来了解布氏枸橼酸杆菌是否与腹泻相关。
杨氏枸橼酸杆菌很少引起感染。有杨氏枸橼酸杆菌引起腹膜炎的报道[27]。本研究中,18株杨氏枸橼酸杆菌从腹泻患者粪便中分离到,暗示杨氏枸橼酸杆菌是潜在的腹泻相关病原菌。
综上所述,本研究分离到的包括弗氏枸橼酸杆菌、布氏枸橼酸杆菌和杨氏枸橼酸杆菌在内的62株分离株分成43个PTs和53个STs。从腹泻患者粪便、食品和食品加工者肛拭分离到的分离株具有相同的PTs和STs。为了控制和预防枸橼酸杆菌引起的腹泻感染,加强食品加工环节和人群感染者中枸橼酸杆菌的监测是必要的。
作者贡献:
刘丽云 ORCID:0000-0002-6620-4183
汪永禄 ORCID:0000-0003-0937-6365
汪永禄:菌株分离,鉴定
金东:PFGE实验
原方芳:MLST实验和分析
叶长芸和徐建国:实验指导
刘丽云:实验设计,数据分析,文章撰写
| [1] | Lipsky BA, Hook EW, Smith AA, et al. Citrobacter infections in humans:experience at the seattle veterans administration medical center and a review of the literature[J]. Rev Infect Dis, 1980, 2 (5) : 746–760. DOI:10.1093/clinids/2.5.746 |
| [2] | Nada T, Baba H, Kawamura K, et al. A small outbreak of third generation cephem-resistant Citrobacter freundii infection on a surgical ward[J]. Jpn J Infect Dis, 2004, 57 (4) : 181–182. |
| [3] | Park YJ, Yu JK, Lee S, et al. Prevalence and diversity of qnr alleles in AmpC-producing Enterobacter cloacae, Enterobacter aerogenes, Citrobacter freundii and Serratia marcescens:a multicentre study from Korea[J]. J Antimicrob Chemother, 2007, 60 (4) : 868–871. DOI:10.1093/jac/dkm266 |
| [4] | Guerrant RL, Dickens MD, Wenzel RP, et al. Toxigenic bacterial diarrhea:Nursery outbreak involving multiple bacterial strains[J]. J Pediatr, 1976, 89 (6) : 885–891. DOI:10.1016/S0022-3476(76)80591-4 |
| [5] | Tschape H, Prager R, Streckel W, et al. Verotoxinogenic Citrobacter freundii associated with severe gastroenteritis and cases of haemolytic uraemic syndrome in a nursery school:green butter as the infection source[J]. Epidemiol Infect, 1995, 114 (3) : 441–450. DOI:10.1017/S0950268800052158 |
| [6] | Warner RD, Carr RW, McCleskey FK, et al. A large nontypical outbreak of Norwalk virus. Gastroenteritis associated with exposing celery to nonpotable water and with Citrobacter freundii[J]. Arch Intern Med, 1991, 151 (12) : 2419–2424. DOI:10.1001/archinte.1991.00400120061010 |
| [7] | Doulgeraki AI, Paramithiotis S, Nychas GJ. Characterization of the Enterobacteriaceae community that developed during storage of minced beef under aerobic or modified atmosphere packaging conditions[J]. Int J Food Microbiol, 2011, 145 (1) : 77–83. DOI:10.1016/j.ijfoodmicro.2010.11.030 |
| [8] | Giammanco GM, Aleo A, Guida I, et al. Molecular epidemiological survey of Citrobacter freundii misidentified as Cronobacter spp. (Enterobacter sakazakii) and enterobacter hormaechei isolated from powdered infant milk formula[J]. Foodborne Pathog Dis, 2011, 8 (4) : 517–525. DOI:10.1089/fpd.2010.0719 |
| [9] | Ifeadike C, Ironkwe OC, Adogu PO, et al. Prevalence and pattern of bacteria and intestinal parasites among food handlers in the Federal Capital Territory of Nigeria[J]. Niger Med J, 2012, 53 (3) : 166–171. DOI:10.4103/0300-1652.104389 |
| [10] | Settanni L, Miceli A, Francesca N, et al. Microbiological investigation of Raphanus sativus L. grown hydroponically in nutrient solutions contaminated with spoilage and pathogenic bacteria[J]. Int J Food Microbiol, 2013, 160 (3) : 344–352. DOI:10.1016/j.ijfoodmicro.2012.11.011 |
| [11] | Saba CK, Gonzalez-Zorn B. Microbial food safety in Ghana:a meta-analysis[J]. J Infect Dev Ctries, 2012, 6 (12) : 828–835. |
| [12] | Kouamá AK, Djeni TN, N'Guessan FK, et al. Postprocessing microflora of commercial attieke (a fermented cassava product) produced in the south of Cote d'Ivoire[J]. Lett Appl Microbiol, 2013, 56 (1) : 44–50. DOI:10.1111/lam.2012.56.issue-1 |
| [13] | Tassew H, Abdissa A, Beyene G, et al. Microbial flora and food borne pathogens on minced meat and their susceptibility to antimicrobial agents[J]. Ethiop J Health Sci, 2010, 20 (3) : 137–143. |
| [14] | Samonis G, Karageorgopoulos DE, Kofteridis DP, et al. Citrobacter infections in a general hospital:characteristics and outcomes[J]. Eur J Clin Microbiol Infect Dis, 2009, 28 (1) : 61–68. DOI:10.1007/s10096-008-0598-z |
| [15] | Mohanty S, Singhal R, Sood S, et al. Citrobacter infections in a tertiary care hospital in Northern India[J]. J Infect, 2007, 54 (1) : 58–64. DOI:10.1016/j.jinf.2006.01.015 |
| [16] | Bai L, Xia SL, Lan RT, et al. Isolation and characterization of cytotoxic, aggregative Citrobacter freundii[J]. PLoS One, 2012, 7 (3) : e33054. DOI:10.1371/journal.pone.0033054 |
| [17] | Gupta N, Yadav A, Choudhary U, et al. Citrobacter bacteremia in a tertiary care hospital[J]. Scand J Infect Dis, 2003, 35 (10) : 765–768. DOI:10.1080/00365540310016376 |
| [18] | Chao CT, Lee SY, Yang WS, et al. Citrobacter peritoneal dialysis peritonitis:rare occurrence with poor outcomes[J]. Int J Med, 2013, 10 (9) : 1092–1098. |
| [19] | Basra P, Koziol A, Wong A, et al. Complete genome sequences of Citrobacter braakii strains GTA-CB01 and GTA-CB04, isolated from ground beef[J]. Genome Announc, 2015, 3 (1) : 1307–1314. |
| [20] | Kwak HL, Han SK, Park S, et al. Development of a rapid and accurate identification method for citrobacter species isolated from pork products using a matrix-assisted laser-desorption ionization time-of-flight mass spectrometry (MALDI-TOF MS)[J]. J Microbiol Biotechnol, 2015, 25 (9) : 1537–1541. DOI:10.4014/jmb.1503.03071 |
| [21] | Ch'ng SL, Octavia S, Xia Q, et al. Population structure and evolution of pathogenicity of Yersinia pseudotuberculosis[J]. Appl Environ Microbiol, 2011, 77 (3) : 768–775. DOI:10.1128/AEM.01993-10 |
| [22] | Anonymus. Gastrointestinal Illness Associated with Imported Brie Cheese-District of Columbia[J]. Centers for Disease Control (CDC).MMWR Morb Mortal Wkly Rep, 1983, 32 (41) : 533. |
| [23] | Pardia SN, Verma IC, Deb M, et al. An outbreak of diarrhea due to Citrobacter freundii in a neonatal special care nursery[J]. Indian J Pediatr, 1980, 22 (47) : 81–84. |
| [24] | Thurm V, Gericke B. Identification of infant food as a vehicle in a nosocomial outbreak of Citrobacter freundii:epidemiological subtyping by allozyme, whole-cell protein and antibiotic resistance[J]. J Appl Bacteriol, 1994, 76 (6) : 553–558. DOI:10.1111/jam.1994.76.issue-6 |
| [25] | Ibenyassine K, Mhand RA, Karamoko Y, et al. Bacterial pathogens recovered from vegetables irrigated by wastewater in Morocco[J]. J Environ Health, 2007, 69 (10) : 47–51. |
| [26] | Gaibani P, Ambretti S, Farruggia P, et al. Outbreak of Citrobacter freundii carrying VIM-1 in an Italian Hospital, identified during the carbapenemases screening actions, June 2012[J]. Int J Infect Dis, 2013, 17 (9) : e714–e717. DOI:10.1016/j.ijid.2013.02.007 |
| [27] | Chen KJ, Chen TH, Sue YM. Citrobacter youngae and pantoea agglomerans peritonitis in a peritoneal dialysis patient[J]. Perit Dial Int, 2013, 33 (3) : 336–337. DOI:10.3747/pdi.2012.00151 |
 2016, Vol. 31
2016, Vol. 31


