扩展功能
文章信息
- 陈海霞, 贾俊楠, 李卫民, 高基民
- CHEN Hai-xia, JIA Jun-nan, LI Wei-min, GAO Ji-min
- 结核分枝杆菌单核苷酸多态性特征的分析
- Characteristics of single nucleotide polymorphism of Mycobacterium tuberculosis
- 疾病监测, 2017, 32(4): 332-336
- Disease Surveillance, 2017, 32(4): 332-336
- 10.3784/j.issn.1003-9961.2017.04.018
-
文章历史
- 收稿日期:2016-12-01
2. 首都医科大学附属北京胸科医院国家结核病临床实验室, 北京 101149;
3. 北京市结核病胸部肿瘤研究所, 北京 101149
2. National Tuberculosis Clinical Laboratory of China, Beijing Chest Hospital Affiliatedto Capital Medical University, Beijing 101149, China;
3. Beijing Tuberculosis and Thoracic Tumor Research Institute, Beijing 101149, China
结核病主要由结核分枝杆菌 (Mycobacterium tuberculosis,MTB) 引起。近年来,耐药结核病特别是耐多药结核病 (multidrug-resistant TB,MDR-TB)、广泛耐药结核病 (extensively-resistant TB,XDR-TB) 的出现使全球结核病控制形式变得更加严峻[1]。当前的防控以及诊断治疗手段都亟须注入高效新技术以改善当前的现状。
随着测序技术的发展以及价格下降,越来越多的研究者倾向于将全基因组测序技术广泛应用于MTB研究中,因此,可用于分析的全基因组序列越来越多。已有研究表明其应用于分子流行病学调查,能更精确地追踪传染源、查证传播途径[2-3],也能够准确区分内源性复发和外源性感染[4],并提高发现耐药基因和突变位点的效率。Zhang等[5]对161株中国分离株进行基因组比较发现Rv0026等72个新基因、Rv0010 c-Rv0011c等28个基因间隔区 (intergenic region,IGR) 以及11个非同义单核苷酸多态性位点 (single nucleotide plymorphism,SNP) 和10个IGR SNP可能与耐药相关。基因组是MTB传播、致病、耐药和进化的遗传基础,对其全面认识是MTB研究的重要组成。而目前多项研究结果表明MTB耐药[5-6]、毒力[7]、致病性[8]、进化[9]可能与其基因组内的SNP变化相关。Coll等[10]和Homolka等[11]研究发现SNP位点可以作为一项分辨率高且稳定性好的分型技术,同时也能够用于系统发育及进化相关分析。因此本研究对2 372株菌株的全基因组序列进行分析,系统地分析MTB SNP位点特征,从而为结核病的预防及治疗提供科学依据。
1 材料与方法 1.1 材料本研究于美国国立生物技术信息中心 (NCBI) 和欧洲核酸数据库 (ENA) 中下载来自于全球代表性2 726株菌株全基因组序列。部分测序样本由于测序误差或者菌株污染等原因,造成序列GC含量偏离正常值以及回帖率过低,则按照GC含量 (<60%或>70%的菌株舍弃)、回帖率 < 85%的标准将序列进行过滤。最终2 372株MTB的全基因组序列纳入本研究,见表 1。
| 序列号(1) | 过滤前样本(2)(株) | 过滤后样本(3)(株) | 菌株来源 |
| SRA065095 | 161 | 161 | 中国 |
| SRA050212 | 26 | 11 | 中国 |
| SRP029424 | 32 | 31 | 中国 |
| ERP000520 | 51 | 50 | 乌干达 |
| ERP000111 | 213 | 133 | 荷兰 |
| ERP000436 | 358 | 305 | 马拉维 |
| ERP001885 | 7 | 5 | 吉布提 |
| ERP002611 | 56 | 52 | 葡萄牙 |
| SRP002589 | 36 | 35 | 加拿大 |
| PRJEB11653 | 127 | 94 | 吉布提 |
| ERP000192 | 982 | 851 | 俄罗斯 |
| ERP000276 | 390 | 387 | 英国 |
| PRJEB9201 | 62 | 59 | 埃塞俄比亚 |
| ERP001731 | 225 | 198 | 全球代表性菌株 (中美洲、中亚、东非、东亚、欧洲、东欧洲、中非、北非、北美洲、南非、南美洲、西亚、西非、南东亚) |
| 总菌株数 | 2 725 | 2 372 | |
| 注:(1) 从NCBI和ENA数据库中获取菌株全基因组序列的序列号;(2) 数据库获取原始测序的样本;(3) 将数据库获取原始测序的样本按照质控相关要求处理后得到的样本。 | |||
用BWA v 0.7.1软件[12]将菌株的测序文件回帖到结核杆菌参考基因组H37Rv上,将比对后的序列采用samtools v 1.3[13]和picard-tools v 1.112软件对生成的Sam文件进行Bam转换、排序以及去重处理。采用变异鉴定软件Varscan v 2.4.0[14]根据经过处理得到的Bam文件进行SNP鉴定,将基因组比对质量<30或者碱基质量<27的SNP位点舍去。为提高SNP位点可信度,降低假阳性,对SNP位点按照下列标准进一步进行过滤[15]:(1) 选取最小等位基因突变频率>75%且有>20条reads支持该位点的SNP位点;(2) 去除SNP miss call>15%的菌株;(3) 去除分布在MTB PE/PPE家族SNP位点。
1.2.2 优化SNP集合以M.canettii菌株为外群菌株,将2 372株测序菌株用初步得到的SNP位点作为靶标采用最大似然法软件RAxML v 8.2.8[16]在GTRGAMMA模型下构建系统进化树。根据系统进化树每个分支将分支内菌株和剩余菌株分别作为一个群体,采用Genepop v 4.5.1软件[17]计算每个SNP位点的遗传分化系数 (Fst),选用Fst>0.99的SNP位点作为谱系相关SNP位点[10]。将谱系相关SNP位点用SnpEff v 4.3c软件[18]进行注释,选取关键基因上的同义突变SNP位点[19-20],最终得到SNP优化集合。
2 结果 2.1 SNP总集合通过初步将2 347株菌株与参考基因组H37Rv比对,筛选得到107 654个SNP位点,以M.canettii菌株为外群菌株,构建的系统进化树将2 347株MTB明确地划分为7个大分支,见图 1,即分别对应7个谱系 (Lineage1、Lineage2、Lineage3、Lineage4、Lineage5、Lineage6、Lineage7) 以及69个亚型。Lineage1、Lineage2、Lineage3、Lineage4、Lineage5、Lineage6、Lineage7分别发现16 830、31 017、11 312、49 406、4 176、4 380、3 188个SNP位点。且Lineage2、Lineage3、Lineage4的SNP位点明显高于Lineage1、Lineage5、Lineage6、Lineage7。
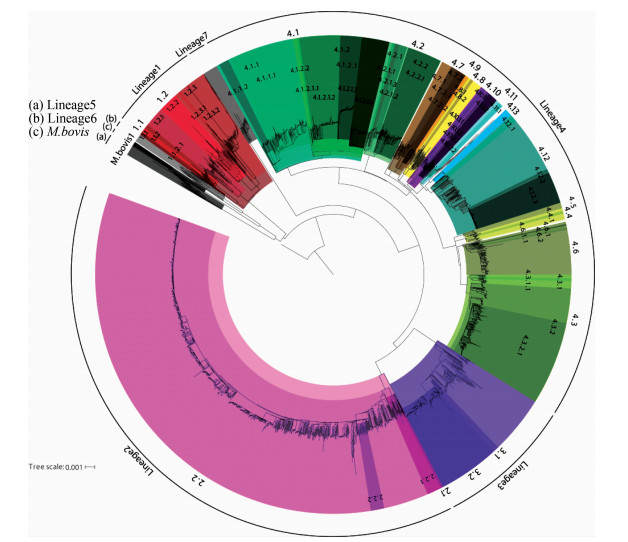
|
| 图 1 全球2 347株结核分枝杆菌菌株系统进化树-107 654个SNP位点 Figure 1 Phylogenetic tree of 2 347 isolates-107 654 SNP sites 注:不同颜色分支代表不同谱系。 |
| |
按照上述方法SNP集合优化,最终285个SNP位点可作为系统进化及分型位点。以该套SNP分型位点作为分型靶标,M.canettii菌株为外群菌株,通过最大似然法得到系统进化树,见图 2。该系统进化树与图 1结果基本相似,将2 347株MTB明确地划分为7个大分支,67个亚谱系。分辨力稍低于107 654个SNPs分型效果。
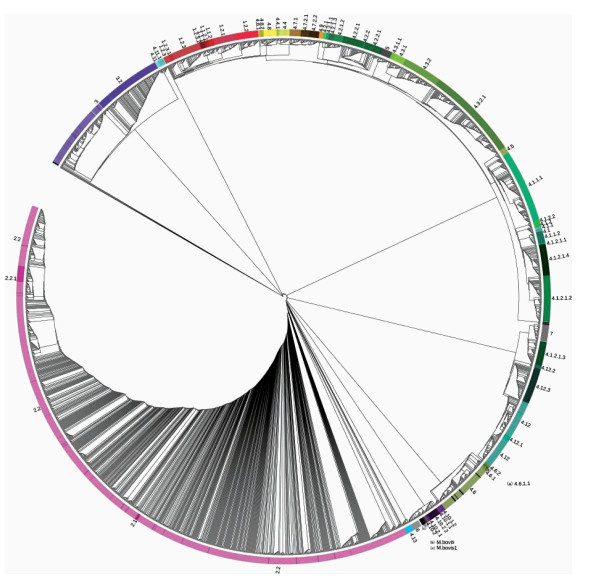
|
| 图 2 全球2 347株结核分枝杆菌菌株系统进化树-285个SNP位点 Figure 2 Phylogenetic tree of 2 347 isolates-285 SNP sites 注:不同颜色分支代表不同谱系。 |
| |
近年来,全基因组测序技术已经逐渐应用到MTB多个方面的研究。因此可获得越来越多的全基因组序列,而且目前多项研究结果表明MTB耐药、毒力、致病性与其基因组内SNP变化相关,SNP位点可以分为同义突变和导致氨基酸改变的非同义突变,这2类突变为研究细菌进化提供了极其有用的信息。非同义突变可能因内在或外部环境的选择压力带来趋同进化的结果。如MTB的耐药往往与基因组上特定药物靶标的基因发生突变或缺失相关,因此在耐药基因关键位点上的非同义突变可能改变菌株的耐药性。相反,同义突变不会带来氨基酸序列的改变,这种突变被视为中性。当同义突变发生在管家基因时,就成为研究菌株间遗传和进化关系的关键位点。因此SNP不仅能对MTB进行基因分型,也是研究种系发生的有力工具。因此本研究下载近10年来共2 347株MTB全基因组序列,系统地探讨MTB的SNP特征。
本研究将2 347株菌株全基因组序列与参考基因组H37Rv比较,发现107 654个SNP位点,通过构建系统进化树发现将2 347株菌株划分为7个谱系,与之前的全基因序列数据所构建的系统进化树结果一致[20]。同时发现不同谱系SNP位点数量不同,Lineage2、Lineage3、Lineage4的SNP位点明显高于Lineage1、Lineage5、Lineage6、Lineage7。已有研究表明Lineage2、Lineage3、Lineage4属于现代谱系,Lineage1、Lineage5、Lineage6、Lineage 7属于古老谱系,现代谱系的多态性高于古老谱系[21],本研究结果与其相一致。另外将优化后发现的285个SNP位点为谱系定义靶标,通过构建系统进化树发现,该SNP位点集合也能够准确地将2 347株菌鉴别为67亚谱系,与107 654个SNP位点分型效果几乎一致,因此,可将其组合成一套用于MTB分型靶标。而且之前多项研究表明插入序列6110-限制性片段长度多态性 (IS 6110-Restricted fragment longevity polymorphism,IS6110FLP)、间隔区寡核苷酸分型 (Spoligotyping)、分枝杆菌散在重复单位-数目可变串联重复序列 (MIRU-VNTR) 等分型技术所用的分型靶标具有较高的趋同进化速率[22],由于SNP在结核菌基因组中属于唯一且不可逆转的事件,具有稳定的遗传性,将其作为分型技术的靶标则克服了其他传统分型技术所带来的同源异质性 (homplasy) 缺点。与长序列片段多态性 (Large Sequence Polymorphism,LSP) 相比较,该套分型靶标的分辨率更高。另外与国外研究者所筛选的SNP分型靶标相比,如Coll等[10]研究发现62个SNP位点可以将MTB一共划分为55个亚谱系,Homolka等[11]用71个SNP位点将MTB划分为10个亚谱系,由于本研究中所涉及的菌株数量多,覆盖全球多个地区,因此该套SNP分型靶标的分辨率更高及更具代表性,因涉及的SNP位点较多,可能导致花费更高。因此,可以在该套SNP基础上进行更深入的研究,减少SNP位点并同时保证分辨率,从而将该技术应用到实际工作当中,本研究为其提供了科学依据。另一方面,Gardy等[2]针对在加拿大不列颠哥伦比亚地区经VNTR成簇调查确定为暴发流行的32株结核分枝杆菌,将全基因组数据经系统进化分析发现,分别属于2个有遗传进化关系的分支,并且根据进化的时间先后顺序,重新确定了其传播途径。可见全基因组测序技术是目前研究其时间和空间传播机制最好的工具,但其价格昂贵。而本研究中基于系统发育的285 SNP位点也可以用MTB的系统进化研究,进而能够精准推算MTB的演化历史,因此也能够实现从时间和空间掌握MTB传播特征。
该套分型靶标具有较高的分辨率及稳定性可以用于MTB的分子流行病学调查,准确鉴别传播发生的路径、顺序及是否存在多个传播源,为制定结核病的防控策略提供科学依据。另一方面,这285 SNP位点同时具有系统进化意义,可以用于作为研究种系发生的有力工具,研究MTB的群体遗传学特征。
作者贡献:
陈海霞 ORCID:0000-0001-8553-1094
陈海霞:撰写论文和数据分析
贾俊楠:数据分析
李卫民:项目设计
高基民:项目设计
| [1] | Gandhi NR, Nunn P, Dheda K, et al. Multidrug-resistant and extensively drug-resistant tuberculosis:a threat to global control of tuberculosis[J]. Lancet, 2010, 375(9728): 1830–1843. DOI:10.1016/S0140-6736(10)60410-2 |
| [2] | Gardy JL, Johnston JC, Ho Sui SJ, et al. Whole-genome sequencing and social-network analysis of a tuberculosis outbreak[J]. N Engl J Med, 2011, 364(8): 730–739. DOI:10.1056/NEJMoa1003176 |
| [3] | Luo T, Yang CG, Peng Y, et al. Whole-genome sequencing to detect recent transmission of Mycobacterium tuberculosis in settings with a high burden of tuberculosis[J]. Tuberculosis, 2014, 94(4): 434–440. DOI:10.1016/j.tube.2014.04.005 |
| [4] | Bryant JM, Harris SR, Parkhill J, et al. Whole-genome sequencing to establish relapse or re-infection with Mycobacterium tuberculosis:a retrospective observational study[J]. Lancet Respir Med, 2013, 1(10): 786–792. DOI:10.1016/S2213-2600(13)70231-5 |
| [5] | Zhang HT, Li DF, Zhao LL, et al. Genome sequencing of 161Mycobacterium tuberculosis isolates from China identifies genes and intergenic regions associated with drug resistance[J]. Nat Genet, 2013, 45(10): 1255–1260. DOI:10.1038/ng.2735 |
| [6] | Riska PF, Jacobs WR Jr, Alland D. Molecular determinants of drug resistance in tuberculosis[J]. Int J Tuberc Lung Dis, 2000, 4(2 Suppl 1): S4–S10. |
| [7] | van Pittius NCG, Sampson SL, Lee H, et al. Evolution and expansion of the Mycobacterium tuberculosis PE and PPE multigene families and their association with the duplication of the ESAT-6(esx) gene cluster regions[J]. BMC Evol Biol, 2006, 6: 95. DOI:10.1186/1471-2148-6-95 |
| [8] | Lipsitch M, O'Hagan JJ. Patterns of antigenic diversity and the mechanisms that maintain them[J]. J R Soc Interface, 2007, 4(16): 787–802. DOI:10.1098/rsif.2007.0229 |
| [9] | Stucki D, Gagneux S. Single nucleotide polymorphisms in Mycobacterium tuberculosis and the need for a curated database[J]. Tuberculosis, 2013, 93(1): 30–39. DOI:10.1016/j.tube.2012.11.002 |
| [10] | Coll F, McNerney R, Guerra-Assunção JA, et al. A robust SNP barcode for typing Mycobacterium tuberculosis complex strains[J]. Nat Commun, 2014, 5: 4812. DOI:10.1038/ncomms5812 |
| [11] | Homolka S, Projahn M, Feuerriegel S, et al. High resolution discrimination of clinical Mycobacterium tuberculosis complex strains based on single nucleotide polymorphisms[J]. PLoS One, 2012, 7(7): e39855. DOI:10.1371/journal.pone.0039855 |
| [12] | Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform[J]. Bioinformatics, 2009, 25(14): 1754–1760. DOI:10.1093/bioinformatics/btp324 |
| [13] | Li H, Handsaker B, Wysoker A, et al. The sequence alignment/map format and SAMtools[J]. Bioinformatics, 2009, 25(16): 2078–2079. DOI:10.1093/bioinformatics/btp352 |
| [14] | Koboldt DC, Chen K, Wylie T, et al. VarScan:variant detection in massively parallel sequencing of individual and pooled samples[J]. Bioinformatics, 2009, 25(17): 2283–2285. DOI:10.1093/bioinformatics/btp373 |
| [15] | Guerra-Assunção JA, Crampin AC, Houben RMGJ, et al. .Large-scale whole genome sequencing of M.tuberculosis provides insights into transmission in a high prevalence area[J]. eLife, 2015, 4: e05166. |
| [16] | Stamatakis A. RAxML version 8:a tool for phylogenetic analysis and post-analysis of large phylogenies[J]. Bioinformatics, 2014, 30(9): 1312–1313. DOI:10.1093/bioinformatics/btu033 |
| [17] | Rousset F. genepop'007:a complete re-implementation of the genepop software for Windows and Linux[J]. Mol Ecol Resour, 2008, 8(1): 103–106. DOI:10.1111/j.1471-8286.2007.01931.x |
| [18] | Cingolani P, Platts A, Wang LL, et al. A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff:SNPs in the genome of Drosophila melanogaster strain w1118; iso-2;iso-3[J]. Fly, 2012, 6(2): 80–92. DOI:10.4161/fly.19695 |
| [19] | Sassetti CM, Boyd DH, Rubin EJ. Genes required for mycobacterial growth defined by high density mutagenesis[J]. Mol Microbiol, 2003, 48(1): 77–84. DOI:10.1046/j.1365-2958.2003.03425.x |
| [20] | Sassetti CM, Rubin EJ. Genetic requirements for mycobacterial survival during infection[J]. Proc Natl Acad Sci USA, 2003, 100(22): 12989–12994. DOI:10.1073/pnas.2134250100 |
| [21] | Brosch R, Gordon SV, Marmiesse M, et al. A new evolutionary scenario for the Mycobacterium tuberculosis complex[J]. Proc Natl Acad Sci USA, 2002, 99(6): 3684–3689. DOI:10.1073/pnas.052548299 |
| [22] | Comas I, Homolka S, Niemann S, et al. Genotyping of genetically monomorphic bacteria:DNA sequencing in Mycobacterium tuberculosis highlights the limitations of current methodologies[J]. PLoS One, 2009, 4(11): e7815. DOI:10.1371/journal.pone.0007815 |
 2017, Vol. 32
2017, Vol. 32


