扩展功能
文章信息
- 方斌, 余晓, 李翔, 叶国军, 刘琳琳
- FANG Bin, YU Xiao, LI Xiang, YE Guo-jun, LIU Lin-lin
- 2010-2015年湖北省H3N2亚型流感病毒抗原漂移分析
- Analysis of antigen drift of influenza A (H3N2) virus in Hubei, 2010-2015
- 疾病监测, 2017, 32(5): 387-393
- Disease Surveillance, 2017, 32(5): 387-393
- 10.3784/j.issn.1003-9961.2017.05.009
-
文章历史
- 收稿日期:2016-11-07
自1968年H3N2亚型流感病毒大流行以来,该病毒通过片段重组、抗原漂移和抗原转换逃逸宿主免疫,在全球不断变异引起新的流行,每年造成的死亡人数多达25万~50万[1]。H3N2亚型流感病毒的抗原漂移和致死率更强[1-12],该病毒的新变异给疫苗株的筛选带来极大挑战,使得过去匹配的疫苗效力减少[3]。
2014-2015年流感流行的主要特征是H3N2亚型流感病毒的抗原漂移造成2013-2014年3C.1簇疫苗株A/Texas/50/2012的不匹配[4],这些不匹配的H3N2亚型流行流感主要属于3C.2a簇病毒,全球流感监测网络表明3C.2a簇病毒很难通过血凝抑制试验(hemagglutination inhibition test,HI)准确鉴定,加拿大只有不到15%的3C.2a簇病毒被鉴定[5]。通过与A/Texas/50/2012疫苗株的序列比较分析表明3C.2a簇病毒进化簇的代表突变子为F159Y和K160T,而158~160氨基酸位点属于血凝素(hemagglutinin,HA)蛋白顶端暴露区域的抗原决定簇B表位,仅3C.2a簇病毒拥有的F159Y和K160T突变能够修饰病毒抗原表位和阻碍抗体结合免疫抗原决定簇B[6-7]。该突变的研究有助于3C.2a簇病毒抗原性相关问题的解释和疫苗保护效果的评价[5]。
我国对H3N2亚型流感3C.2a簇病毒的进化情况进行过报道[8-10],本文尝试分析2010-2015年湖北省H3N2亚型流感病毒流行和进化树分布情况,通过H3N2亚型流感3C.2a和3C.3a簇病毒在3个主要抗原决定簇上氨基酸突变位点[11]和三维结构的对比分析,了解湖北省H3N2亚型流感3C.2a簇病毒的进化和突变情况,氨基酸突变位点对3C.2a簇病毒HA蛋白主要抗原决定簇的结构影响,为流感病毒监测和疫苗筛选工作提供科学的理论支持。
1 材料与方法 1.1 湖北省H3N2亚型流感病毒时间分布2010-2015年湖北省H3N2亚型流感病毒核酸阳性数、阳性率和毒株分离数源于中国流感监测信息系统(http://10.249.1.170/UVSSERVER2.0/)。将流感病毒核酸检测阳性率超过流行季峰值的30%判定为流行高峰[12-13],对6年H3N2亚型流感病毒流行季划分流行高峰,分析其流行高峰时间段、各时间段内病毒核酸检测阳性数和毒株分离数。
1.2 毒株核酸提取、HA和神经氨酸酶(neuraminid-ase,NA)基因扩增及测序采用QIAGEN公司BIO ROBOT EZ1核酸提取仪及其配套EZ1 virus mini kit v2.0试剂盒提取各毒株核酸。测序引物序列由国家流感中心提供[10],上海生工公司合成。采用QIAGEN公司OneStep RT-PCR Kit试剂盒进行聚合酶链式反应(polymease chain reaction,PCR)扩增,反应条件:45 ℃,45 min,1个循环;94 ℃预变性2 min,94 ℃,30 s,55 ℃,1 min,68 ℃,2 min,扩增40个循环后,68 ℃延伸7 min。PCR产物凝胶电泳阳性样本送北京华大基因公司测序。
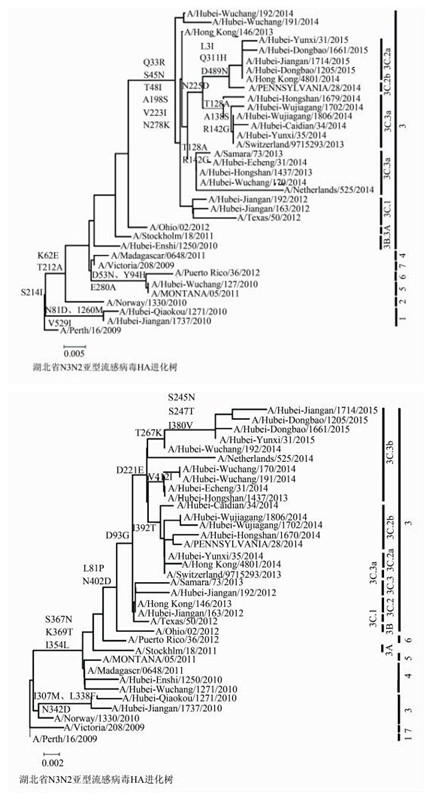
1.3 HA和NA氨基酸序列来源和进化树分析选取的毒株通过国家流感中心复核且正确,时间分布在2010-2015年各流行季且来源于湖北省流感监测网络,共选取20株H3N2亚型流感毒株,其中7株由湖北省疾病预防控制中心(CDC)测序,13株由国家流感中心测序,均上传至全球流感共享数据库(global initiative on sharing all influenza data,GISAID)共享其HA和NA氨基酸序列。HA和NA进化分支参考株参照欧洲CDC(http://ecdc.europa.eu/)的流感进化分析报告[14-15],其中A/Perth/16/2009、A/Norway/1330/2010、A/Madagascar/0648/2011、A/MONTANA/05/2011、A/Puerto Rico/36/2012、A/Victoria/208/2009参考株分别代表进化簇1、2、4~7;A/Stockholm/18/2011、A/Ohio/02/2012、A/Texas/50/2012、A/Hong Kong/146/2013、A/Hong Kong/4801/2014、A/Pennsylvania/28/2014、A/Samara/73/2013、A/Switzerland/9715293/2013和A/Netherlands/525/2014参考株分别代表进化簇3A、3B、3C.1、3C.2、3C.2a、3C.2b、3C.3、3C.3a和3C.3b,HA和NA氨基酸进化树采用Mega 5.2软件绘制,建树方法为邻接法(neighbor-joining,NJ),Bootstrap=1 000,进化树编辑采用Microsoft Office Word 2007软件。
1.4 HA蛋白抗原表位氨基酸突变位点、进化簇和细胞分离率分析采用Mega 5.2软件分析湖北省H3N2亚型流感病毒HA蛋白抗原表位氨基酸突变位点[11],通过对比参考株A/Perth/16/2009 HA蛋白抗原表位氨基酸序列获得。同时分析各毒株采集时间、进化簇和HA蛋白抗原表位氨基酸突变位点间的关系。
1.5 HA蛋白抗原表位三维建模分析通过SWISS-MODEL搜索3C.3a簇毒株A/Hubei-Caidian/34/2014和3C.2a簇毒株A/Hubei-Yunxi/31/2015的HA氨基酸序列的共同三维结构模板并模拟其三维结构[16]。5个主要的HA蛋白抗原表位和2种基因簇存在抗原表位氨基酸位点差异的结构均通过PyMOL软件编辑并标出[17]。
2 结果 2.1 湖北省H3N2亚型流感病毒时间分布情况2010年第1周至2015年第52周湖北省H3N2亚型流感病毒8个流行季的核酸检测阳性数和毒株分离数分别为5 903件和2 310株,而8个流行高峰的核酸检测阳性数和毒株分离数分别占流行季的80.48%(4 751/5 903) 和79.57%(1 838/2 310)。每年流行高峰核酸检测阳性数、毒株分离数和流行季峰值核酸阳性率各有不同,见表 1。2012年以后流行高峰均呈冬春季和夏季双峰季节性流行,但2013年双峰均较弱,其冬季峰核酸检测阳性数和毒株分离数仅68件和40株,峰值核酸阳性率仅11.20%;2010年呈夏秋季流行,2011年零星分布,见图 1。H3N2亚型流感病毒流行高峰的差异主要是因为受到其他型别流感流行峰的影响,如2010年冬春季流行峰主要流行B型流感病毒,2011年仅有的冬春季流行峰主要流行甲型H1N1型流感病毒,2013年的双峰同期主要流行甲型H1N1型流感病毒。
| 流行高峰 序号 |
流行期时间段 (年.周) |
核酸阳性数 (件) |
毒株分离数 (株) |
峰值核酸阳 性率(%) |
| Ⅰ | 2010.30-2010.40 | 766 | 243 | 34.28 |
| Ⅱ | 2012.06-2012.17 | 563 | 256 | 22.74 |
| Ⅲ | 2012.27-2012.37 | 619 | 336 | 42.36 |
| Ⅳ | 2013.02-2013.04 | 68 | 40 | 11.20 |
| Ⅴ | 2013.49-2014.06 | 595 | 251 | 20.70 |
| Ⅵ | 2014.28-2014.37 | 836 | 322 | 36.34 |
| Ⅶ | 2014.52-2015.07 | 492 | 209 | 26.40 |
| Ⅷ | 2015.23-2015.34 | 812 | 181 | 25.95 |

|
| 图 1 2010-2015年湖北省H3N2亚型流感病毒核酸检测阳性率周分布 Figure 1 Weekly distribution of positive nucleic acid rate of influenza A (H3N2) virus in Hubei, 2010-2015 |
| |
HA氨基酸进化树表明4株2010年毒株分别隶属1、5和3A进化簇;2株2012年毒株均隶属3C.1进化簇;1株2013年和2株2014年毒株隶属3C.3b进化簇;5株2014年毒株隶属3C.3a进化簇;4株2015年毒株隶属3C.2a进化簇;另外2株2014年毒株则属于早期的3C.3进化簇。NA氨基酸进化树表明4株2010年毒株分别隶属3和4进化簇;2株2012年毒株分别隶属3C.2和3C.3进化簇;1株2013年、4株2014年和4株2015年毒株隶属3C.3b进化簇;1株2014年毒株隶属3C.2a进化簇;另外4株2014年毒株则属于3C.2b进化簇。20株湖北省H3N2亚型流感病毒在HA和NA氨基酸进化树上的进化簇分布存在差异见图 2,这种差异在欧洲CDC报告中的H3N2亚型流感病毒HA和NA氨基酸进化树分布也有报道[15]。
20株湖北省H3N2亚型流感病毒分离株和A/Perth/16/2009参考株进行HA蛋白抗原决定簇比对分析发现A、B、C、D和E抗原决定簇上均有氨基酸突变[11],其突变位点数占其总氨基酸数的比例分别为A(5/19)、B(6/22)、C(8/27)、D(3/41) 和E(6/22)。同一流行高峰内75%(15/20) 分离株的进化簇相同,虽然A/Hubei-Wuchang/1271/2010属于第Ⅰ流行高峰,但却属于进化簇5。A/Hubei-Enshi/1250/2010(3A簇)、A/Hubei-Hongshan/1437/2013(3C.3b簇)、A/Hubei-Caidian/34/2014(3C.3a簇)和A/Hubei-Dongbao/1205/2015(3C.2a簇)则都在非流行高峰期发现。相隔A/Hubei-Caidian/34/2014(3C.3a簇)9周后发现的A/Hubei-Yunxi/31/2015隶属3C.2a进化簇且属于第Ⅶ流行高峰,两毒株间7个不同的氨基酸变异分布于3个抗原决定簇上,见表 2。第Ⅶ和第Ⅷ流行高峰分离到的毒株均为3C.2a簇,且3C.2a簇病毒均携带了其特征氨基酸突变位点158N-159Y-160T[6-7]。
| 毒株名 | 采集日期 (年/周) |
进化簇 | 流行 高峰a |
A | B | C | D | E | ||||||||||||||||||||||||||
| 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | 0 | 0 | 2 | 2 | 3 | 3 | 2 | 2 | 2 | 0 | 0 | 0 | 0 | 2 | 2 | ||||||||
| 2 | 3 | 4 | 4 | 4 | 2 | 5 | 5 | 6 | 6 | 9 | 4 | 4 | 5 | 7 | 8 | 0 | 1 | 1 | 1 | 3 | 6 | 8 | 9 | 9 | 6 | 6 | ||||||||
| 4b | 8 | 2 | 4 | 5 | 8 | 8 | 9 | 0 | 5 | 8 | 5 | 8 | 3 | 8 | 0 | 4 | 1 | 2 | 4 | 0 | 2 | 1 | 2 | 4 | 0 | 1 | ||||||||
| A/Perth/16/2009c | 1 | - | Sd | A | R | K | N | T | N | F | K | N | A | S | T | D | N | E | A | Q | T | S | I | K | N | K | Y | I | R | |||||
| A/Hubei-Qiaokou/1271/2010 | 10/36 | 1 | Ⅰ | . | . | . | . | . | . | . | . | . | Ke | . | . | . | . | . | . | . | . | . | I | . | . | D | . | . | M | Q | ||||
| A/Hubei-Jiangan/1737/2010 | 10/36 | 1 | Ⅰ | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | V | . | . | I | . | . | D | . | . | M | Q | ||||
| A/Hubei-Wuchang/1271/2010 | 10/40 | 5 | Ⅰ | . | . | . | N | . | . | . | . | . | . | . | . | . | N | . | A | . | . | A | I | V | E | . | . | H | . | . | ||||
| A/Hubei-Enshi/1250/2010 | 10/28 | 3A | - | . | . | . | N | . | T/I | . | . | . | . | . | . | A | . | . | . | . | . | A | I | . | E | . | R | . | . | . | ||||
| A/Hubei-Jiangan/163/2012 | 12/07 | 3C.1 | Ⅱ | . | . | . | N | . | . | . | . | . | . | S | N | I | . | K | . | . | . | A | I | . | E | . | . | . | . | . | ||||
| A/Hubei-Jiangan/192/2012 | 12/10 | 3C.1 | Ⅱ | . | . | . | N | . | . | . | . | . | . | S | N | I | N | K | . | . | . | A | I | . | E | . | . | . | . | . | ||||
| A/Hubei-Wuchang/191/2014 | 14/04 | 3C.3 | Ⅴ | . | . | . | N | S | . | . | . | . | . | S | N | I | . | K | . | . | . | A | I | . | E | . | . | H | . | . | ||||
| A/Hubei-Wuchang/192/2014 | 14/04 | 3C.3 | Ⅴ | . | . | . | N | S | . | . | . | . | . | S | N | I | . | K | . | . | . | A | I | . | E | . | . | H | . | . | ||||
| A/Hubei-Hongshan/1437/2013 | 13/40 | 3C.3b | - | . | . | G | N | S | A | . | . | . | . | S | N | I | . | K | . | . | . | A | I | . | E | . | . | . | . | . | ||||
| A/Hubei-Wuchang/170/2014 | 14/03 | 3C.3b | Ⅴ | . | . | G | N | S | A | . | . | . | . | S | N | I | . | K | . | . | . | A | I | . | E | . | . | . | . | . | ||||
| A/Hubei-Echeng/31/2014 | 14/01 | 3C.3b | Ⅴ | . | . | G | N | S | A | . | . | . | . | S | N | I | . | K | . | . | . | A | I | . | E | . | . | . | . | . | ||||
| A/Hubei-Wujiagang/1702/2014 | 14/32 | 3C.3a | Ⅵ | . | S | G | N | S | A | . | S | . | . | S | N | I | . | K | . | . | . | A | I | . | E | . | . | . | . | . | ||||
| A/Hubei-Hongshan/1670/2014 | 14/37 | 3C.3a | Ⅵ | . | S | G | N | S | A | . | Y | . | . | S | N | I | N | K | . | . | . | A | I | . | E | . | . | . | . | . | ||||
| A/Hubei-Yunxi/35/2014 | 14/32 | 3C.3a | Ⅵ | . | S | G | N | S | A | . | S | . | . | S | N | I | . | K | . | . | . | A | I | . | E | . | . | . | . | . | ||||
| A/Hubei-Wujiagang/1806/2014 | 14/36 | 3C.3a | Ⅵ | . | S | G | N | S | A | . | S | . | . | S | N | I | . | K | . | . | . | A | I | . | E | . | . | . | . | . | ||||
| A/Hubei-Caidian/34/2014 | 14/46 | 3C.3a | - | . | S | G | N | S | A | . | S | . | . | S | N | I | . | K | . | . | . | S | I | . | E | . | . | . | . | . | ||||
| A/Hubei-Yunxi/31/2015 | 15/03 | 3C.2a | Ⅶ | . | . | . | S | S | . | . | Y | T | . | S | N | I | . | K | . | . | H | A | I | . | E | . | . | . | . | . | ||||
| A/Hubei-Dongbao/1205/2015 | 15/15 | 3C.2a | - | . | . | . | S | S | . | . | Y | T/I | . | S | N | I | . | K | . | . | H | A | I | . | E | . | . | . | . | . | ||||
| A/Hubei-Dongbao/1661/2015 | 15/36 | 3C.2a | Ⅷ | R | . | . | S | S | . | . | Y | T | . | S | N | I | . | K | . | . | H | A | I | . | E | . | . | . | . | . | ||||
| A/Hubei-Jiangan/1714/2015 | 15/37 | 3C.2a | Ⅷ | . | . | . | S | S | . | . | Y | T | . | S | N | I | . | K | . | . | H | A | I | . | E | . | . | . | . | . | ||||
| 注:a流行高峰下的“Ⅰ~Ⅷ”表示各流行高峰,“-”表示不属于各流行高峰; b分别代表HA蛋白抗原决定簇A、B、C、D和E上的氨基酸位点; c和d对比参考株和对应氨基酸位点上的氨基酸突变位点; e毒株与对比参考株不同的氨基酸,“.”为毒株与参考株相同的氨基酸。 | ||||||||||||||||||||||||||||||||||
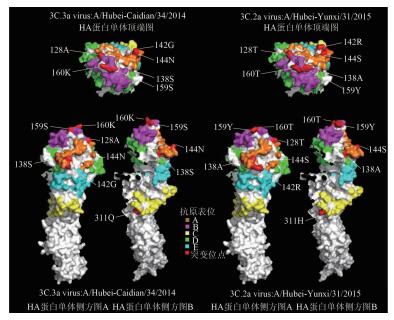
3C.2a和3C.3a病毒在HA蛋白抗原表位上存在差异的7处氨基酸突变位点主要集中抗原决定簇A(138、142和144位点)、B(128、159和160位点)和C(311位点)。为了分析这些突变位点在HA蛋白抗原表位区域上的差异,在SWISS-MODEL蛋白质结构数据库(http://swissmodel.expasy.org)获得3C.3a病毒株A/Hubei-Caidian/34/2014和3C.2a簇病毒株A/Hubei-Yunxi/31/2015的HA蛋白共同建模模板(PDB ID:4we8) 并分别建模,采用PyMOL软件编辑HA蛋白单体,每个单体均采用表面模式(Surface)输出,2种病毒的HA单体结构分别利用顶端图、侧方图A和侧方图B分析7处突变氨基酸构象的不同。
3C.2a和3C.3a病毒株间7处氨基酸突变位点在表面模式结构图中差异明显。158-159-160连续氨基酸位点处于抗原表位B的顶端,HA蛋白顶端图表明3C.2a簇病毒的159Y的突起比3C.3a病毒的159S大,160T突起比3C.3a病毒的160K小,3C.2a簇病毒的159Y-160T和3C.3a病毒的159S-160K肽平面存在差异。142~144氨基酸位点在抗原决定簇A的空间结构上是连续的,且3C.2a簇病毒的142R-144S的突起结构比3C.3a病毒的142G-144N更大,但抗原决定簇A的138位点、抗原决定簇B的128位点和抗原决定簇C的311位点在2种病毒HA蛋白构象上的差异不大,见图 3。
|
| 图 3 湖北省H3N2亚型流感病毒HA单体模拟图 Figure 3 Simulated monomer of HA protein of influenza A (H3N2) virus in Hubei |
| |
2010-2015年全球主要流行的人季节性流感病毒型别分别是甲型H1N1亚型、H3N2亚型和乙型Yamagata系,但H3N2亚型的进化和传播速度更快,造成的疾病负担更重[18-19]。2009年以来H3N2亚型病毒持续流行,抗原漂移不断发生,已进化出7个基因簇,而2014年后发生抗原漂移的H3N2亚型流感依次流行3C.3a和3C.2a簇病毒[10, 20]。它们的流行导致应用了2年的A/Texas/50/2012(2013年2月21日推荐)疫苗推荐株失效,使得WHO更新北半球H3N2亚型流感疫苗推荐株为A/Hong Kong/4801/2014(2016年2月25日推荐)[20-21]。
2010-2015年湖北省H3N2亚型流感有8个流行季,其中8个流行高峰的核酸检测阳性数和毒株分离数分别占流行季的80.48%和79.57%,H3N2亚型流感流行高峰会受到同时期其他型别流感流行的影响。同一流行高峰内75%的H3N2亚型流感病毒的进化簇相同,但也存在多个进化簇属于同一流行高峰,一些3A、3C.3b、3C.3a和3C.2a簇病毒在其对应流行高峰的前后被发现。自2015年第3周湖北省3C.2a簇病毒在第Ⅶ流行高峰被发现后,其流行态势一直延续第Ⅷ流行高峰,而同期却未发现3C.3a病毒,这和国内外报道2015年冬春季后3C.2a簇病毒数量明显多于3C.3a簇病毒的现象一致[4-5, 10, 22]。
2009年以来湖北省H3N2亚型流感病毒在5处HA抗原决定簇上不断发生氨基酸突变,其中各抗原决定簇上突变位点数占其总氨基酸数的比例分别为A(5/19)、B(6/22)、C(8/27)、D(3/41) 和E(6/22)。这些突变使流感病毒的抗原性不断发生变化,相应的疫苗株也不断更新,2010-2016年全球H3N2亚型流感推荐使用的疫苗株有:A/Perth/16/2009 (H3N2)(2010-2012年),A/Victoria/361/2011 (H3N2)(2013年),A/Texas/50/2012(2013-2014年),A/Switzerland/9715293/2013(H3N2)(2015年)和A/Hong Kong/4801/2014(2016年)。2015年和2016年的疫苗株分别为3C.3a簇病毒和3C.2a簇病毒[20-21],两种簇病毒抗原性的差异不大[10],但对湖北省9株毒株HA蛋白抗原表位上氨基酸变异的分析表明,这两种簇病毒在抗原决定簇B(128位点,159~160位点)、A(138、142和144位点)和C(311位点)均存在差异。3C.3a簇病毒株A/Hubei-Caidian/34/2014和3C.2a簇病毒株A/Hubei-Yunxi/31/2015间7处位于3个抗原决定簇上的氨基酸突变在HA蛋白表面模式结构模拟图中有明显差异,这些结构上的差异可能和3C.2a簇病毒的抗原漂移相关。
本文对湖北省H3N2亚型流感病毒的流行情况和进化树分布情况做了深入分析,结合HA蛋白抗原决定簇氨基酸突变位点和结构模拟分析,探讨了3C.2a簇病毒和3C.3a簇病毒结构差异,建议加强流感病毒进化学监测和抗原漂移分析,从而提高H3N2亚型流感病毒的病原学和流行病学监测。
作者贡献:
方斌 ORCID:0000-0003-4963-3960
方斌:负责文章的设计,测序,分析和文章撰写
余晓:负责实验毒株复核工作,参与文章的修改
李翔:负责实验毒株的分离工作,参与文章的修改
叶国军:负责流感检测数据的统计,参与文章的修改
刘琳琳:负责文章的理论指导和修改
| [1] | Rambaut A, Pybus OG, Nelson MI, et al. The genomic and epidemiological dynamics of human influenza A virus[J]. Nature, 2008, 453(7195): 615–619. DOI:10.1038/nature06945 |
| [2] | Taubenberger JK, Morens DM. Influenza:the mother of all pandemics[J]. Emerg Infect Dis, 2006, 12(1): 15–22. DOI:10.3201/eid1209.05-0979 |
| [3] | Cheng YH, Gao F, Tan MJ, et al. Post immunization sero-antibody level to circulating influenza viruses induced by seasonal influenza vaccine in China[J]. Disease Surveillance, 2015, 30(4): 260–264. (in Chinese) 成艳辉, 高枫, 谭敏菊, 等. 2013-2014年季节性流行性感冒疫苗免疫血清针对我国流行株的抗体水平分析[J]. 疾病监测, 2015, 30(4): 260–264. DOI:10.3784/j.issn.1003-9961.2015.04.005 |
| [4] | Skowronski DM, Chambers C, Sabaiduc S, et al. A perfect storm:impact of genomic variation and serial vaccination on low influenza vaccine effectiveness during the 2014-2015 season[J]. Clin Infect Dis, 2016, 63(1): 21–32. DOI:10.1093/cid/ciw176 |
| [5] | Skowronski DM, Sabaiduc S, Chambers C, et al. Mutation sacquired during cell culture isolation may affect antigenic characterisation of influenza A(H3N2) clade 3C[J]. Euro Surveill, 2016, 21(3): 30112. DOI:10.2807/1560-7917.ES.2016.21.3.30112 |
| [6] | An YM, McCullers JA, Alymova I, et al. Glycosylation analysis of engineered H3N2 influenza a virus hemagglutinins with sequentially added historically relevant glycosylation sites[J]. J Proteome Res, 2015, 14(9): 3957–3969. DOI:10.1021/acs.jproteome.5b00416 |
| [7] | Tate MD, Job ER, Deng YM, et al. Playing hide and seek:how glycosylation of the influenza virus hemagglutinin can modulate the immune response to infection[J]. Viruses, 2014, 6(3): 1294–1316. DOI:10.3390/v6031294 |
| [8] | Liang LJ, Ni HZ, Zhang X, et al. Evolution and variation of the genes encoding hemagglutinin of H3N2 influenza virus strains prevalent in Guangdong during 2011-2015[J]. Chinese Journal of Microbiology and Immunology, 2015, 35(12): 900–904. (in Chinese) 梁丽君, 倪汉忠, 张欣, 等. 2011-2015年广东省H3N2亚型流感病毒血凝素基因变异和进化分析[J]. 中华微生物学和免疫学杂志, 2015, 35(12): 900–904. DOI:10.3760/cma.j.issn.0254-5101.2015.12.008 |
| [9] | Xia YL, Zhang WJ, Tian D, et al. Antigenic analysis of the human influenza A(H3N2) virus during 2012-2015 in Shanghai, China[J]. Chinese Journal of Experimental and Clinical Virology, 2016, 30(2): 133–137. (in Chinese) 夏益兰, 张万菊, 田棣, 等. 2012-2015年上海地区成人甲型H3N2流感病毒抗原漂变分析[J]. 中华实验和临床病毒学杂志, 2016, 30(2): 133–137. |
| [10] | Huang WJ, Cheng YH, Li XY, et al. Virological characteristics of influenza A(H3N2) virus strains isolated in mainland China during 2014-2015 influenza surveillance year[J]. Chinese Journal of Microbiology and Immunology, 2016, 36(1): 3–8. (in Chinese) 黄维娟, 成艳辉, 李希妍, 等. 2014-2015监测年度中国H3N2亚型流感病毒病原学特征分析[J]. 中华微生物学和免疫学杂志, 2016, 36(1): 3–8. |
| [11] | Skowronski DM, Janjua NZ, de Serres G, et al. Low 2012-13 influenza vaccine effectiveness associated with mutation in the egg-adapted H3N2 vaccine strain not antigenic drift in circulating viruses[J]. PLoS One, 2014, 9(3): e92153. DOI:10.1371/journal.pone.0092153 |
| [12] | Tong ZD, Pu LY, Yu QY, et al. Early warning of influenza epidemic based on influenza sentinel surveillance[J]. Disease Surveillance, 2011, 26(5): 386–387. (in Chinese) 仝振东, 蒲柳艳, 虞奇跃, 等. 一种基于流感哨点监测的流感预警分析方法[J]. 疾病监测, 2011, 26(5): 386–387. |
| [13] | Cowling BJ, Wong IO, Ho LM, et al. Methods for monitoring influenza surveillance data[J]. Int J Epidemiol, 2006, 35(5): 1314–1321. DOI:10.1093/ije/dyl162 |
| [14] | McCauley J, Daniels R, Liu YP, et al. Report prepared for the WHO annual consultation on the composition of influenza vaccine for the Southern Hemisphere 2015[R]. London:WHO Influenza Centre London, 2014:38-87. https://www.crick.ac.uk/media/221823/nimr-vcm-report-sep-14-web.pdf. |
| [15] | Daniels R, Gregory V, Ermetal B, et al. Influenza virus characterisation, summary Europe, February 2016[R]. Stockholm:ECDC, 2016:12-15.http://ecdc.europa.eu/en/publications/surveillance_reports/Pages/index.aspx. |
| [16] | Arnold K, Bordoli L, Kopp J, et al. The SWISS-MODEL workspace:a web-based environment for protein structure homology modelling[J]. Bioinformatics, 2006, 22(2): 195–201. DOI:10.1093/bioinformatics/bti770 |
| [17] | Delano WL. The PyMOL molecular graphics system[M]. San Carlos, CA: DeLano Scientific LLC, 2002. |
| [18] | Bedford T, Suchard MA, Lemey P, et al. Integrating influenza antigenic dynamics with molecular evolution[J]. eLife, 2014, 3(4): e01914. |
| [19] | Li J, Zhou YY, Kou Y, et al. Interim estimates of divergence date and vaccine strain match of human influenza A(H3N2) virus from systematic influenza surveillance (2010-2015) in Hangzhou, southeast of China[J]. Int J Infect Dis, 2015, 40: 17–24. DOI:10.1016/j.ijid.2015.09.013 |
| [20] | Daniels R, Ermetal B, Rattigan A, et al. Influenza virus characterisation, summary Europe, September 2015[R]. Stockholm:ECDC, 2015:8-9. http://ecdc.europa.eu/en/publications/surveillance_reports/Pages/index.aspx. |
| [21] | WHO. Recommended composition of influenza virus vaccines for use in the 2016-2017 northern hemisphere influenza season[EB/OL]. (2016-02-25)[2016-11-01]. http://www.who.int/influenza/vaccines/virus/recommendations/201602_recommendation.pdf. |
| [22] | Flannery B, Zimmerman RK, Gubareva LV, et al. Enhanced genetic characterization of influenza A(H3N2) viruses and vaccine effectiveness by genetic group, 2014-2015[J]. J Infect Dis, 2016, 214(7): 1010–1019. DOI:10.1093/infdis/jiw181 |
 2017, Vol. 32
2017, Vol. 32