扩展功能
文章信息
- 范如岳, 白向宁, 傅珊珊, 许艳梅, 熊衍文
- FAN Ru-yue, BAI Xiang-ning, FU Shan-shan, XU Yan-mei, XIONG Yan-wen
- 非O157产志贺毒素大肠埃希菌分离株血清型鉴定及主要毒力基因分析
- Serotype and virulent gene profiles of non-O157 Shiga toxin-producing Escherichia coli isolates
- 疾病监测, 2017, 32(9): 716-720
- Disease Surveillance, 2017, 32(9): 716-720
- 10.3784/j.issn.1003-9961.2017.09.004
-
文章历史
- 收稿日期:2017-03-03
产志贺毒素大肠埃希菌(Shiga toxin-producing Escherichia coli,STEC)能产生一种或多种志贺毒素,引起人类腹泻、出血性结肠炎(hemorrhagic colitis,HC)和溶血性尿毒综合征(hemolytic uremic syndrome,HUS)而引起广泛关注[1-2]。O157 : H7血清型是与STEC感染暴发相关的主要类型,但近年来越来越多的非O157 STEC与人类疾病相关,其引起的病例在某些国家甚至超过了STEC O157[3-4],特别是2011年5月德国暴发的O104 : H4疫情,波及范围广、感染人数众多,引起了人们对非O157 STEC的重视[5]。反刍动物,特别是牛,被认为是STEC主要的宿主,在我国,猪、青藏高原的牦牛、鼠兔以及生肉类食品为非O157 STEC的重要来源[6-8]。人通过摄入受污染的食物、水,或直接与动物接触,可导致散发感染或疾病暴发。
志贺毒素(Shiga toxin,Stx)是STEC最关键的毒力因子,包括无免疫交叉反应的Stx1和Stx2二种型别[3],不同Stx类型和亚型可能与疾病的严重程度相关[6,9-10]。eae是位于LEE致病岛上的重要基因,其编码的紧密黏附素可导致人肠上皮细胞产生黏附抹平损伤,在致病过程中发挥着重要作用,eae和stx基因的组合可能与STEC菌株的毒力增强相关[11]。除stx和eae基因外,其他毒力因子也可能存在于STEC菌株中并增加其致病性,如STEC O157菌株质粒携带的肠溶血素基因(ehxA)[12]。
血清分型是大肠埃希菌流行病学调查和系统进化研究的一种重要分型方法,对于指导疫情调查和疾病监测具有重要的意义。菌体脂多糖抗原(O抗原)和鞭毛抗原(H抗原)是大肠埃希菌最常用的血清分型体系。目前,大肠埃希菌中发现由不同的O抗原基因簇编码的O1~O188共185种O血清群[13]和53种(H1~H56) H型。H型中44种主要由鞭毛蛋白fliC基因编码,H3、H35、H36、H47和H53由flkA编码,H44和H55由fllA编码,H54由flmA编码而H17由flnA编码[14]。我国自2000年开展STEC O157主动监测,对于非O157 STEC的流行情况了解甚少。本研究对我国400余株不同来源的非O157 STEC菌株进行血清分型和主要毒力基因检测,结果报道如下。
1 材料与方法 1.1 菌株来源2009-2016年共收集非O157 STEC菌株434株,分离自河南、青海、北京、四川、上海、广东等省(直辖市),见表 1。菌株来源于牦牛[8]、羊[15]、猪[7]、生肉类[6]、鼠兔[16]及人[17]等,以及地方疾病预防控制中心(CDC)送检复核的菌株。所有菌株均通过系统生化反应鉴定为大肠埃希菌、PCR检测含有stx基因并确定与O157诊断血清不凝集后由实验室保存。
| 来源 | 地点(省、直辖市) | 时间 | 样本数量 | 分离株数量 |
| 牦牛 | 青海 | 2012 | 728 | 128 |
| 鼠兔 | 青海 | 2012, 2013, 2015 | 1 116 | 22 |
| 羚羊 | 青海 | 2014 | 505 | 5 |
| 旱獭 | 青海 | 2012, 2013 | 335 | 8 |
| 牛 | 黑龙江、山东 | 2009, 2012, 2015 | 440 | 14 |
| 羊 | 河南 | 2011 | 不详a | 28 |
| 猪 | 重庆、北京、贵州、山东、黑龙江 | 2011-2012,2015 | 1 003 | 135 |
| 食品 | 北京、四川 | 2013-2014 | 853 | 60 |
| 腹泻患者 | 河南、广东、上海、四川、山东 | 2010-2014 | 不详a | 30 |
| 健康从业人员 | 青海、广东 | 2013-2014, 2016 | 2 857 | 4 |
| 合计 | 7 837 | 434 | ||
| 注:a来自不同的样品,部分为地方疾病预防控制中心送检复核的菌株 | ||||
LB培养基购自北京陆桥技术有限责任公司;细菌基因组DNA提取试剂盒购自北京康为世纪生物科技有限公司;Ex Taq DNA聚合酶、10×PCR Buffer、dNTPs及DL 2000 DNA Marker购自大连TaKaRa公司;PCR引物由天一辉远(北京)生物科技有限公司合成;PCR仪(SensoQuest Labcycler)购自德国Senso公司;引物由生工生物工程(上海)有限公司合成;PCR产物用QIAquick Gel Extraction Kit(QIAGEN)试剂盒回收纯化并由天一辉远(北京)生物科技有限公司完成测序;凝胶成像系统(GEL Doc XR+)购自美国Bio-Rad公司;大肠埃希菌全套O抗诊断血清购自丹麦血清学研究所(statens serum institut,SSI)和天津生物芯片技术有限责任公司。
1.3 血清型的确定 1.3.1 DNA模板的制备采用细菌基因组提取试剂盒提取细菌基因组DNA,置-20 ℃保存备用。
1.3.2 菌悬液的制备挑取LB培养基上的5~10个菌落加入200 μl的生理盐水中(约1个麦氏单位),100 ℃水浴1 h后,放室温静置1~2 h后使用。
1.3.3 大肠埃希菌O血清群的确定首先采用大肠埃希菌O抗原PCR分型方法对菌株进行O血清群的初筛[9],然后按照血清使用说明书以相应的O抗诊断血清对菌悬液进行凝集。对于血清复核与PCR结果不一致以及PCR阴性的菌株,再以O抗全套诊断血清进行测定。全套O抗血清均不能凝集和发生自凝的菌株按不可分型处理。
1.3.4 大肠埃希菌H型的确定采用已建立的PCR方法,利用引物fliC-F和 fliC-R (表 2)[18]对所有菌株进行PCR扩增,扩增产物进行测序,测得的序列经过整理后在SerotypeFinder公共数据库(https://cge.cbs.dtu.dk/services/SerotypeFinder/)中搜索比对,确定相应的菌株H型。
| 目的基因 | 引物序列(5′~3′) | 退火温度(℃) | 扩增产物长度(bp) | 参考文献 |
| fliC | F:ATG GCA CAA GTC ATT AAT ACC CAA C R:CTA ACC CTG CAG CAG AGA CA | 55 | 1 300~2 600 | [18] |
| stx1 | F:AAA TCG CCA TTC GTT GAC TAC TTC T R:TGC CAT TCT GGC AAC TCG CGA TGC A | 58 | 370 | [8] |
| stx2 | F: CAG TCG TCA CTC ACT GGT TTC ATC AC R:GGA TAT TCT CCC CAC TCT GAC ACC | 58 | 283 | [8] |
| eae | F:TCA ATG CAG TTC CGT TAT CAG TT R:GTA AAG TCC GTT ACC CCA ACC TG | 58 | 482 | [8] |
| ehxA | F: GGT GCA GCA GAA AAA GTT GTA G R: TCT CGC CTG ATA GTG TTT GGT A | 57 | 1 551 | [8] |
采用PCR对434株非O157 STEC进行stx1、stx2、eae及ehxA基因检测[8]。PCR反应体系为20 μl,内含2×Taq MasterMix 10 μl,10 μmol/L的上下游引物各1 μl,加水至19 μl,模板DNA 1.0 μl。PCR引物序列见表 2。
2 结果 2.1 非O157 STEC分离株的血清型434株菌株中可确定O血清群的为367株,占分离菌株的84.56%,见图 1。本研究中有5株菌株同时与单抗O2和O50发生凝集,1株同时与O20和O60单抗发生凝集,1株同时与O110与O113单抗发生凝集,1株同时与O12和O78单抗发生凝集,共鉴定出82种不同O血清群,其中主要血清群为O8、O2、O20、O78、O117,分别占分离株的10.14%、9.22%、7.83%、4.38%和4.15%。
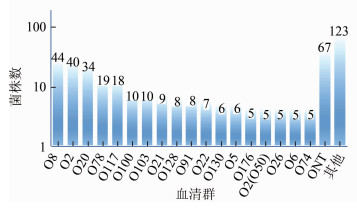
|
| 图 1 434株非O157 STEC血清群分布 Figure 1 Serogroups of 434 non-O157 STEC isolates |
| |
除16株无法确定H型外,434株菌中共检测到28种H型。H30(83/434,19.12%)、H8(62/434,14.29%)、H21(46/434,10.60%)、H45(28/434,6.45%)和H32(25/434,5.76%)为优势型,占可分型菌株的57.96%(244/421)。
在434株菌O : H血清型分型中,O20 : H30血清型的菌株有28株,O2 : H32有22株,O2 : H45有17株,O8 : H16有15株,O117 : H21和O8 : H9血清型的菌株各11株,共占分离株的23.96%(104/434),为非O157 STEC的主要血清型,其余血清型有1~9株菌。
2.2 不同宿主来源非O157 STEC分离株的血清型分布从腹泻患者样本分离出的30株菌株中,除1株O抗原无法分型外,共检出15种血清群,其中O130、O26、O107、O3、O68、O48、O111血清群仅在腹泻患者分离株中发现。腹泻患者优势血清型是O130 : H8和O26 : H11,分别占20.00%(6/30) 和16.67%(5/30)。
健康从业人员粪便分离的4株菌的血清型分别为O43 : H2、O91 : H14、O8 : H19和ONT : H21。其中血清型O8 : H19和O91 : H14同时也在动物粪便和食品样品中检出。
分离自动物粪便的340株菌株,除64株O血清群无法分型外,属于65个O血清群,O2血清群数量最多,含40株菌,其余依次为O8、O20、O78、O117,分别占动物宿主STEC的10.29%(35/340)、9.41%(32/340)、5.59%(19/340) 和4.41%(15/340)。在可分型菌株中,O78、O6、O74、O165、O172、O53等44种血清群仅在动物宿主中检测到。
60株分离自食品的STEC菌株中,除1株O血清群无法分型外,共检测出25种血清群,其中O128、O8血清群菌株数量最多,均包含8株菌株。O176、O98、O40、O57和O161血清群仅在食品来源STEC中检测出,而其他20种血清群在其他不同宿主中均有发现。
2.3 主要毒力基因检测结果434株不同来源宿主的非O157 STEC菌株共有stx1、stx2及stx1+stx2 3种志贺毒素基因型,阳性率分别为25.35%(110/434)、64.98%(282/434) 和9.68%(42/434),见表 3。17株菌检出eae基因,占分离株的3.92%,而ehxA基因的检出率为32.95%(143/434)。15株分离株同时携带stx、eae和ehxA基因,见表 4。
| 主要毒力基因 | 腹泻患者粪便 | 健康人粪便 | 动物粪便 | 动物性食品 | 合计 |
| stx1 | 27 | 1 | 62 | 20 | 110 |
| stx2 | 3 | 2 | 248 | 29 | 282 |
| eae | 8 | 0 | 5 | 4 | 17 |
| ehxA | 9 | 1 | 110 | 23 | 143 |
| 菌株编号 | 标本来源 | 血清型 | stx1 | stx2 | eae | ehxA |
| ST056 | 牦牛粪便 | O78:NT | - | + | + | + |
| ST068 | 牦牛粪便 | O78:H21 | - | + | + | + |
| ST310 | 腹泻患者 | O5:HNT | + | - | + | + |
| ST312 | 腹泻患者 | O26:H11 | + | - | + | + |
| ST348 | 牛肉 | O103:H25 | + | - | + | + |
| ST350 | 牛肉 | O103:H25 | + | - | + | + |
| ST365 | 牛肉 | O12:HNT | - | + | + | + |
| ST377 | 羊肉 | O5:H9 | + | - | + | + |
| ST406 | 腹泻患者 | O26:H11 | + | - | + | + |
| ST407 | 腹泻患者 | O5:HNT | + | - | + | + |
| ST411 | 腹泻患者 | O26:H11 | + | - | + | + |
| ST415 | 腹泻患者 | O84:H2 | + | - | + | + |
| ST416 | 腹泻患者 | O26:H11 | + | - | + | + |
| ST461 | 牛粪便 | O177:HNT | - | + | + | + |
| ST508 | 腹泻患者 | O26:H11 | + | - | + | + |
携带stx1基因的110株菌中,除了18株菌O抗原血清群无法分型外,优势血清群为O2、O8、O103、O21和O128,其中O2 : H45血清型数量最多(15.45%,17/110)。检测出stx2基因的282株菌中,有55株菌O抗原无法分型,O20、O2、O78、O117和O100为优势血清群,占stx2基因分离株的37.59%(106/282),优势血清型为O20 : H30、O2 : H32和O8 : H16。同时具有stx1和stx2的42株菌中,以O8 : H9、O128 : H2、O91 : H14的数量较多,共19株。eae基因主要存在于O26 : H11血清型的菌株中(29.41%,5/17)。携带ehxA基因的菌株主要属于O8(17/143)、O78(13/143) 和O103(10/143) 血清群。
分离自腹泻患者样本的非O157 STEC中,stx1基因的携带率为90.00%(27/30),仅有3株菌检测出stx2基因,而eae和ehxA的检出率分别为26.67%(8/30) 和30.00%(9/30)。分离自健康人的4株菌中,2株携带stx2基因,1株携带stx1基因,1株stx1和 stx2同时检测出。动物宿主的分离株中,stx1、stx2、eae和ehxA基因的阳性率分别为18.24%(62/340)、72.94%(248/340)、1.47%(5/340) 和32.35%(110/340)。分离自食品的菌株中,stx1、stx2阳性率分别为33.33%(20/60)、48.33%(29/60),有11株同时携带stx1和stx2基因(18.33%,11/60),仅有4株菌检测到eae基因,ehxA基因阳性率为38.33%(23/60)。同时携带4种毒力基因的菌株主要来源于腹泻患者分离株(53.33%,8/15)。
3 讨论目前从人类分离的STEC血清型有400余种,其中O157 : H7依然是致病力强、流行范围广、最为常见的血清型。但近年来,数据表明30.00%~60.00%的人类STEC感染由非O157 STEC引起[3,19]。O26、O45、O103、O111、O121和O145血清群占美国1983-2002年非O157 STEC病例分离株的75.00%,这6种血清群也被称为“Top Six”[20]。本研究中分离菌株的优势血清群为O8、O2、O20、O78、O117,与国外报道存在差异,可能与本研究中多数菌株来源于动物及动物源性食品有关。有报道O20 : H30为STEC常见血清型,与本研究一致[7]。本研究从腹泻患者分离株中发现5株O26血清群的菌株,与一些国家主要流行的血清群相似[18,21]。而动物样本分离株的优势血清群为O2、O8、O20,提示某些血清群的菌株主要流行于动物宿主中。由于我国非O157 STEC血清型复杂多样,根据目前的分析结果,不同来源的菌株尚不能发现地域与时间的聚集性或演变趋势。
志贺毒素作为一种细胞毒素,可以通过噬菌体的水平传递以及遗传获得[7]。本研究中stx1、stx2和stx1+stx2 3种志贺毒素均有检出,stx2阳性率最高。许多研究强调了stx2在疾病严重程度(HUS发生率)中的关键作用[22-23]。本研究中腹泻患者主要为轻度和中度腹泻,尚无出血性肠炎及HUS病例,可能与菌株主要携带stx1有关。紧密黏附素(intimin)和肠溶血素(enterohemolysin)是O157 STEC的重要毒力因子[24-25],本研究中分离株的eae和ehxA的检测率较低,可能与这些菌株的致病性较O157 STEC低有关,同时携带stx、eae和ehxA基因的菌株也主要来自腹泻患者,也间接说明了这些基因在协同致病中的作用。
本研究系统地分析了我国不同来源的非O157 STEC菌株血清型的异同以及我国腹泻患者分离株与欧美国家主要流行血清型的异同。同时通过主要毒力基因和血清型检测分析,评价了我国非O157 STEC菌株的致病潜力,对于了解我国非O157 STEC流行情况,开展有针对性的检测和监测提供参考。为有效应对暴发疫情,降低人群STEC感染的风险,进一步调整完善中国O157 : H7监测方案,选择部分有较强监测能力和工作积极性的地区开展包括O157 : H7在内的不同血清型STEC的监测具有重要意义。
作者贡献:
范如岳 ORCID:0000-0002-5958-6128
范如岳:进行实验室检测、数据整理与分析及文章撰写
白向宁、傅珊珊、许艳梅:实验室检测、实验指导及数据收集
熊衍文:进行实验设计、实验指导和论文修改
| [1] |
Grisaru S, Xie J, Samuel S, et al. Associations between hydration status, intravenous fluid administration, and outcomes of patients infected with Shiga toxin-producing Escherichia coli: a systematic review and meta-analysis[J]. JAMA Pediatr, 2017, 171(1): 68-76. DOI:10.1001/jamapediatrics.2016.2952 |
| [2] |
Purwar S, Roy S, Metgud S. Non-O157 : H7 Shiga toxin producing diarrhoeagenic Escherichia coli (STEC) in Southern India: a tinderbox for starting epidemic[J]. J Clin Diagn Res, 2016, 10(10): DC11-15. |
| [3] |
Smith JL, Fratamico PM, Gunther IV NW. Chapter three-Shiga toxin-producing Escherichia coli[J]. Adv Appl Microbiol, 2014, 86: 145-197. DOI:10.1016/B978-0-12-800262-9.00003-2 |
| [4] |
Luna-Gierke RE, Griffin PM, Gould LH, et al. Outbreaks of non-O157 Shiga toxin-producing Escherichia coli infection: USA[J]. Epidemiol Infect, 2014, 142(11): 2270-2280. DOI:10.1017/S0950268813003233 |
| [5] |
Frank C, Werber D, Cramer JP, et al. Epidemic profile of Shiga-toxin-producing Escherichia coli O104 : H4 outbreak in Germany[J]. N Engl J Med, 2011, 365(19): 1771-1780. DOI:10.1056/NEJMoa1106483 |
| [6] |
Bai XN, Wang H, Xin YQ, et al. Prevalence and characteristics of Shiga toxin-producing Escherichia coli isolated from retail raw meats in China[J]. Int J Food Microbiol, 2015, 200: 31-38. DOI:10.1016/j.ijfoodmicro.2015.01.018 |
| [7] |
Meng Q, Bai XN, Zhao AL, et al. Characterization of Shiga toxin-producing Escherichia coli isolated from healthy pigs in China[J]. BMC Microbiol, 2014, 14: 5. DOI:10.1186/1471-2180-14-5 |
| [8] |
Bai XN, Zhao AL, Lan RT, et al. Shiga toxin-producing Escherichia coli in yaks (Bos grunniens) from the Qinghai-Tibetan Plateau, China[J]. PLoS One, 2013, 8(6): e65537. DOI:10.1371/journal.pone.0065537 |
| [9] |
Anjum MF, Jones E, Morrison V, et al. Use of virulence determinants and seropathotypes to distinguish high-and low-risk Escherichia coli O157 and non-O157 isolates from Europe[J]. Epidemiol Infect, 2014, 142(5): 1019-1028. DOI:10.1017/S0950268813001635 |
| [10] |
Tseng M, Fratamico PM, Bagi L, et al. Diverse virulence gene content of Shiga toxin-producing Escherichia coli from finishing swine[J]. Appl Environ Microbiol, 2014, 80(20): 6395-6402. DOI:10.1128/AEM.01761-14 |
| [11] |
Etcheverría AI, Padola NL. Shiga toxin-producing Escherichia coli: factors involved in virulence and cattle colonization[J]. Virulence, 2013, 4(5): 366-372. DOI:10.4161/viru.24642 |
| [12] |
Cookson AL, Bennett J, Thomson-Carter F, et al. Molecular subtyping and genetic analysis of the enterohemolysin gene (ehxA) from Shiga toxin-producing Escherichia coli and atypical enteropathogenic E. coli[J]. Appl Environ Microbiol, 2007, 73(20): 6360-6369. DOI:10.1128/AEM.00316-07 |
| [13] |
Iguchi A, Iyoda S, Seto K, et al. Escherichia coli O-genotyping PCR: a comprehensive and practical platform for molecular O serogrouping[J]. J Clin Microbiol, 2015, 53(8): 2427-2432. DOI:10.1128/JCM.00321-15 |
| [14] |
Feng L, Liu B, Liu YQ, et al. A genomic islet mediates flagellar phase variation in Escherichia coli strains carrying the flagellin-specifying locus flk[J]. J Bacteriol, 2008, 190(13): 4470-4477. DOI:10.1128/JB.01937-07 |
| [15] |
Bai XN, Zhao AL, Xia SL, et al. Multilocus sequence typing of non-O157 Shiga toxin-producing Escherichia coli isolates[J]. Chinese Journal of Zoonoses, 2012, 28(6): 544-560. (in Chinese) 白向宁, 赵爱兰, 夏胜利, 等. 非O157产志贺毒素大肠杆菌分离株的多位点序列分型研究[J]. 中国人兽共患病学报, 2012, 28(6): 544-560. |
| [16] |
Bai XN, Zhang W, Tang XY, et al. Shiga toxin-producing Escherichia coli in plateau pika (Ochotona curzoniae) on the Qinghai-Tibetan plateau, China[J]. Front Microbiol, 2016, 7: 375. |
| [17] |
Bai XN, Hu B, Xu YM, et al. Molecular and phylogenetic characterization of non-O157 Shiga toxin-producing Escherichia coli strains in China[J]. Front Cell Infect Microbiol, 2016, 6: 143. |
| [18] |
Fields PI, Blom K, Hughes HJ, et al. Molecular characterization of the gene encoding H antigen in Escherichia coli and development of a PCR-restriction fragment length polymorphism test for identification of E. coli O157 : H7 and O157 : NM[J]. J Clin Microbiol, 1997, 35(5): 1066-1070. |
| [19] |
EFSA Panel on Biological Hazards (BIOHAZ). Scientific opinion on VTEC-seropathotype and scientific criteria regarding pathogenicity assessment[J]. EFSA J, 2013, 11(4): 3138. DOI:10.2903/j.efsa.2013.3138 |
| [20] |
European Food Safety Authority. Technical specifications for the monitoring and reporting of verotoxigenic Escherichia coli (VTEC) on animals and food (VTEC surveys on animals and food)[J]. EFSA J, 2009, 7(11): 1366. DOI:10.2903/j.efsa.2009.1366 |
| [21] |
Brooks JT, Sowers EG, Wells JG, et al. Non-O157 Shiga toxin-producing Escherichia coli infections in the United States, 1983-2002[J]. J Infect Dis, 2005, 192(8): 1422-1429. DOI:10.1086/jid.2005.192.issue-8 |
| [22] |
Ethelberg S, Olsen KEP, Scheutz F, et al. Virulence factors for hemolytic uremic syndrome, Denmark[J]. Emerg Infect Dis, 2004, 10(5): 842-847. DOI:10.3201/eid1005.030576 |
| [23] |
Byrne L, Vanstone GL, Perry NT, et al. Epidemiology and microbiology of Shiga toxin-producing Escherichia coli other than serogroup O157 in England, 2009-2013[J]. J Med Microbiol, 2014, 63(9): 1181-1188. |
| [24] |
Slanec T, Fruth A, Creuzburg K, et al. Molecular analysis of virulence profiles and Shiga toxin genes in food-borne Shiga toxin-producing Escherichia coli[J]. Appl Environ Microbiol, 2009, 75(19): 6187-6197. DOI:10.1128/AEM.00874-09 |
| [25] |
Knust Z, Schmidt G. Cytotoxic necrotizing factors (CNFs)-a growing toxin family[J]. Toxins, 2010, 2(1): 116-127. DOI:10.3390/toxins2010116 |
 2017, Vol. 32
2017, Vol. 32


