扩展功能
文章信息
- 魏民, 邵一鸣
- WEI Min, SHAO Yi-ming
- 人体细胞膜跨膜蛋白丝氨酸整合因子3和5的抗艾滋病毒作用
- Inhibition of HIV-1 by SERINC3 and SERINC5
- 疾病监测, 2017, 32(12): 911-913
- Disease Surveillance, 2017, 32(12): 911-913
- 10.3784/j.issn.1003-9961.2017.12.004
-
文章历史
- 收稿日期:2017-12-02
2. 中国疾病预防控制中心性病艾滋病预防控制中心, 北京 102206
2. National Center for AIDS/STD Prevention and Control, Chinese Center for Disease Control and Prevention, Beijing 102206, China
艾滋病作为严重的传染病,需要监测才能了解其在我国以及全世界的流行情况。疾病监测能更好地控制艾滋病的流行,并做好疾病的干预和预防。这是从大人群和疾病流行的角度分析问题。从个体角度来说,艾滋病病毒(或人类免疫缺陷病毒,HIV-1)进入人体,人体的免疫系统会发现病毒,并将之消灭。这就涉及病毒的人体免疫监测或者免疫监视。人体的免疫监视是重要的发现病毒、控制病毒复制的重要步骤,构成了人体的HIV-1免疫防御体系[1]。而另外一方面,HIV-1为了能够在人体内生存,病毒本身也编码蛋白拮抗人体的免疫防御系统[1-2]。
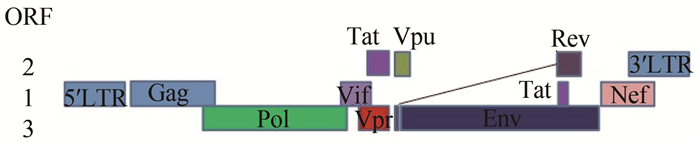
在HIV-1的基因组中,两端为5′LTR(长末端重复序列)和3′LTR,中间为编码衣壳蛋白(Gag)、聚合酶蛋白(Pol)、包膜蛋白(Env)的结构蛋白,还有很多辅助蛋白,如Tat、Rev、Vif、Vpr、Vpu和Nef[2],见图 1。自发现HIV-1以来,这些辅助蛋白的功能,就成为科学家们研究的对象。Tat蛋白与HIV-1 LTR区的TAR RNA结合,反式激活病毒的转录;Rev蛋白和HIV-1 Env区的RRE(Rev Response Element)结合,负责将HIV-1基因组带出细胞核[3]。胞嘧啶脱氨酶Apobec3G(ApolipoproteinB mRNA editing enzyme-catalytic polypeptide 3G)和黏附素Tetherin(BST2)的发现,开启了人类发现宿主限制性因子的先河,具有重要的抗病毒作用,属于人体免疫的一部分;而病毒分别编码Vif和Vpu蛋白拮抗这2个宿主限制性因子[1, 4-5]。
|
| 图 1 HIV-1 HXB2基因组示意图 Figure 1 Genome of HIV-1 HXB2 注:基因组编码的蛋白分别处3个不同的蛋白读码框(open reading frame,ORF),不同颜色分别标注HIV-1的结构蛋白Gag、Pol、Env和辅助蛋白Vif、Vpu、Vpr、Tat、Rev、Nef。Tat蛋白带有2个外显子;Rev蛋白也有2个外显子。粉色的为Nef蛋白(摘自HIV Sequence Compendium 2012,Los Alamos HIV Sequence Database and Analysis Staff) |
| |
HIV-1也编码Nef蛋白,Nef蛋白大约27×103~32×103,邻近HIV-1基因组的3′端,与3′LTR区部分重叠[1-2](图 1)。早期的研究表明,突变Nef蛋白后,HIV-1的复制能力增强;Nef蛋白有减慢HIV-1复制的能力,因此把此蛋白称为负调控因子(Negative factor,Nef)[6-7]。越来越多的证据表明,这个结果是错误的,Nef蛋白对于HIV-1的复制是正向的,带有Nef(Nef+)的HIV-1比缺失Nef(Nef-)的病毒复制能力更强,生长更快[8-9]。以前的差错可能是实验中干扰了HIV-1 3′LTR的转录,造成复制减慢[8-9],但Nef蛋白的名称却一直延用至今。Nef蛋白对于HIV-1是一个完全的正向调控因子,负调控因子的名称是误导[9]。Nef蛋白对于维持HIV-1在血浆中的高病毒载量起到重要作用,与艾滋病的疾病进展有关;如果出现突变,艾滋病进展缓慢。曾在长期病情不进展的艾滋病患者中发现有Nef突变的现象[10]。Nef蛋白的N端具有豆蔻酰基化修饰,可以和细胞膜结合。现在已知Nef蛋白是一个多功能蛋白,Nef蛋白加速疾病进展;促进病毒在CD4+细胞间的传播;下调CD4分子、MHC类等分子;Nef蛋白增强病毒的复制能力和感染性[9]。
1 人体细胞膜跨膜蛋白丝氨酸整合因子3和5的发现自从1994年发现Nef蛋白可以增强病毒的感染性,20多年来,其机制却并不清楚。这里的感染性是指病毒在细胞中建立感染的能力,通常用一轮病毒复制的能力表示,使用报告基因检测,如带有荧光素酶的Hela-TZM-bl细胞等。在以前的研究中,人们就发现不同的细胞对于Nef的依赖程度不同,有些细胞依赖Nef蛋白的程度很低。2015年,意大利Pizzato课题组做了深入细致的研究[11],在不同的细胞系中,把Nef+和Nef-的HIV-1感染性的比值(ratio)做比较。发现在T淋巴细胞系Jurkat TAg细胞中(带有SV40大T抗原的Jurkat E6.1细胞),Nef+/Nef-的HIV-1感染性比值最高为40;而在HT1080细胞中,Nef+/Nef-的感染性比值最低为2[11]。而其他细胞(如Hela、H9、MT2、HEK293T等)位于中间[11]。也就是说,Jurkat TAg细胞最依赖Nef蛋白,而HT1080细胞对于Nef的依赖程度很低,其他的细胞位于中间。Pizzato课题组的工作人员继续把Jurkat TAg细胞和HT1080做细胞融合,发现融合后的细胞对于Nef依然依赖,接近于Jurkat TAg细胞,提示可能有1个抑制因子在起作用。细胞融合实验是用来验证有抑制因子或者辅助因子的实验。如果上述的融合实验结果接近于HT1080细胞,也就是融合后的细胞表型接近于HT1080,对于Nef不依赖,那么可能为1个Nef的辅助因子。他们对7个最依赖Nef的细胞和8个对Nef依赖低的细胞做了全部的转录子(transcriptome)测序分析,并进行相关性分析,发现SERINC5(serine incorporator 5,丝氨酸整合因子5)与Nef的相关性最高,相关系数达到0.945[11]。
意大利Pizzato课题组为进一步验证SERINC5的作用,在Jurkat TAg细胞中,使用CRISPR-Cas9技术敲除了SERINC5,发现Nef-的HIV-1感染性增加了20~30倍,而Nef+的病毒感染性只增加了2~3倍[11]。另外,在敲除SERINC5的Jurkat TAg中,当回补SERINC5,则Nef依赖的表型又重新恢复。他们又进一步在Nef依赖性低的HEK293T细胞中表达SERINC5,使得Nef-的HIV-1感染性降低了10~40倍[11]。这些结果表明SERINC5是抗Nef蛋白的抑制因子。这一结果也得到了美国麻省医学院Göttlinger课题组的支持[12]。他们比较了Nef+和Nef-的HIV-1中宿主蛋白,使用蛋白质组分析的方法,也发现了丝氨酸整合因子3(serine incorporator 3,SERINC3)和SERINC5的抗病毒作用。
目前,已知人类SERINC家族包括5个成员,SERINC1~5为跨膜蛋白,功能可能是负责合成细胞膜含有丝氨酸的脂类,如磷脂酰丝氨酸(phosphatidylserine)和鞘磷脂(sphingolipids)[13]。该家族中,只有SERINC3和SERINC5具有抗HIV-1作用,且SERINC5的作用强于SERINC3[11-12]。
2 SERINC3和SERINC5的作用机制目前,SERINC3和SERINC5的抗HIV-1作用还不完全清楚。简单来讲,在无Nef的情况下,细胞膜跨膜蛋白SERINC3和SERINC5可以进入到HIV-1粒子中,而当该病毒感染新的细胞时,病毒感染性降低,机制尚不明确[11-12]。Nef蛋白可以拮抗这一作用,Nef蛋白可以和SERINC5结合,使SERINC5到达Rab7阳性的溶酶体中,可能进一步降解[11-12]。这一机制与Vpu拮抗黏附素Tetherin(BST2)有相似之处。
HIV-1 Nef蛋白拮抗SERINC5的作用也可以被另外一个无关蛋白——鼠白血病病毒(murine leukemia virus,MuLV)的糖基化的Gag蛋白(glycoGag)代替[11-12]。MuLV的glycoGag蛋白,除了在N末端多了88个氨基酸外,其他的氨基酸与MuLV结构蛋白Gag相同,在一个读码框。MuLV的glycoGag蛋白使用CUG作为起始密码,在Gag的AUG起始密码上游[9, 11]。也就是MuLV也编码glycoGag蛋白拮抗SERINC5。另外反转录病毒——马传染性贫血病毒(equine infectious anemia virus)也编码S2蛋白拮抗SERINC3和SERINC5[14]。
3 小结SERINC3和SERINC5的发现在已有的宿主限制性因子上,又增加了2个。使得人们对于病毒与宿主的关系有了更深入的了解。这一发现也成为抗艾滋病药物研发的新靶点。更深入的研究,可能会揭示更广阔的发现。
作者贡献:
魏民 ORCID:0000-0003-0949-007X
魏民:收集整理文献、阅读文献,起草文章、最终定稿
邵一鸣:修改文章、最终定稿
| [1] |
Malim MH, Emerman M. HIV-1 accessory proteins-ensuring viral survival in a hostile environment e[J]. Cell Host Microbe, 2008, 3(6): 388-398. DOI:10.1016/j.chom.2008.04.008 |
| [2] |
Frankel AD, Young JAT. HIV-1:fifteen proteins and an RNA[J]. Annu Rev Biochem, 1998, 67: 1-25. DOI:10.1146/annurev.biochem.67.1.1 |
| [3] |
Emerman M, Malim MH. HIV-1 regulatory/accessory genes:keys to unraveling viral and host cell biology[J]. Science, 1998, 280(5371): 1880-1884. DOI:10.1126/science.280.5371.1880 |
| [4] |
Sheehy AM, Gaddis NC, Choi JD, et al. Isolation of a human gene that inhibits HIV-1 infection and is suppressed by the viral Vif protein[J]. Nature, 2002, 418(6898): 646-650. DOI:10.1038/nature00939 |
| [5] |
Neil SJD, Zang T, Bieniasz PD. Tetherin inhibits retrovirus release and is antagonized by HIV-1 Vpu[J]. Nature, 2008, 451(7177): 425-430. DOI:10.1038/nature06553 |
| [6] |
Ahmad N, Venkatesan S. Nef protein of HIV-1 is a transcriptional repressor of HIV-1 LTR[J]. Science, 1988, 241(4872): 1481-1485. DOI:10.1126/science.3262235 |
| [7] |
Luciw PA, Cheng-Mayer C, Levy JA. Mutational analysis of the human immunodeficiency virus:the orf-B region down-regulates virus replication[J]. Proc Natl Acad Sci USA, 1987, 84(5): 1434-1438. DOI:10.1073/pnas.84.5.1434 |
| [8] |
Kim S, Ikeuchi K, Byrn R, et al. Lack of a negative influence on viral growth by the nef gene of human immunodeficiency virus type 1[J]. Proc Natl Acad Sci USA, 1989, 86(23): 9544-9548. DOI:10.1073/pnas.86.23.9544 |
| [9] |
Basmaciogullari S, Pizzato M. The activity of Nef on HIV-1 infectivity[J]. Front Microbiol, 2014, 5: 232. DOI:10.3389/fmicb.2014.00232 |
| [10] |
Deacon NJ, Tsykin A, Solomon A, et al. Genomic structure of an attenuated quasi species of HIV-1 from a blood transfusion donor and recipients[J]. Science, 1995, 270(5238): 988-991. DOI:10.1126/science.270.5238.988 |
| [11] |
Rosa A, Chande A, Ziglio S, et al. HIV-1 Nef promotes infection by excluding SERINC5 from virion incorporation[J]. Nature, 2015, 526(7572): 212-217. DOI:10.1038/nature15399 |
| [12] |
Usami Y, Wu YF, Göttlinger HG. SERINC3 and SERINC5 restrict HIV-1 infectivity and are counteracted by Nef[J]. Nature, 2015, 526(7572): 218-223. DOI:10.1038/nature15400 |
| [13] |
Inuzuka M, Hayakawa M, Ingi T. Serinc, an activity-regulated protein family, incorporates serine into membrane lipid synthesis[J]. J Biol Chem, 2005, 280(42): 35776-35783. DOI:10.1074/jbc.M505712200 |
| [14] |
Chande A, Cuccurullo EC, Rosa A, et al. S2 from equine infectious anemia virus is an infectivity factor which counteracts the retroviral inhibitors SERINC5 and SERINC3[J]. Proc Natl Acad Sci USA, 2016, 113(46): 13197-13202. DOI:10.1073/pnas.1612044113 |
 2017, Vol. 32
2017, Vol. 32


