扩展功能
文章信息
- 纪蕾, 陈莉萍, 沈月华, 严伟, 吴晓芳, 徐德顺, 朱晓娟, 查赟峰
- Ji Lei, Chen Liping, Shen Yuehua, Yan Wei, Wu Xiaofang, Xu Deshun, Zhu Xiaojuan, Zha Yunfeng
- 2016年浙江省湖州市急性胃肠炎病例诺如病毒感染状况及其基因型别特征
- Epidemiologic and genetic characteristics of norovirus caused acute gastroenteritis in Huzhou, Zhejiang, 2016
- 疾病监测, 2018, 33(3): 235-240
- Disease Surveillance, 2018, 33(3): 235-240
- 10.3784/j.issn.1003-9961.2018.03.016
-
文章历史
- 收稿日期:2017-12-04
诺如病毒属于杯状病毒科诺如病毒属, 分为7个基因组(genogroup, GⅠ~GⅦ), 其中GⅠ、GⅡ和GⅣ可感染人类[1]。GⅡ为引起人类感染的主要基因组。根据病毒主要衣壳蛋白(major capsid protein, VP)1的核苷酸序列差异, GⅠ和GⅡ基因组诺如病毒又进一步分为9(G Ⅰ. 1~G Ⅰ. 9)和22(GⅡ.1~GⅡ.22)个基因型[2]。目前, 诺如病毒被认为是全球范围内引起急性病毒性胃肠炎的重要病原体之一, 各年龄段人群对其普遍易感。该病毒感染剂量低、抵抗力强, 具有快速传播能力和较高传染性, 常可通过粪-口、接触及污染的水和食物等途径在学校、医院等人群聚集场所引起暴发流行, 为常见的食源性传染性疾病的病原体[3]。作为RNA病毒, 诺如病毒基因型别众多且易发生变异, 新变异株的出现往往导致新一轮诺如病毒急性胃肠炎的流行。为了解湖州市急性胃肠炎病例诺如病毒的感染状况及其流行的基因型别和变化趋势, 收集2016年湖州市急性胃肠炎病例粪便标本, 进行诺如病毒核酸检测及序列分析, 现将结果报告如下。
1 资料与方法 1.1 样本来源2016年收集湖州市食源性疾病主动监测哨点医院的急性胃肠炎病例粪便标本501份。病例纳入标准:以腹泻为主要症状, 每日排便≥3次并伴有性状改变;可伴有发热、恶心、呕吐、腹痛等症状。患者粪便以无菌容器收集, 于-70 ℃冰箱保存备用。
1.2 病毒RNA的提取取粪便少量于离心管中,加入无菌PBS缓冲液制成10%的悬液。置于涡旋振荡器充分振荡混匀后, 5 000 r /min(离心半径6 cm)离心5 min, 吸取上清液用于核酸提取。使用苏州天隆生物科技有限公司的全自动核酸提取仪和RNA提取试剂盒进行病毒RNA的提取, 具体步骤参照说明书进行。
1.3 诺如病毒核酸检测采用real-time RT-PCR方法, 利用GⅠ和GⅡ基因组特异性引物、探针对提取的病毒RNA进行诺如病毒核酸检测。PCR反应试剂为宝生物工程(大连)有限公司的One Step RTPCR Kit(货号DRR064A), PCR反应仪器为ABI公司的7300 real-time-PCR仪。PCR反应条件:42 ℃反转录10 min,95 ℃预变性10 s;95 ℃变性5 s, 55 ℃退火延伸40 s, 40个循环。Ct≤35判定为阳性。核酸检测所用引物探针参照文献[4]设计。
1.4 诺如病毒基因分型 1.4.1 衣壳蛋白区部分片段的序列测定分别采用引物G1SKF/G1SKR和CoG2F/GⅡSKR对GⅠ和GⅡ核酸阳性标本进行病毒衣壳蛋白区部分序列的扩增。扩增引物序列参考文献[5], 目的片段分别为330和387 bp。RT-PCR反应试剂为宝生物工程(大连)有限公司的One StepRNAPCR Kit(货号DRR024A)。反应条件:42 ℃反转录10 min,95 ℃预变性10 s;94 ℃变性30 s、51 ℃退火30 s、72 ℃延伸30 s, 35个循环;72 ℃再延伸10 min, 4 ℃保温。经1.5%琼脂糖凝胶电泳观察PCR结果, 阳性条带送南京金斯瑞生物科技有限公司进行测序。
1.4.2 序列系统进化分析参考文献[1]下载诺如病毒各基因型代表株序列进行分析。利用MegAlign进行序列比对和同源性分析;应用MEGA 6. 0软件构建系统进化树, 采用Neighbor-Joining法建树, 结合诺如病毒在线分型工具(http://www.rivm.nl/mpf/norovirus/typingtool)的BLAST比对结果综合判定病毒基因型别[6]。
1.5 统计学分析采用SPSS 13.0软件进行数据统计分析, 率的比较采用χ2检验, 检验水准α= 0.05。
2 结果 2.1 诺如病毒核酸检测共收集急性胃肠炎病例粪便标本501份。女性260例, 男性241例。患者最大年龄91岁, 最小11月龄。诺如病毒核酸阳性101份, 阳性率20.16%。其中GⅠ基因组阳性8份, GⅡ基因组阳性87份, GⅠ和GⅡ同时阳性6份。男、女性诺如病毒检出率分别为20.75%(50/241)和19.54%(51/260),差异无统计学意义(χ2=0.114, P=0.736)。
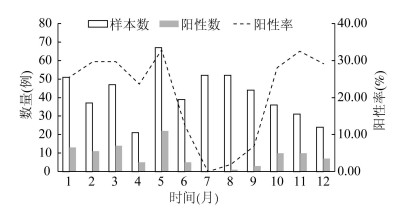
2.2 病例年龄和时间分布不同年龄组病例均检出诺如病毒,其中<5岁年龄组阳性率最高(30.77%), ≥60岁最低(9.09%), 各年龄组阳性率差异有统计学意义(χ2=15.608, P=0.016), 见表 1。在6-9月阳性率较低, 平均检出率为4.80%, 其他月份阳性率均>20.00%。诺如病毒阳性率高峰期在2-3、5、10-12月, 以5月阳性率最高(32.84%), 其次为11月(32.26%), 见图 1。
| 年龄组(岁) | 检测数(例) | 阳性数(例) | 阳性率(%) |
| < 5 | 13 | 4 | 30.77 |
| 5~ | 62 | 9 | 14.52 |
| 21~ | 131 | 34 | 25.95 |
| 31~ | 98 | 18 | 18.37 |
| 41~ | 47 | 14 | 29.79 |
| 51~ | 62 | 14 | 22.58 |
| ≥60 | 88 | 8 | 9.09 |
|
| 图 1 2016年湖州市急性胃肠炎患者诺如病毒感染时间分布 Figure 1 Time distribution of norovirus caused acute gastroenteritis cases in Huzhou, 2016 |
| |
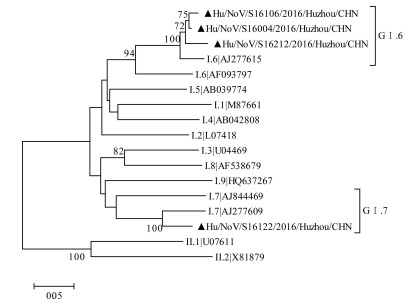
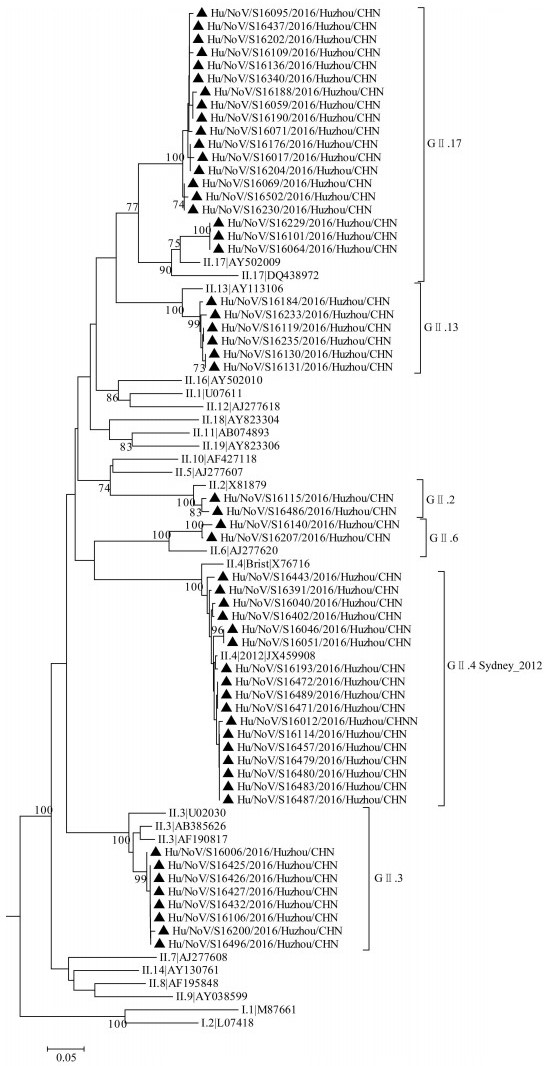
101份核酸阳性标本中共有60份成功扩增出衣壳蛋白片段。系统进化分析和在线比对分型结果显示, 2016年湖州市急性胃肠炎病例诺如病毒包括8种基因型别(图 2、3), 依次为GⅡ.17(21 / 60,35.00%)、G Ⅱ. 4 Sydney_2012(18 / 60,30.00%)、G Ⅱ. 3(7 / 60, 11.67%)、G Ⅱ. 13(6 / 60,10.00%)、GⅠ.6(3/60, 5.00%)、GⅡ.2(2/60, 3.33%)、GⅡ.6(2/60, 3.33%)、GⅠ.7(1/60, 1.67%)。其中GⅡ.17和GⅡ.4 Sydney_2012为主要基因型别。但各基因型别流行的月份不同。2-8月检出GⅡ.17占绝对优势, 其次为GⅡ.13, 分别占56.25%(18/32)和18.75%(6/32);1月和9-12月以GⅡ.4Sydney_ 2012为优势型别, 占57.14%(16/28), 其次为GⅡ.3(25.00%, 7/28), GⅡ.17居第3位(10.71%, 3/28), 其中GⅡ.3是10月检出的主要型别, 占66.66%, 见表 2。
|
| 图 2 GⅠ型诺如病毒衣壳蛋白片段的系统进化分析 Figure 2 Phylogenetic analysis based on partial capsid gene sequences of GⅠ norovirus 注:▲为湖州市检出的诺如病毒 |
| |
| 月份 | GⅡ.4 Sydney_2012 | GⅡ.17 | GⅡ.3 | GⅡ.13 | 其他 | 总计(例) | |||||||||
| 例数 | 构成比(%) | 例数 | 构成比(%) | 例数 | 构成比(%) | 例数 | 构成比(%) | 例数 | 构成比(%) | ||||||
| 1 | 4 | 57.16 | 1 | 14.28 | 1 | 14.28 | 0 | 0.00 | 1 | 14.28 | 7 | ||||
| 2 | 0 | 0.00 | 4 | 100.00 | 0 | 0.00 | 0 | 0.00 | 0 | 0.00 | 4 | ||||
| 3 | 1 | 8.33 | 5 | 41.67 | 0 | 0.00 | 3 | 25.00 | 3 | 25.00 | 12 | ||||
| 4 | 0 | 0.00 | 1 | 33.33 | 0 | 0.00 | 0 | 0.00 | 2 | 66.67 | 3 | ||||
| 5 | 1 | 12.50 | 5 | 62.50 | 0 | 0.00 | 1 | 12.50 | 1 | 12.50 | 8 | ||||
| 6 | 0 | 0.00 | 2 | 50.00 | 0 | 0.00 | 2 | 50.00 | 0 | 0.00 | 4 | ||||
| 7 | 0 | 0.00 | 0 | 0.00 | 0 | 0.00 | 0 | 0.00 | 0 | 0.00 | 0 | ||||
| 8 | 0 | 0.00 | 1 | 100.00 | 0 | 0.00 | 0 | 0.00 | 0 | 0.00 | 1 | ||||
| 9 | 3 | 100.00 | 0 | 0.00 | 0 | 0.00 | 0 | 0.00 | 0 | 0.00 | 3 | ||||
| 10 | 1 | 16.67 | 1 | 16.67 | 4 | 66.66 | 0 | 0.00 | 0 | 0.00 | 6 | ||||
| 11 | 3 | 75.00 | 0 | 0.00 | 1 | 25.00 | 0 | 0.00 | 0 | 0.00 | 4 | ||||
| 12 | 5 | 62.50 | 1 | 12.50 | 1 | 12.50 | 0 | 0.00 | 1 | 12.50 | 8 | ||||
| 合计 | 18 | 30.00 | 21 | 35.00 | 7 | 11.67 | 6 | 10.00 | 8 | 13.33 | 60 | ||||
|
| 图 3 GⅡ型诺如病毒衣壳蛋白片段的系统进化分析 Figure 3 Phylogenetic analysis based on partial capsid gene sequences of GⅡ norovirus 注:▲为湖州市检出的诺如病毒 |
| |
目前, 浙江省将诺如病毒纳入到可引起食源性疾病的特定病原体主动监测工作, 并在全省开展哨点医院监测工作, 对哨点医院收集的急性胃肠炎病例进行诺如病毒核酸检测。本调查结果显示, 诺如病毒是本地急性胃肠炎病例的重要病原体, 阳性率为20.16%,高于江苏省、新疆维吾尔自治区等地[7-8], 与全国病毒性腹泻监测点的诺如病毒检出率相近[9]。诺如病毒流行时间特征与全国大部分地区研究结果一致[9-11], 温度较高的6-9月检出率相对较低, 平均检出率为4.80%, 呈低流行状态。基因分型结果显示, 2016年本市急性胃肠炎病例的诺如病毒基因型别多样, 共8种不同基因亚型。其中GⅡ.17和GⅡ.4 Sydney_2012为主要基因型别。
2014年前, GⅡ.4型诺如病毒为全球范围内诺如病毒的流行优势型别, 一般每隔2~3年会通过变异或重组方式产生新的变异株, 新的GⅡ.4变异株一旦出现, 就会逐渐取代旧的变异株持续流行, 成为主要流行株[12-13]。2014年底, 中国、日本等亚洲国家出现新的GⅡ.17变异株, 取代了GⅡ.4 Sydeny_ 2012成为2014-2015秋冬季的诺如病毒流行优势型别。GⅡ.17流行株与以往新的GⅡ.4变异株出现后呈现持续流行的模式不同, 其在湖州地区检出后与G Ⅱ. 4Sydeny_2012变异株呈交替流行现象。2015年湖州市散发腹泻病例监测数据表明, GⅡ.17最早于2014年10月检出,后逐渐取代G Ⅱ. 4 Sydeny_2012而成为流行优势株, 但2015年9-12月, GⅡ.4 Sydeny_2012又重新成为主要型别[14]。监测结果显示, 2016年1、9-12月检出主要流行株为GⅡ.4 Sydney_2012, 其次为GⅡ.3, 而2-8月主要流行株又变为GⅡ.17, 其次为GⅡ.13。表明目前本地区诺如病毒的基因型别多样, G Ⅱ. 17和G Ⅱ. 4 Sydeny_2012存在月份交替流行现象。湖北、青岛两地也同样存在该现象[10, 15]。
除GⅡ.17和GⅡ.4 Sydeny_2012, 以往比较少见的GⅡ.13型在本次监测中的检出率也较高, 且其在2-8月检出率仅次于GⅡ.17。近年来, GⅡ.13在意大利、德国、尼泊尔和我国青岛市和湖北省[10, 15-18]等均有检出, 提示该型别具有广泛流行能力, 值得关注。此外, 2016年底广东省和我国香港等地出现了GⅡ.P16-GⅡ.2重组株引起的诺如病毒胃肠炎暴发[19-21]。诺如病毒重组株的多聚酶和衣壳蛋白区一般分属于不同基因型, 而本研究仅对病毒衣壳蛋白区进行测序分型, 故不能及时发现新的重组株。基于衣壳蛋白序列分型, 2016年哨点医院监测发现,3月和12月各检测到1株GⅡ.2, 同时在1月和12月各检出1起GⅡ.2引起的聚集性疫情, 故推测2016年GⅡ.P16-GⅡ.2重组株在本地的急性胃肠炎病例中尚未成为流行趋势。
综上所述, 湖州市急性胃肠炎病例诺如病毒流行基因型别多样且呈交替流行现象, 应进一步加强监测各型别的诺如病毒在本地的流行范围及型别分布情况, 及时发现新的流行株, 为预防和控制诺如病毒的流行提供数据支持。
作者贡献:
纪蕾 ORCID:0000-0001-6753-6465
纪蕾:论文的主要完成人, 质量控制、实验室检测工作和数据分析
陈莉萍、沈月华、严伟:样本收集、实验室检测
吴晓芳、徐德顺、朱晓娟、查赟峰:实验室检测
| [1] |
Vinjé J. Advances in laboratory methods for detection and typing of norovirus[J]. J Clin Microbiol, 2015, 53(2): 373-381. DOI:10.1128/JCM.01535-14 |
| [2] |
Kroneman A, Vega E, Vennema H, et al. Proposal for a unified norovirus nomenclature and genotyping[J]. Arch Virol, 2013, 158(10): 2059-2068. DOI:10.1007/s00705-013-1708-5 |
| [3] |
Glass RI, Parashar UD, Estes MK. Norovirus gastroenteritis[J]. N Engl J Med, 2009, 361(18): 1776-1785. DOI:10.1056/NEJMra0804575 |
| [4] |
Jotbikumar N, Lowther JA, Henshilwood K, et al. Rapid and sensitive detection of Norovirases by using TaqMan-based onestep revelse transcription-PCR assays and application to naturally contaminated shellfish samples[J]. Appl Environ Microbiol, 2005, 71(4): 1870-1875. DOI:10.1128/AEM.71.4.1870-1875 |
| [5] |
Yan HN, Yagyu F, Okitsu S, et al. Detection of norovirus(GⅠ, G Ⅱ), sapovirus and astrovirus in fecal samples using reverse transcription single-round multiplex PCR[J]. J Virol Methods, 2003, 114(1): 37-44. DOI:10.1016/j.jviromet.2003.08.009 |
| [6] |
Kroneman A, Vennema H, Deforche K, et al. An automated genotyping tool for enteroviruses and noroviruses[J]. J Clin Virol, 2011, 51(2): 121-125. DOI:10.1016/j.jcv.2011.03.006 |
| [7] |
唐震, 郑东宇, 马恺, 等. 2013-2014年江苏省食源性疾病中诺如病毒的分子流行病学分析[J]. 南京医科大学学报:自然科学版, 2016, 36(10): 1213-1217, 1236. Tang Z, Zheng DY, Ma K, et al. Molecular characteristics of norovirus(NV)in acute foodborne disease in Jiangsu province from 2013 to 2014[J]. J Nanjing Med Univ:Nat Sci, 2016, 36(10): 1213-1217, 1236. DOI:10.7655/NYDXBNS20161013 |
| [8] |
马鑫, 于烽, 刘艳, 等. 新疆2015年食源性疾病诺如病毒感染状况分析[J]. 中国卫生检验杂志, 2016, 26(19): 2834-2835. Ma X, Yu F, Liu Y, et al. Analysis of norovirus infection status in food-borne disease at 2015 in Xinjiang[J]. Chin J Health Lab Tec, 2016, 26(19): 2834-2835. |
| [9] |
张静, 常昭瑞, 孙军玲, 等. 我国诺如病毒感染性腹泻流行现状及防制措施建议[J]. 疾病监测, 2014, 29(7): 516-521. Zhang J, Chang ZR, Sun JL, et al. Infectious diarrhea epidemics caused by norovirus and its control strategy in China[J]. Dis Surveill, 2014, 29(7): 516-521. DOI:10.3784/j.issn.1003-9961.2014.07.004 |
| [10] |
李静, 张婷, 徐军强, 等. 2015年湖北地区诺如病毒流行特点及基因型别特征[J]. 中华疾病控制杂志, 2017, 21(4): 349-352, 361. Li J, Zhang T, Xu JQ, et al. Epidemic characteristics and genotypes of norovirus in Hubei province, 2015[J]. Chin J Dis Control Prev, 2017, 21(4): 349-352, 361. DOI:10.16462/j.cnki.zhjbkz.2017.04.007 |
| [11] |
汪慧, 王大虎, 陈纯, 等. 广州市腹泻监测病例诺如病毒分子流行病学特征[J]. 疾病监测, 2016, 31(10): 838-842. Wang H, Wang DH, Chen C, et al. Molecular epidemiological characteristics of norovirus in diarrhea cases in Guangzhou[J]. Dis Surveill, 2016, 31(10): 838-842. DOI:10.3784/j.issn.1003-9961.2016.10.009 |
| [12] |
Lindesmith LC, Donaldson EF, Lobue AD, et al. Mechanisms of G Ⅱ. 4 norovirus persistence in human populations[J]. PLoS Med, 2008, 5(2): e31. DOI:10.1371/journal.pmed.0050031 |
| [13] |
Tran TNH, Trainor E, Nakagomi T, et al. Molecular epidemiology of noroviruses associated with acute sporadic gastroenteritis in children:global distribution of genogroups, genotypes and G Ⅱ. 4 variants[J]. J Clin Virol, 2013, 56(3): 185-193. DOI:10.1016/j.jcv.2012.11.011 |
| [14] |
Zhang P, Chen LP, Fu Y, et al. Clinical and molecular analyses of norovirus-associated sporadic acute gastroenteritis:the emergence of GⅡ.4, Huzhou, China, 2015[J]. BMC Infect Dis, 2016, 16(1): 717. DOI:10.1186/s12879-016-2033-x |
| [15] |
赵丹, 苏志磊, 张凤, 等. 青岛市2015年成人散发腹泻病例诺如病毒感染分子流行病学特征[J]. 中华实验和临床病毒学杂志, 2017, 31(2): 104-107. Zhao D, Su ZL, Zhang F, et al. Molecular epidemiology of Norovirus infection among sporadic hospitalized adults with diarrhea in Qingdao, 2015[J]. Chin J Exp Clin Virol, 2017, 31(2): 104-107. DOI:10.3760/cma.j.issn.1003-9279.2017.02.004 |
| [16] |
Mäde D, Trübner K, Neubert E, et al. Detection and typing of norovirus from frozen strawberries involvedin a large-scale gastroenteritis outbreak in Germany[J]. Food Environ Virol, 2013, 5(3): 162-168. DOI:10.1007/s12560-013-9118-0 |
| [17] |
Medici MC, Tummolo F, Martella V, et al. Novel recombinant GⅡ. P16_GⅡ. 13 and GⅡ. P16_GⅡ.3 norovirus strains in Italy[J]. Virus Res, 2014, 188: 142-145. DOI:10.1016/j.virusres.2014.04.005 |
| [18] |
Hoa-Tran TN, Nakagomi T, Sano D, et al. Molecular epidemiology of noroviruses detected in Nepalese children with acute diarrhea between 2005 and 2011:Increaseand predominance of minor genotype GⅡ.13[J]. Infect Genet Evol, 2015, 30: 27-36. DOI:10.1016/j.meegid.2014.12.003 |
| [19] |
Kwok K, Niendorf S, Lee N, et al. Increased detection of emergent recombinant norovirus GⅡ.P16-GⅡ.2 strains in young adults, Hong Kong, China, 2016-2017[J]. Emerg Infect Dis, 2017, 23(11): 1852-1855. DOI:10.3201/eid2311.170561 |
| [20] |
Ao Y, Wang J, Ling H, et al. Norovirus G Ⅱ. P16/G Ⅱ. 2-Associated Gastroenteritis, China, 2016[J]. Emerg Infect Dis, 2017, 23(7): 1172-1175. DOI:10.3201/eid2307 |
| [21] |
Lu J, Fang L, Sun L, et al. Association of G Ⅱ. P16-G Ⅱ. 2 recombinant norovirus strain with increased norovirus outbreaks, Guangdong, China, 2016[J]. Emerg Infect Dis, 2017, 23(7): 1188-1190. DOI:10.3201/eid2307.170333 |
 2018, Vol. 33
2018, Vol. 33


