扩展功能
文章信息
- 李岩, 王晶, 张桂, 杨晶, 金东
- Li Yan, Wang Jing, Zhang Gui, Yang Jing, Jin Dong
- 2012-2014年辽宁省大连市某医院分离粪肠球菌耐药性及多位点序列分析
- Antibiotic susceptibility and multi locus sequence type of Enterococcus faecalis strains isolated from patients of a hospital in Dalian
- 疾病监测, 2018, 33(5): 391-396
- Disease Surveillance, 2018, 33(5): 391-396
- 10.3784/j.issn.1003-9961.2018.05.010
-
文章历史
- 收稿日期:2018-01-16
2. 中国疾病预防控制中心传染病预防控制所, 传染病预防控制国家重点实验室, 北京 102206
2. State Key Laboratory of Communicable Disease Prevention and Control, National Institute for Communicable Disease Control and Prevention, Chinese Center for Disease Control and Prevention, Beijing 102206, China
肠球菌是重要的院内感染病原菌,可引起尿路、腹腔、盆腔感染以及败血症等多种感染性疾病。由于其耐酸、耐盐、耐干燥等特性,可以长期存在于医院的各种环境中,引起院内感染。随着抗生素的广泛应用,肠球菌的耐药现象日益严重,造成了临床抗感染治疗的困难。根据2013-2015年CHINET的监测结果,肠球菌在我国主要地区多所教学医院临床分离株中的比例逐年升高[1-3]。肠球菌属中引起院内感染的主要是粪肠球菌和屎肠球菌。在2015年CHINET分离的粪肠球菌和屎肠球菌占肠球菌属临床分离株的92.83%,而粪肠球菌的比例为42.70%。
基于序列的分子分型方法,特别是多位点序列分型(Multilocus sequence typing,MLST)方法,广泛用于包括粪肠球菌在内的多种病原菌在长期和全球范围的数据比较分析[4]。多项基于MLST的研究表明引起院内感染的、产内酰胺酶或者是万古霉素耐药的粪肠球菌多属于某几个克隆复合(Clonal complex,CC)群,包括CC2、CC9、CC16和CC87等[5-8]。粪肠球菌感染机制复杂,有多个致病基因参与其致病的过程,目前研究较多的致病基因包括细胞溶血素(Cytolysin,cylA)、明胶酶E(Gelatinase,gelE)、表面蛋白(Enterococcal surface protein,esp)和聚集物质(Aggregation substance,asa1)4种[9-12]。
本研究对大连市某医院2012-2014年住院患者分离粪肠球菌耐药性和毒力基因携带情况进行检测,并使用MLST方法对其进行分子分型,以了解该医院住院患者分离粪肠球菌的流行型别以及耐药和携带毒力基因情况,为指导临床用药和控制院内感染提供依据。
1 材料与方法 1.1 菌株分离和鉴定收集2012-2014年大连市某医院住院患者分离的粪肠球菌,剔除同一患者分离的重复菌株。共检出粪肠球菌55株,其中2012年29株,2013年10株,2014年16株。其中21株来源于脓液及分泌物,占38.18%;18株来源于尿液,占32.73%;8株来源于血液标本,占14.55%;8株来源于其他标本(腹腔积液、胆汁、引流液、心包积液),占14.55%。采用API20 Strep生化鉴定系统(法国梅里埃公司)对纯培养物进行生化鉴定;同时使用BacteriaGen DNA试剂盒(康为世纪生物科技有限公司)按照说明书提取纯培养物的基因组核酸,进行16S rDNA序列和sodA基因序列扩增比对鉴定[13]。
1.2 药敏试验使用美国贝克曼库尔特公司Micro Scan Walk Away 40 SI全自动微生物鉴定仪及其配套的Pos MIC Panel Type 29革兰阳性菌药敏板,按照仪器和药敏板使用说明,使用快速接种法进行药敏试验。药敏结果判断标准参照美国2017年CLSI标准[14]。该药敏板中给出药敏结果判定的抗生素有13种。包括青霉素类抗生素青霉素和氨苄西林;糖肽类抗生素万古霉素;恶唑烷酮类抗生素利奈唑胺;脂肽类抗生素达托霉素;氨基糖苷类抗生素高浓度庆大霉素和高浓度链霉素;氟喹诺酮类抗生素环丙沙星和左旋氧氟沙星;四环素类抗生素四环素;氯霉素类抗生素氯霉素;大环内酯类抗生素红霉素以及安沙霉素类抗生素利福平。
1.3 毒力基因的检测使用聚合酶链式反应(PCR)方法对粪肠球菌的4种毒力因子,包括esp、gelE、cylA和asa1进行检测[15]。引物序列及产物长度见表 1。20 μl PCR反应体系,内含2×Ex Taq Mix 10 μl,10 μmol/L的正、反向引物各1 μl,模板DNA 1 μl,加无菌水至20 μl。PCR反应程序:94 ℃,5 min变性,94 ℃ 30 s→54 ℃ 30 s→72 ℃ 45 s,30个循环,72 ℃延伸10 min。阳性对照为屎肠球菌ATCC BAA-472(asa1、gelE基因阳性)和粪肠球菌ATCC 51299(esp基因阳性)。对cylA基因扩增阳性产物进行序列测定和比对以确认结果。
1.4 MLST分析粪肠球菌的MLST分析的实验方案参考E.faecalis MLST数据库(http://pubmlst.org/efaecalis/)提供的分型方案,7个管家基因gdh、gyd、pstS、gki、aroE、xpt和yqiL基因的引物序列及产物大小见表 1。PCR反应体系为50 μl,内含2 ×ExTaq Mix 25 μl,10 μmol/L的正、反向引物各2.5 μl,模板DNA 2.0 μl,加无菌水至50 μl。PCR反应程序:94 ℃ 5 min变性,94 ℃ 30 s→52 ℃ 30 s→72 ℃ 60 s,30个循环,72 ℃延伸7 min。PCR产物经纯化后,进行双向测序。序列分析用SeqMan(DNAStar Inc.,Madison,WI,USA)软件对等位基因序列进行拼接和校正,将整理好的序列提交至MLST数据库,确定每个管家基因的序列号。7个管家基因序列号的组合即为该菌株序列型别(ST)。对于未能完全匹配的序列和ST,重复3次确认无误后,将序列提交到数据库,经数据库管理者审核后获得新的等位基因序列号和ST号。应用Bionumerics 5.1软件(Applied Maths BVBA,比利时)对分离菌株的MLST型别进行分析,并构建最小生成树(minimum spanning tree)。应用eBURST V3软件构建eBURST图以确定分离菌株与数据库其他菌株的关系。应用eBURST和Bionumerics软件构建克隆群时,均以2个等位基因的差异为定义克隆群的标准。
| 基因名称 | 基因产物 | 引物序列(5'~3') | 产物大小(bp) |
| gdh-1 | Glucose-6-phosphate dehydrogenase | GGC GCA CTA AAA GAT ATG GT | 530 |
| gdh-2 | CCA AGA TTG GGC AAC TTC GTC CCA | ||
| gyd-1 | Glyceraldehydes-3-phosphatedehydrogena | CAA ACT GCT TAG CTC CAA TGG C | 395 |
| gyd-2 | CAT TTC GTT GTC ATA CCA AGC | ||
| pstS-1 | Phosphate ATP binding cassette transport | CGG AAC AGG ACT TTC GC | 583 |
| pstS-2 | ATT TAC ATC ACG TTC TAC TTG C | ||
| gki-1 | Glucokinase | GAT TTT GTG GGA ATT GGT ATG G | 438 |
| gki-2 | ACC ATT AAA GCA AAA TGA TCG C | ||
| aroE-1 | Shikimate-5-dehydrogenase | TGG AAA ACT TTA CGG AGA CAG C | 459 |
| aroE-2 | GTC CTG TCC ATT GTT CAA AAG C | ||
| xpt-1 | Xanthine phosphoribosyltransferase | AAA ATG ATG GCC GTG TAT TAG G | 456 |
| xpt-2 | AAC GTC ACC GTT CCT TCA CTT A | ||
| yiqL-1 | Acetyl-CoA acetyltransferase | CAG CTT AAG TCA AGT AAG TGC CG | 436 |
| yiqL-2 | GAA TAT CCC TTC TGC TTG TGC T | ||
| esp-F | Enterococcal surface protein | AGA TTT CAT CTT TGA TTC TTG G | 510 |
| esp-R | AAT TGA TTC TTT AGC ATC TGG | ||
| asa1-F | Aggregation substance | GCA CGC TAT TACGAACTATGA | 375 |
| asa1-R | TAA GAA AGA ACA TCACCA CGA | ||
| gelE-F | Gelatinase | TATGAC AAT GCT TTT TGG GAT | 213 |
| gelE-R | AGA TGC ACC CGA AAT AAT ATA | ||
| cylA-F | Cytolysin | ACT CGG GGA TTG ATA GGC | 688 |
| cylA-R | GCT GCT AAA GCT GCG CTT |
应用SPSS 19.0软件进行数据分析,率的比较采用χ2检验,P < 0.05为差异有统计学意义。
2 结果 2.1 菌株耐药情况55株粪肠球菌对14种常见抗生素的耐药情况见表 2。在55株粪肠球菌中除2株对14种抗生素均敏感的菌株,1株对红霉素中介,1株红霉素和利福平均中介的菌株外,其他菌株均为耐药菌株(92.72%,51/55)。在耐药菌株中有7株对1种抗生素耐药,5株对2种抗生素耐药,39株对3种及以上的抗生素耐药。对3种及以上的抗生素耐药的菌株中,有36株多重耐药菌(对3种或3种以上不同种类抗生素耐药的菌株),占菌株总数的65.45%(36/55)。
| 抗生素 | 耐药 | 中介 | 敏感 | |||||
| 菌株数 | 百分比 (%) |
菌株数 | 百分比 (%) |
菌株数 | 百分比 (%) |
|||
| 青霉素 | 4 | 7.27 | 0 | 0.00 | 51 | 92.73 | ||
| 氨苄西林 | 1 | 1.82 | 0 | 0.00 | 54 | 98.18 | ||
| 万古霉素 | 0 | 0.00 | 0 | 0.00 | 55 | 100.00 | ||
| 利奈唑胺 | 0 | 0.00 | 2 | 3.64 | 53 | 96.36 | ||
| 达托霉素 | 0 | 0.00 | 0 | 0.00 | 55 | 100.00 | ||
| 高浓度庆大霉素 | 27 | 49.09 | 0 | 0.00 | 28 | 50.91 | ||
| 高浓度链霉素 | 22 | 40.00 | 0 | 0.00 | 33 | 60.00 | ||
| 环丙沙星 | 18 | 32.73 | 8 | 14.55 | 29 | 52.73 | ||
| 左旋氧氟沙星 | 16 | 29.09 | 1 | 1.82 | 38 | 69.09 | ||
| 四环素 | 43 | 78.18 | 0 | 0.00 | 12 | 21.82 | ||
| 氯霉素 | 16 | 29.09 | 1 | 1.82 | 38 | 69.09 | ||
| 红霉素 | 40 | 72.73 | 8 | 14.55 | 7 | 12.73 | ||
| 利福平 | 23 | 41.82 | 11 | 20.00 | 21 | 38.18 | ||
55株粪肠球菌对四环素的耐药率最高,达到了78.18%(43 / 55);对红霉素的耐药率也达到了72.73%(40/55)。对利福平的耐药率达到了41.82% (23/55),而氯霉素的耐药率为29.09%(16/55)。对高浓度氨基糖苷类抗生素包括高浓度庆大霉素和高浓度链霉素均有较高的耐药率,分别达到了49.09%(27/55)和40.00%(22/55)。在32株氨基糖苷类高水平耐药肠球菌中,仅对高浓度庆大霉素耐药型菌10株,仅对高浓度链霉素耐药型菌5株,同时对高浓度庆大霉素和链霉素耐药型菌17株。本研究中的菌株对氟喹诺酮类抗生素包括环丙沙星和左旋氧氟沙星也有较高的耐药率,分别为32.73% (18/55)和29.09%(16/55)。本研究分离的菌株对青霉素类抗生素的普遍敏感,其中仅有4株对青霉素耐药(7.27%),1株对氨苄西林耐药。分离到1株对呋喃妥因中介的菌株,未发现对糖肽类和脂肽类抗生素耐药的菌株。
2.2 毒力基因检测对55株粪肠球菌进行4种常见毒力基因(cylA、gelE、esp和asa1)的携带情况检测,结果表明4种毒力基因在55株分离株中的阳性率都超过了70%。阳性率最高的为asa1基因,携带率为80.00%(44/55),其他3种基因的阳性率分别为76.36%(gelE),72.73%(cylA)和70.91%(esp)。值得一提的是,除1株不携带毒力基因的粪肠球菌外,其他菌株都携带有毒力基因;有23株粪肠球菌4种毒力基因均为阳性(41.28%)。
2.3 MLST分析55株粪肠球菌共有28个MLST型别,其中2012年共获得12个ST型,2013年10个ST型,2014年12个ST型。优势ST型为ST16(15株),ST179(9株)和ST4(4株);获得6个新的ST型(ST781~ST786)。通过构建55株粪肠球菌的最小生成树(图 1),分离菌株分为3个优势克隆群。ST16和ST179属于同一个克隆群(1个等位基因的差异),这一克隆群是55株粪肠球菌的优势克隆群,占菌株总数的43.64%;2012-2014年均分离到这一克隆群的菌株。另外一个优势的克隆群包括ST4、ST585和ST781型共6株菌,占菌株总数的10.91%。还有包括3株菌的ST21和ST22克隆群。菌株ST型别及关系见图 1。
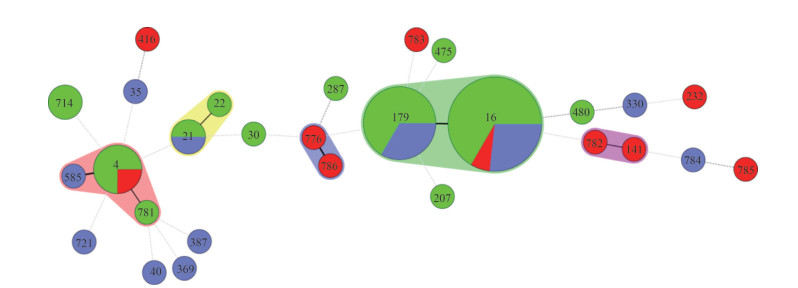
|
| 图 1 55株粪肠球菌序列型别的最小生成树 Figure 1 Minimum spanning tree of 55 E. faecalis strains 注:每个圆圈中的数字代表一种序列型别,圆圈大小代表菌株数量,圆圈中的颜色填充代表菌株的分离年代,其中绿色代表 2012年分离的菌株,红色为2013年分离的菌株,紫色为2014年分离的菌株。有阴影覆盖的圆圈代表其属于同一个克隆群。连接不同圆圈的实线代表不同序列型别等位基因的差异,线的长短代表了等位基因差异的数目;虚线则表示不同序列型别之间等位基因的差异在3个以上 |
| |
为了明确55株粪肠球菌在整个MLST数据库(https://pubmlst.org/efaecalis/)的位置及所属的克隆群,应用eBURST软件构建整个数据库的eBURST图(图 2)。其中优势的ST型ST16和ST179属于CC16,ST4属于CC4、ST21和ST22属于CC21。另外分离到ST721(CC2)、ST207(CC87)、ST30(CC30)以及ST40 (CC40)的菌株各1株。
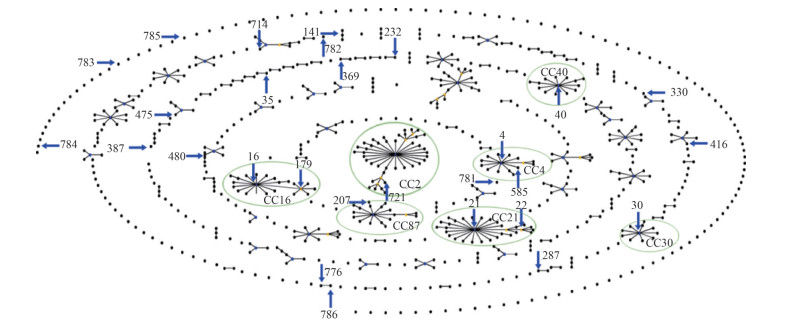
|
| 图 2 55株粪肠球菌序列型别的eBURST图 Figure 2 eBURST analysis of 55 E. faecalis strains 注:蓝色箭头标记的是本文中分离的序列型别。主要的CC群包括CC2、CC4、CC16、CC21、CC28、CC30、CC40以及CC87,用绿色的圈标记出来 |
| |
有多株相同ST的粪肠球菌有相同的耐药谱和毒力携带情况。在15株ST16型粪肠球菌中,有14株为多重耐药菌;其中7株对高浓度庆大霉素和高浓度链霉素均耐药,6株对一种高浓度氨基糖苷类抗生素耐药。在ST16型别菌株中有4株具有一致的耐药表型,均表现为高浓度氨基糖苷类-四环素-红霉素-利福平耐药,其中3株携带的毒力基因情况一致。在8株ST179粪肠球菌中有4株为多重耐药菌株,其中3株有一致的耐药表型,均表现为高浓度庆大霉素-四环素-红霉素耐药,除1株gelE基因阴性为,其他菌株均携带4种毒力基因。在4株ST4型粪肠球菌中,有3株的耐药谱和毒力携带情况完全一致,都表现为对青霉素和高浓度氨基糖苷类抗生素均耐药,同时也对氟喹诺酮类抗生素以及四环素和红霉素耐药,这3株菌均携带4种毒力基因。另外分离到1株(菌株编号13ENT27,分离自尿液标本,ST782型)对青霉素-氨苄西林-高浓度庆大霉素-高浓度链霉素-红霉素-利福平均耐药的菌株,该菌株对呋喃妥因表现为中介耐药。
3 讨论目前院内感染已经成为重要的公共卫生问题,在医院环境中患者、带菌者和健康人密切接触,容易造成病原菌特别是耐药的病原菌在人群中的传播。粪肠球菌是重要的院内感染病原菌,研究显示耐药的粪肠球菌可以在医院多个物品表面上长期存活,其在医护人员的手或手套上也可存活1 h以上[16],这种很强的环境适应性,增加了粪肠球菌引起院内感染的概率。
本研究对大连市某医院2012-2014年住院患者分离粪肠球菌的耐药情况进行了研究。研究表明在55株粪肠球菌中耐药菌株的比例达到了92.72%,而多重耐药菌的比例达到了65.45%。分离的55株粪肠球菌菌株耐药率最高的抗生素为四环素,其次分别为红霉素、高浓度庆大霉素和利福平,这与2012-2014年CHINET监测的结果相似;粪肠球菌耐药前3位的抗生素分别为红霉素、利福平和高浓度庆大霉素(未提供四环素的数据)。本研究分离的粪肠球菌对高浓度氨基糖苷类抗生素有较高的耐药率,其中对高浓度庆大霉素耐药率为49.09%,高于2014年CHINNET监测的耐药水平(31.19%)(P=0.004 6)。临床通常选择β-内酰胺类与氨基糖苷类抗生素联合使用来治疗肠球菌感染,对高浓度氨基糖苷类抗生素耐药使青霉素或糖肽类与氨基糖苷类抗生素的协同作用消失,影响治疗的效果。
基于MLST的分析发现,属于CC16的ST16和ST179粪肠球菌是大连市某医院2012-2014年的优势克隆,这一克隆群中多重耐药菌株的比例为78.26%(18/23),且有多株具有一致耐药表型和毒力基因型的菌株。在欧洲的一项对多个国家医院和社区分离粪肠球菌的研究表明,CC2、CC4、CC16、CC21、CC30、CC40以及CC87是院内流行的主要克隆群[4]。抗生素使用的选择压力促进这些克隆群成为优势克隆群,这些克隆群的流行也进一步造成了耐药性的广泛传播。在这些克隆群中CC16多重耐药粪肠球菌在医院和社区都有分布,且社区中对高浓度氨基糖苷类抗生素耐药的粪肠球菌绝大多数都属CC16[5]。在日本的一项对院内感染粪肠球菌的研究表明,在6年间临床分离的1 711株粪肠球菌中,多数多重耐药的粪肠球菌属于CC16[17]。在笔者早前的研究中也发现我国食品中分离到4株ST16型别的粪肠球菌,这些菌株对多种抗生素耐药且携带多种毒力基因[18]。动物实验表明,表达asa1、cylA和gelE基因产物的粪肠球菌菌株毒力显著高于其不表达株。多项流行病学研究也表明,致病的粪肠球菌中,esp、asa1、cylA和gelE基因的检出率均高于健康人粪便标本中的检出率[9-12]。本研究中分离到多株属于CC16、多重耐药且携带多种毒力基因的粪肠球菌,这提示需要针对这一克隆的特点进行院内感染的预防控制。另外这一克隆中存在多株耐药表型、毒力基因携带情况一致的菌株,提示是否存在这一克隆造成的院内感染暴发流行的可能。这一克隆群的多重耐药粪肠球菌在社区、医院以及食品等多种环境中都存在,造成院内感染的CC16粪肠球菌的来源值得进一步研究。除CC16克隆群菌株外,笔者也分离到了多株属于“高致病克隆”的粪肠球菌,如CC2、CC4、CC21、CC28、CC30以及CC40的菌株。特别是CC4克隆中有3株有一致的耐药表型和毒力基因携带情况,而且对青霉素和高浓度氨基糖苷类抗生素均耐药。另外笔者自尿液标本分离到1株新ST型的粪肠球菌,对青霉素-氨苄西林-高浓度庆大霉素-高浓度链霉素均耐药,对呋喃妥因表现为中介耐药。呋喃妥因常用于肠球菌尿路感染的治疗,这株对呋喃妥因中介的菌株也需要引起重视。
作者贡献:
李岩 ORCID:0000000158206237
李岩:菌株分离,MLST分析,文章撰写
王晶:菌株鉴定,核酸提取
张桂:药敏实验
杨晶:毒力基因检测,数据整理
金东:实验设计,MLST数据整理,文章修改指导
| [1] |
胡付品, 朱德妹, 汪复, 等. 2013年中国CHINET细菌耐药性监测[J]. 中国感染与化疗杂志, 2014, 14(5): 365-374. Hu FP, Zhu DM, Wang F, et al. CHINET 2013 surveillance of bacterial resistance in China[J]. Chin J Infect Chemother, 2014, 14(5): 365-374. DOI:10.3969/j.issn.1009-7708.2014.05.001 |
| [2] |
胡付品, 朱德妹, 汪复, 等. 2014年CHINET中国细菌耐药性监测[J]. 中国感染与化疗杂志, 2015, 15(5): 401-410. Hu FP, Zhu DM, Wang F, et al. CHINET 2014 surveillance of bacterial resistance in China[J]. Chin J Infect Chemother, 2015, 15(5): 401-410. DOI:10.3969/j.issn.1009-7708.2015.05.001 |
| [3] |
胡付品, 朱德妹, 汪复, 等. 2015年CHINET细菌耐药性监测[J]. 中国感染与化疗杂志, 2016, 16(6): 685-694. Hu FP, Zhu DM, Wang F, et al. Report of CHINET antimicrobial resistance surveillance program in 2015[J]. Chin J Infect Chemother, 2016, 16(6): 685-694. DOI:10.16718/j.1009-7708.2016.06.003 |
| [4] |
Aanensen DM, Spratt BG. The multilocus sequence typing network:mlst[J]. Nucleic Acids Res, 2005, 33(Suppl 2): W728-33. DOI:10.1093/nar/gki415 |
| [5] |
Kuch A, Willems RJ, Werner G, et al. Insight into antimicrobial susceptibility and population structure of contemporary human Enterococcus faecalis isolates from Europe[J]. J Antimicrob Chemother, 2012, 67(3): 551-558. DOI:10.1093/jac/dkr544 |
| [6] |
Freitas AR, Novais C, Ruiz-Garbajosa P, et al. Clonal expansion within clonal complex 2 and spread of vancomycin-resistant plasmids among different genetic lineages of Enterococcus faecalis from Portugal[J]. J Antimicrob Chemother, 2009, 63(6): 1104-1111. DOI:10.1093/jac/dkp103 |
| [7] |
Kawalec M, Pietras Z, Daniłowicz E, et al. Clonal structure of Enterococcus faecalis isolated from Polish hospitals:characterization of epidemic clones[J]. J Clin Microbiol, 2007, 45(1): 147-153. DOI:10.1128/JCM.01704-06 |
| [8] |
Willems RJL, Hanage WP, Bessen DE, et al. Population biology of Gram-positive pathogens:high-risk clones for dissemination of antibiotic resistance[J]. FEMS Microbiol Rev, 2011, 35(5): 872-900. DOI:10.1111/j.1574-6976.2011.00284.x |
| [9] |
Haas W, Gilmore MS. Molecular nature of a novel bacterial toxin:the cytolysin of Enterococcus faecalis[J]. Med Microbiol Immunol, 1999, 187(4): 183-190. DOI:10.1007/s004300050091 |
| [10] |
Gaspar F, Teixeira N, Rigottier-Gois L, et al. Virulence of Enterococcus faecalis dairy strains in an insect model:the role of fsrB and gelE[J]. Microbiology, 2009, 155(Pt 11): 3564-3571. DOI:10.1099/mic.0.030775-0 |
| [11] |
Shankar V, Baghdayan AS, Huycke MM, et al. Infection-derived Enterococcus faecalis strains are enriched in esp, a gene encoding a novel surface protein[J]. Infect Immun, 1999, 67(1): 193-200. |
| [12] |
Galli D, Lottspeich F, Wirth R. Sequence analysis of Enterococcus faecalis aggregation substance encoded by the sex pheromone plasmid pAD1[J]. Mol Microbiol, 1990, 4(6): 895-904. DOI:10.1111/j.1365-2958.1990.tb00662.x |
| [13] |
Poyart C, Berche P, Trieu-Cuot P. Characterization of superoxide dismutase genes from gram-positive bacteria by polymerase chain reaction using degenerate primers[J]. FEMS Microbiol Lett, 1995, 131(1): 41-45. DOI:10.1016/0378-1097(95)00232-T |
| [14] |
Clinical and Laboratory Standards Institute. M100S-S27 Performance standards for antimicrobial susceptibility testing[S]. Wayne, Pennsylvania: CLSI, 2017.
|
| [15] |
Vankerckhoven V, van Autgaerden T, Vael C, et al. Development of a multiplex PCR for the detection of asa1, gelE, cylA, esp, and hyl genes in enterococci and survey for virulence determinants among European hospital isolates of Enterococcus faecium[J]. J Clin Microbiol, 2004, 42(10): 4473-4479. DOI:10.1128/JCM.42.10.4473-4479.2004 |
| [16] |
Noskin GA, Stosor V, Cooper I, et al. Recovery of vancomycin-resistant enterococci on fingertips and environmental surfaces[J]. Infect Control Hosp Epidemiol, 1995, 16(10): 577-581. DOI:10.2307/30141097 |
| [17] |
Kudo M, Nomura T, Yomoda S, et al. Nosocomial infection caused by vancomycin-susceptible multidrug-resistant Enterococcus faecalis over a long period in a university hospital in Japan[J]. Microbiol Immunol, 2014, 58(11): 607-614. DOI:10.1111/1348-0421.12190 |
| [18] |
杨晶, 王红, 张桂, 等. 自贡市肉及其制品中粪肠球菌耐药性毒力基因和多位点序列分析[J]. 中国食品卫生杂志, 2017, 29(3): 277-282. Yang J, Wang H, Zhang G, et al. Antimicrobial susceptibility, virulence genes and multilocus sequence typing of Enterococcus faecalis strains isolated from meat and meat products in Zigong city[J]. Chin J Food Hyg, 2017, 29(3): 277-282. DOI:10.13590/j.cjfh.2017.03.005 |
 2018, Vol. 33
2018, Vol. 33


