扩展功能
文章信息
- 王宇萌, 梁莹, 张恩民, 马娜, 申小娜, 蔡虹, 张志凯, 夏连续, 梁未丽, 代瑞霞, 李伟
- Wang Yumeng, Liang Ying, Zhang Enmin, Ma Na, Shen Xiaona, Cai Hong, Zhang Zhikai, Xia Lianxu, Liang Weili, Dai Ruixia, Li Wei
- 青藏高原喜马拉雅旱獭鼠疫疫源地鼠疫耶尔森菌遗传特征分析
- Genetic analysis of Yersinia pestis strains isolated from Marmota himalayana plague focus of Qinghai-Tibet Plateau
- 疾病监测, 2018, 33(6): 457-462
- Disease Surveillance, 2018, 33(6): 457-462
- 10.3784/j.issn.1003-9961.2018.06.005
-
文章历史
- 收稿日期:2018-02-13
2. 青海省地方病预防控制所, 青海 西宁 811602
2. Qinghai Endemic Disease Control Institute, Xining 811602, Qinghai, China
鼠疫是由鼠疫耶尔森菌(Yersinia pestis,鼠疫菌)引起的一类人兽共患自然疫源性疾病,历史上数次大流行将鼠疫菌传播到世界各地,给人类社会带来巨大灾难[1]。我国鼠疫自然疫源地面积广阔,类型多样,分布在至少全国19个省份的305个县(市、旗)[2]。目前,青藏高原已发现两种类型鼠疫自然疫源地,即1954年发现的喜马拉雅旱獭鼠疫自然疫源地和1997年发现的青海田鼠鼠疫自然疫源地[3-5]。其中,喜马拉雅旱獭鼠疫自然疫源地分布在青海、西藏、甘肃、新疆维吾尔自治区(新疆)地区的75个县(市)。青海田鼠鼠疫自然疫源地分布在四川省石渠县和青海省称多县。
目前,大多鼠疫菌基因组已经测序完成,其中包括1株分离自青藏高原那曲地区的喜马拉雅旱獭疫源地菌株Z176003 [6],既往Shen等[6]研究将世界范围内8株已测序的鼠疫菌进行比对,将鼠疫菌的基因组划分成61个板块,并用61个板块缺失和排列的特征对鼠疫菌进行分型发现,菌株Z176003缺失了一段独特的序列。利用BLAST软件将Z176003菌株与61个板块比对发现,Z176003缺失序列即编号第59号板块,且发现在该菌株中第58号板块发生了位移,致使在该菌株中第57板块与第60板块直接相连[7]。为确定该缺失是否为那曲、比如地区鼠疫菌的共同特有特征,本研究采用了板块重排、差异区段分析(different region,DFR)、间区规律短回文重复(clustered regularly interspaced short palindromic repeats,CRISPR)、多位点可变数目串联重复序列分析(multiple - locus variable - number tandem repeat analysis,MLVA)4种基于PCR技术的分子分型方法对所有分离自那曲、比如地区的青藏高原喜马拉雅旱獭鼠疫疫源地菌株,以及一些分离自青藏高原和四川省、云南省鼠疫自然疫源地的98株鼠疫菌进行基因组分型研究和比较,确定该类型菌株在我国的地理分布。
1 材料与方法 1.1 菌株来源选取1954-2012年分离自青藏高原地区的98株鼠疫菌代表菌株,其中76株菌分离自喜马拉雅旱獭鼠疫自然疫源地,19株鼠疫菌分离自青海田鼠鼠疫自然疫源地,3株分离自云南省剑川和玉龙鼠疫疫源地。菌株分离地见图 1。
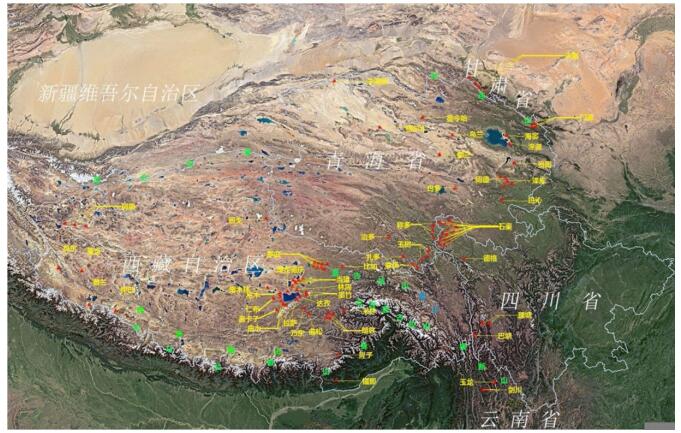
|
| 图 1 98株鼠疫菌的地理分布 Figure 1 Geographic distribution of 98 Y. pestis strains |
| |
采用以PCR技术和序列测定为核心的板块重排、CRISPR、DFR、MLVA 4种鼠疫菌型别鉴定方法。在Z176003全基因组序列的基础上利用Primer 5.0软件对第57、58、59、60板块的连接部位进行引物设计,并用已测序菌株验证引物。重排研究引物信息见表 1。CRISPR、DFR、MLVA引物序列依次参照文献[8-10],由北京天一辉远生物科技有限公司合成。
| 板块重排分型 | 在CO92序列中的位置 | 引物序列(5′~3′) | 产物长度(kbp) | 退火温度(℃) | |
| A | MF | 4417256~4417280 | ATTT GAG CAG CGA GTG AAG CCA GAC | 2 | 63 |
| MR | 4419215~4419194 | TGA ACA GCC GAT TGA CGC CAG A | |||
| B | MF | 4519897~4519920 | ACA TCG TGC CGT GTA TGC TCC TCA | 2 | 63 |
| MR | 4522022~4521998 | CGT GGC TTG CTA CTT TCA CTG TTG G | |||
| C | MF | 4564017~4564038 | TCC GAT GAA CCA ACC GCC AAT G | 2 | 63 |
| MR | 4565772~4565751 | TGA GCC ACC GAG TGA TGC CAG A | |||
| D | MF | 4417258~4417280 | TTG AGC AGC GAG TGA AGC CAG AC | 2 | 63 |
| MR | 4565772~4565751 | TGA GCC ACC GAG TGA TGC CAG A | |||
利用BioNumerics V5.10软件,采用效用均等的分类资料分析方法(Unweighted Pair Group Method with Arithmeticmeans,UPGMA)对数据进行分析。应用MLVA(14+12)方法的26个指标聚类时,为加强该分级分型法中14个低变异度指标在鼠疫种群中的区分能力,减弱高变异度的12个指标对聚类关系的影响,将第一级分型14个指标的权重设为2,用于种内溯源分析的12个指标权重设为1。
2 结果 2.1 板块重排和DFR分型实验结果1976-2012年分离自西藏自治区(西藏)那曲和比如地区的9株鼠疫菌(包括所有收集菌株)的扩增结果全部与测序菌株Z176003一致,表明第57与60板块相连,该特殊排列模式在那曲和比如地区的菌株中比较普遍。而分离自其他地区的鼠疫菌未发现与Z176003菌株一致的扩增结果,说明该板块排列模式为那曲和比如地区菌株所特有的排列模式,见表 2。
98株菌应用MLVA(14+12)方法的26个指标聚类,为区分该策略中14个低变异度指标在鼠疫种群中的区分能力,这14个指标权重设为2,另外12个指标权重设为1。聚类结果显示,98株实验菌株在基因型上被分为4大类,对比菌株的分离地点发现,4类基因型分别分布在青藏高原的冈底斯山一带、祁连山一带、那曲地区、称多和石渠地区,为4个青藏高原田鼠鼠疫疫源地的亚型。在实验中,那曲和比如地区的菌株前14个数目可变串联重复序列(variable number of tandem repeats,VNTR)位点的重复数相同且与Li等[10]实验结果一致,在后12个VNTR位点中,M22、M34、N1606、N2117位点的重复数发生了变化,减少或增加了1~5个重复数不等,说明菌株在传入那曲和比如地区后持续进化,VNTR位点在不断突变。
DFR方法与板块重组相似,利用23对引物对菌株进行分型,将98株菌分为9种型别,其中那曲和比如地区的菌株被分为单独一类,即G06型别。其他实验菌株分别为G05、G07、G08、G10、G14等多种型别[11]。G14型主要分布在称多和石渠的田鼠鼠疫疫源地,G10型主要分布在冈底斯山型疫源地,G08型分布在祁连山型鼠疫疫源地,G07和G05型分布在祁连山和巴颜喀拉山一带的鼠疫疫源县。将DFR实验阴性结果的扩增片段与板块排列中缺失的第59板块的序列进行了比对显示,DFR14和DFR22扩增的产物与第59板块有重合。
CRISPR序列在98株实验菌株中对应YPa、YPb和YPc,有9种排列(型别),见图 2。其中,那曲和比如地区的CRISPR排列为在3个位点排列依次为a1- a2-a3-a4-a5-a6-a7、b1-b2-b3-b4、c1-c2-c3。与包括青海省的10株、四川省的4株、云南省的1株、西藏的5株鼠疫菌的CRISPR类别一致,CRISPR方法不能区分那曲地区菌株。

|
| 图 2 98株菌MLVA(14+12)聚类结果 Figure 2 Phylogenetic tree of 98 Y. pestis strains by MLVA(14+12) 注:DFR和CRISPR的实验结果作为菌株信息呈现在图中 |
| |
随着基因组学技术的发展,多种分析技术被应用于鼠疫菌的分析研究,各技术均有其优势和缺点[12]。鼠疫菌的染色体和质粒中存在大量掺入序列(IS),如IS100、IS1661、IS1541、IS285。这些插入序列占整个鼠疫基因组的3.7%(以CO92为例)。使鼠疫菌染色体DNA具有高度的可塑性,重组、片段获得和缺失、基因失活等现象普遍。板块重排指鼠疫菌染色体中DNA区段的排列顺序和方向发生变异,导致不同区段中所载荷基因的位置、功能及相互关系的变化[13]。鼠疫菌染色体的61个板块相对独立,板块内部基因排列十分稳定,而板块间的相对位置在不同菌株的染色体上各不相同[7]。这些特征可被用于鼠疫菌的种群结构研究。DFR为细菌基因组在进化过程中表现出存在或缺失的片段,这种存在或缺失反映出鼠疫菌在进化过程中横向基因的转移情况。鼠疫菌基因组包含23个差异片段,基于此基因组型的分布具有明显的疫源地特异性,多数疫源地拥有占优势数量的基因组型,可用于鼠疫菌种群关系和微进化分析,反映鼠疫菌在自然界适应性进化及其与生态适应的关系[9, 11, 14]。这两种分型方法实际说明的是一类性质的问题,即由插入序列引起的DNA大片段的缺失和重排。既往单核苷酸的多态性(SNP)研究认为那曲和比如地区的菌株型属于古典型鼠疫菌1.IN群[15]。本研究那曲和比如菌株缺失的第59板块位于CO92全基因组序列4565718~4610718位置,全长45 001 bp,上游插入序列和下游插入序列均为IS1661。可能是导致板块消失的原因之一。59板块内包含33个基因,其基因产物有二元调控系统、氨基酸酶、调控蛋白、膜蛋白、糖转运蛋白、离子转运系统、菌毛蛋白和木酮糖激酶等[16]。
DFR方法用23个差异片段可以很好地区分鼠疫菌的多种基因型别,在灵活度、精确度上优于板块重排方法,但板块重排可能通过更少的扩增来判定基因型别,其应用需进一步研究。
CRISPR为一类有同向重复序列(direct repeat,DR)和将其分隔开的间区序列(spacer)构成的重复序列,广泛分布于原核生物基因组中。CRISPR位点可作为细菌分型和进化分析的理想分子靶标[17]。鼠疫菌基因组中存在3个CRISPR位点,CRISPR位点的spaces分布可用于鼠疫菌的分型研究[8, 17],该方法操作简单快速、结果稳定、重现性好。本研究CRISPR方法将98株菌分为9种类型,来自于四川石渠和青海称多的青海田鼠型菌株单独为一个型别,但那曲和比如地区菌株的间区序列的分布未能与其他同西藏同群的36株菌区分,该方法对于菌株的分辨力不如DFR法。
MLVA通过区分基因组上多个具有多态性的VNTR的重复数来区分不同菌株。国内杨瑞馥[12]团队发展的MLVA(14+12)测序利用两组VNTR的组合,即利用14个变异度低的指标组合(MLVA14)对鼠疫种群进行划分,其分类效果类同于基因组SNP分型方法,另增加12个高变异度的VNTR指标(MLVA12)可用于鼠疫菌的溯源调查,因简便快捷,适合鼠疫监测实验室开展溯源调查。本研究中,该方法将实验菌株分为4大类,与菌株的地理分布一致,即冈底斯山型、青海田鼠型、祁连山型和那曲比如型。在每类中,菌株又分为许多亚种,该分型方法非常精准,其结果可用于观察同类别菌株的细微遗传进化差别。那曲和比如等多地菌株均有再进化现象,呈现遗传多态性。
就那曲和比如地区的菌株而言,有其独特的基因型别,可能归因于该疫源地菌株的特殊地理占位,该地区地处念青唐古拉山脉与唐古拉山脉交汇处,高山阻断使该地区的菌株在一次偶然传入后,经过自然选择,形成了适应该地区的基因型别,该型别很难输出,也很难有外来菌株再次偶然传入,久而久之其地理局限性导致该地区菌株特有的基因型稳定遗传,并保守在地区内,可见地理特征对菌株的基因型分布有一定的划分和阻断作用,更多基因型与地理特征关系需更多研究验证。
志谢: 中国科学院地理和自然资源研究所陈奕先提供青藏高原地形图,并取得版权同意;云南省地方病防治所王鹏、四川和西藏疾病预防控制中心鼠鼠疫防治工作技术人员提供菌株,在此感谢!作者贡献:
王宇萌 ORCID:0000-0002-2651-665x
王宇萌:实验操作,数据分析,文章撰写
梁莹、张恩民、马娜、申小娜、蔡虹、张志凯:实验操作与数据分析技术指导
夏连续、梁未丽:论文写作指导
代瑞霞、李伟:课题经费资助
| [1] |
卫生部卫生应急办公室, 中国疾病预防控制中心. 鼠疫防控应急手册[M]. 北京: 北京大学医学出版社, 2009: 1-2. Ministry of Health Emergency Response Office, China Center for Disease Control and Prevention. Emergency handbook for plague prevention and control[M]. Beijing: Peking University Medical Press, 2009: 1-2. |
| [2] |
张贵军, 段天一, 邵奎东, 等. 全国2015年鼠疫监测结果[J]. 中国地方病防治杂志, 2016, 31(增刊1): 1-7. Zhang GJ, Duan TY, Shao KD, et al. National plague surveillance results 2015[J]. Chin J Endem Dis Prev, 2016, 31(Suppl 1): 1-7. |
| [3] |
方喜业. 中国鼠疫自然疫源地[M]. 北京: 人民卫生出版社, 1990: 1-291. Fang XY. China's plague natural foci[M]. Beijing: People's Medical Publishing House, 1990: 1-291. |
| [4] |
刘振才, 海荣, 李富忠, 等. 青藏高原青海田鼠鼠疫自然疫源地的发现与研究[J]. 中国地方病防治杂志, 2001, 16(6): 321-327. Liu ZC, Hai R, Li FZ, et al. The discovery and study of Microtus fuscus nature plague foci in Qinghai-Tibet Plateau[J]. Chin J Endem Dis Control, 2001, 16(6): 321-327. DOI:10.3969/j.issn.1001-1889.2001.06.001 |
| [5] |
王祖郧, 罗松达卫, 于晓涛, 等. 青海省青海田鼠鼠疫自然疫源地的发现与研究[J]. 中国地方病防治杂志, 2004, 23(1): 69-72. Wang ZY, Luo SDW, Yu XT, et al. The discovery and research of Microtus fuscus plague natural foci in Qinghai province[J]. Chin J Endemiol, 2004, 23(1): 69-72. DOI:10.3760/cma.j.issn.1000-4955.2004.01.024.issn.1000-4955.2004.01.024 |
| [6] |
Shen XN, Wang Q, Xia LX, et al. Complete genome sequences of Yersinia pestis from natural foci in China[J]. J Bacteriol, 2010, 192(13): 3551-3552. DOI:10.1128/JB.00340-10 |
| [7] |
Liang Y, Hou XX, Wang YH, et al. Genome rearrangements of completely sequenced strains of Yersinia pestis[J]. J Clin Microbiol, 2010, 48(5): 1619-1623. DOI:10.1128/JCM.01473-09 |
| [8] |
Cui YJ, Li YJ, Gorgé O, et al. Insight into microevolution of Yersinia pestis by clustered regularly interspaced short palindromic repeats[J]. PLoS One, 2008, 3(7): e2652. DOI:10.1371/journal.pone.0002652 |
| [9] |
杨晓艳, 魏柏青, 靳娟, 等. 中国鼠疫耶尔森菌差异区段分型及其地理分布特征[J]. 中华流行病学杂志, 2014, 35(8): 943-948. Yang XY, Wei BQ, Jin J, et al. Regional genotyping and the geographical distribution regarding Yersinia pestis isolates in China[J]. Chin J Epidemiol, 2014, 35(8): 943-948. DOI:10.3760/cma.j.issn.0254-6450.2014.08.015 |
| [10] |
Li YJ, Cui YJ, Cui BZ, et al. Features of variable number of tandem repeats in Yersinia pestis and the development of a hierarchical genotyping scheme[J]. PLoS One, 2013, 8(6): e66567. DOI:10.1371/journal.pone.0066567 |
| [11] |
Li YJ, Dai EH, Cui YJ, et al. Different region analysis for genotyping Yersinia pestis isolates from China[J]. PLoS One, 2008, 3(5): e2166. DOI:10.1371/journal.pone.0002166 |
| [12] |
杨瑞馥. 基因分析技术在鼠疫耶尔森菌中的应用[J]. 中华预防医学杂志, 2015, 49(1): 1-2. Yang RF. Genotyping techniques in Yersinia pestis[J]. Chin J Prev Med, 2015, 49(1): 1-2. DOI:10.3760/cma.j.issn.0253-9624.2015.01.001 |
| [13] |
梁莹, 张志凯, 海荣, 等. 中国鼠疫耶尔森菌染色体结构特征的研究[J]. 中华地方病学杂志, 2014, 33(5): 479-484. Liang Y, Zhang ZK, Hai R, et al. Chromosomal structural features of Yersinia pestis isolated from China[J]. Chin J Endemiol, 2014, 33(5): 479-484. DOI:10.3760/cma.j.issn.2095-4255.2014.05.003 |
| [14] |
朱俊杰, 王鹏, 李伟, 等. 云南省鼠疫菌株差异片段基因分型及其流行病学特征[J]. 中华地方病学杂志, 2013, 32(6): 599-601. Zhu JJ, Wang P, Li W, et al. Genotyping of Yersinia pestis by different regions and its epidemiological characteristics in Yunnan province[J]. Chin J Endemiol, 2013, 32(6): 599-601. DOI:10.3760/cma.j.issn.2095-4255.2013.06.004 |
| [15] |
Morelli G, Song YJ, Mazzoni CJ, et al. Phylogenetic diversity and historical patterns of pandemic spread of Yersinia pestis[J]. Nat Genet, 2010, 42(12): 1140-1143. DOI:10.1038/ng.705 |
| [16] |
周冬生, 韩延平, 宋亚军, 等. 鼠疫耶尔森菌基因组进化与生态位适应研究[J]. 解放军医学杂志, 2004, 29(3): 204-210. Zhou DS, Han YP, Song YJ, et al. DNA microarray analysis of genome dynamics in Yersinia pestis:insight into bacterial genome microevolution and niche adaptation[J]. Med J Chin People's Liberat Army, 2004, 29(3): 204-210. DOI:10.3321/j.issn:0577-7402.2004.03.005.issn:0577-7402.2004.03.005 |
| [17] |
Pourcel C, Salvignol G, Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies[J]. Microbiology, 2005, 151(3): 653-663. DOI:10.1099/mic.0.27437-0 |
 2018, Vol. 33
2018, Vol. 33


