扩展功能
文章信息
- 黄世腾, 杨瑞军, 吕磊, 陈旭富, 叶承华, 万圣
- Huang Shiteng, Yang Ruijun, Lyu Lei, Chen Xufu, Ye Chenghua, Wan Sheng
- 2015-2017年浙江省衢州市H3N2亚型流感病毒血凝素基因分子进化特征分析
- Molecular characteristics of hemagglutinin gene of influenza A(H3N2)virus in Quzhou, 2015-2017
- 疾病监测, 2018, 33(6): 483-488
- Disease Surveillance, 2018, 33(6): 483-488
- 10.3784/j.issn.1003-9961.2018.06.010
-
文章历史
- 收稿日期:2017-12-26
血凝素(Hemagglutinin,HA)和神经氨酸酶(Neuramindase,NA)基因是流感病毒基因片段中变异频率最高的两个基因,其编码的HA和NA也是流感病毒主要的两种抗原,目前发现有HA亚型18种(H1~H18)和NA亚型11种(N1~N11)[1];其中,HA作为流感病毒的主要表面抗原,在病毒与宿主受体识别过程中起着重要作用,是宿主是否可被病毒感染的先决条件;并且其基因变异不仅能够改变其宿主特异性,从而实现跨物种间转播,同时由于其抗原性不断变异,使病毒实现免疫逃逸,导致流感病毒能够不断引起流行。在过去的数十年间,甲型流感病毒曾多次引发全球疫情,其中,H1N1和H3N2是最主要的型别[2-4],给人类健康造成严重危害。通过近年来的监测数据显示,2013年以来衢州市H3N2亚型一直处于优势流行状态,冬春季节为流行高峰,并且在2017年3月和8月出现双峰流行;本研究选取衢州市2015-2017年具有代表性的H3N2亚型流感病毒18株,对其HA基因序列的分子进化特征进行分析,了解病毒的变异趋势,旨在为流感的防控提供科学依据。
1 材料与方法 1.1 材料 1.1.1 标本来源收集2015年1月至2017年12月由衢州市流感监测哨点医院(衢州市人民医院)每周采集发病3 d内未使用抗病毒药物的流感样病例(ILI)鼻咽拭子标本;具体标准及相关方法参照《全国流感监测方案(2010年版)》 [5]。
1.1.2 主要试剂MagMAXTM - 96 Viral RNA Isolation Kit(批号:00324778)核酸提取试剂盒购自ABI公司,One - step PrimeScriptTMRT - PCR(批号:AK7801)试剂盒购自TaKaRa公司,MEM培养液、胎牛血清及TPCK-胰酶均购于GIBCO公司。
1.2 方法 1.2.1 病毒分离与鉴定核酸检测为阳性的标本,接种至MDCK细胞进行流感病毒的分离,分离毒株采用红细胞凝集试验和凝集抑制试验进行鉴定[5]。
1.2.2 HA基因PCR扩增及测序选取不同时间段的H3N2亚型流感毒株18株,采用核酸提取试剂盒提取病毒核酸;HA基因的PCR扩增采用One-step RT-PCR进行,扩增引物及反应条件参照文献[6],引物由上海伯杰生物科技有限公司合成,扩增产物经纯化后送上海伯杰生物科技有限公司进行双向测序。
1.2.3 HA基因分析测序结果通过DNAStar V7.10生物软件中的SeqMan进行拼接,获得HA基因全长序列,利用MEGA 6.0软件将分离毒株序列与世界卫生组织推荐的北半球2015-2016年度流感疫苗株A/Switzerland/9715293/2013和2016-2018年度流感疫苗株A/Hong Kong/4801/2014(北半球2016-2017年度疫苗株,也是2017-2018年度疫苗株)进行核苷酸和氨基酸比对分析,并构建HA基因系统进化树;利用在线分析软件NetNGlyc 1.0 Server(http://www.cbs.dtu.dk/services/NetNGlyc)对HA的氨基酸序列进行糖基化位点分析。H3N2疫苗株及相关代表毒株的HA基因序列均来自GISAID和GenBanK。
2 结果 2.1 病毒分离结果2015年1月至2017年12月衢州市流感网络实验室共分离出流感病毒150株;各年度分离毒株数分别为36、44和70株,其中甲型H3N2毒株数分别为22株(61.11%)、18株(40.91%)和37株(52.86%);3个年度中,甲型H3N2均为优势毒株,并且在2017年夏季保持在较高水平流行,呈现明显流行高峰。
2.2 同源性分析选取的18株H3N2亚型流感病毒HA基因的核苷酸和氨基酸同源性分别为97.36%~100.00%和96.76%~100.00%,2015年分离株与WHO推荐的北半球2015-2016年度疫苗株A/ Switzerland/9715293/2013核苷酸和氨基酸同源性分别97.96%~99.82%和97.85%~100.00%,2016年和2017年分离株与北半球2016-2018年度疫苗株A/ Hong Kong/4801/ 2014核苷酸和氨基酸同源性分别为98.63%~99.47%和98.39%~99.82%。2015-2017年分离株与同年度疫苗株HA基因的核苷酸平均遗传距离分别为0.008 2、0.007 1和0.011 2,氨基酸平均遗传距离分别为0.009 2、0.003 1和0.010 0。
2.3 HA基因进化分析对分离的18株H3N2亚型流感病毒HA基因序列构建系统进化树(图 1),发现2015-2017年选取的分离株均属于3C系,但支系发生了分化,从2015年的3C.3a支系,逐渐转变为3C.2a支系;其中,2015年分离的6株毒株中前期4株为3C.3a支系,后期2株(A/Zhejiangkecheng/1591/2015和A/Zhejiangkecheng/1692/2015)为3C.2a支系,与同期2015-2016年度疫苗株A/Switzerland/9715293/ 2013(3C.3a支系)的遗传距离较远;2016年和2017年分离株与疫苗株A/Hong Kong/4801/2014均为3C.2a支系,但相比2016年,2017年分离株与疫苗株A/ Hong Kong/4801/2014的平均遗传距离相对较远。
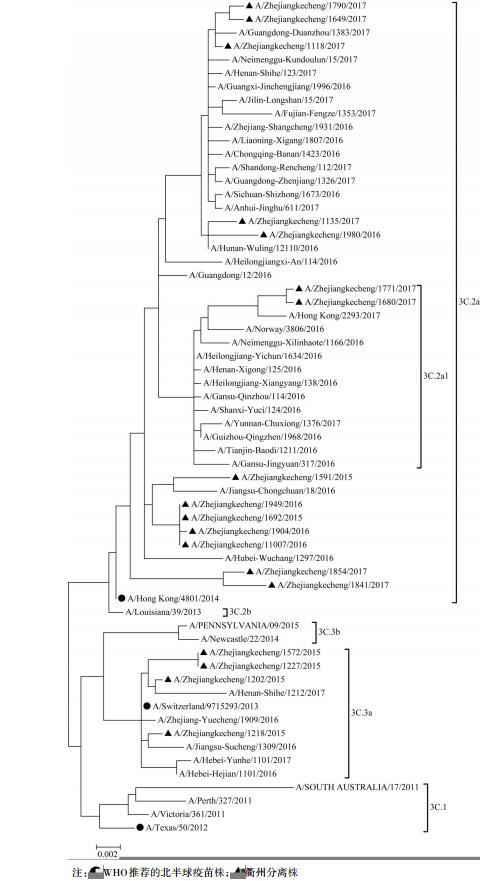
|
| 图 1 2015-2017年衢州市H3N2亚型流感病毒HA基因系统进化树 Figure 1 Phylogenetic tree of HA genes of influenza A(H3N2)virus in Quzhou 2015-2017 |
| |
18株H3N2亚型流感病毒分离株HA基因序列全长均为1 701 bp,编码566个氨基酸;其HA蛋白氨基酸序列与同年度疫苗株比较,变异位点共涉及流感病毒HA蛋白的5个抗原表位(包括A、B、C、D和E区),其中变异频率较高的位点主要有N121K、T131K、R142G/S/K、S144N/K和R261Q(按去信号肽序列排序)。2015年分离的6株毒株与2015 - 2016年度疫苗株A/Switzerland/9715293/2013对比,同时存在2株及以上毒株的氨基酸位点变异有11处,9处位于HA1区,2处位于HA2区,其中抗原表位变异位点涉及有A(S138A、G142S/R、N144S)、B(A128T、S159Y、K160T)及C(Q311H),并且同时涉及3个抗原表位的有2株;2016年和2017年分离的12株毒株与2016-2018年度疫苗株A/Hong Kong/4801/2014对比,同时存在2株及以上毒株的氨基酸位点变异有14处,12处位于HA1区,2处位于HA2区,其中抗原表位变异位点涉及有A(T131K、T135N/K、R142K/G、S144K、R150K)、B(S198P)、C(H311Q)、D(N121K、N171K)和E(K92R、R261Q),并且包括受体结合位点T131K和T135N/K,同时涉及5个抗原表位的有2株,涉及3个抗原表位的有4株(表 1)。
| 毒株名称 | 基因分支 | 抗原表位 | |||||||||||||||||||||||||
| A | B | C | D | E | |||||||||||||||||||||||
| 124 | 131a | 135a | 138 | 142 | 144 | 150 | 128 | 159 | 160 | 198 | 48 | 50 | 304 | 311 | 121 | 171 | 62 | 81 | 88 | 92 | 261 | ||||||
| A/Hong Kong/4801/2014 | 3C.2a | S | T | T | A | R | S | R | T | Y | T | S | I | E | A | H | N | N | E | N | V | K | R | ||||
| A/Switzerland/9715293/2013 | 3C.3a | S | T | T | S | G | N | R | A | S | K | S | I | E | A | Q | N | N | E | N | V | K | R | ||||
| A/JiangsuChongchuan/18/2016 | 3C.2a | S | I | T | A | R | S | R | T | Y | T | S | I | E | A | H | N | N | E | N | V | K | R | ||||
| A/ShanghaiHuangpu/1818/2017 | 3C.2a | S | K | T | A | K | R | R | T | Y | T | S | I | E | A | H | N | N | E | N | V | K | Q | ||||
| A/Zhejiangkecheng/1202/2015 | 3C.3a | - | - | - | S | G | N | - | A | S | K | - | - | - | - | Q | - | - | G | - | - | - | - | ||||
| A/Zhejiangkecheng/1218/2015 | 3C.3a | - | - | - | S | G | N | - | A | S | K | - | T | - | - | Q | - | - | - | - | - | - | - | ||||
| A/Zhejiangkecheng/1227/2015 | 3C.3a | - | - | - | S | G | N | - | A | S | K | - | - | - | - | Q | - | - | - | - | - | - | - | ||||
| A/Zhejiangkecheng/1572/2015 | 3C.3a | - | - | - | S | G | N | - | A | S | K | - | - | - | - | Q | - | - | - | - | - | - | - | ||||
| A/Zhejiangkecheng/1591/2015 | 3C.2a | - | K | - | - | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | ||||
| A/Zhejiangkecheng/1692/2015 | 3C.2a | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | ||||
| A/Zhejiangkecheng/1980/2016 | 3C.2a | - | K | - | - | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | ||||
| A/Zhejiangkecheng/1118/2017 | 3C.2a | - | K | - | - | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | ||||
| A/Zhejiangkecheng/1135/2017 | 3C.2a | - | K | - | - | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | ||||
| A/Zhejiangkecheng/1649/2017 | 3C.2a | - | K | - | - | K | - | - | - | - | - | - | - | K | - | - | - | - | - | - | - | - | Q | ||||
| A/Zhejiangkecheng/1680/2017 | 3C.2a | - | - | N | - | - | - | - | - | - | - | P | - | - | - | Q | K | K | - | - | - | R | - | ||||
| A/Zhejiangkecheng/1771/2017 | 3C.2a | - | - | N | - | - | - | - | - | - | - | P | - | - | - | Q | K | K | - | - | I | R | - | ||||
| A/Zhejiangkecheng/1790/2017 | 3C.2a | - | K | - | - | K | - | - | - | - | - | - | - | - | T | - | - | - | - | - | - | - | Q | ||||
| A/Zhejiangkecheng/1841/2017 | 3C.2a | R | - | K | - | G | K | K | - | - | - | - | - | - | - | - | K | - | - | - | - | - | Q | ||||
| A/Zhejiangkecheng/1854/2017 | 3C.2a | - | - | K | - | - | K | K | - | - | - | - | - | - | - | - | K | - | - | D | - | - | Q | ||||
| 注:氨基酸对比参照株A/Hong Kong/4801/2014;a表示受体结合位点 | |||||||||||||||||||||||||||
糖基化在病毒致病和抗免疫反应中有着重要作用[7];本研究中的分离株通过在线糖基化位点分析软件对HA蛋白氨基酸序列分析显示,18株分离株中共发现潜在糖基化位点15个,其中稳定存在的糖基化位点有9个,分别为8NST、22NGT、38NAT、45NSS、63NCT、165NVT、246NST、285NGS、483NGT/NET,8个位于HA1区,1个位于HA2区。2015年分离株与疫苗株A/Switzerland/9715293/2013对比,有2株分离株发生N144S变异,导致144NSS潜在糖基化位点消失,并且出现A128T和K160T变异,分别新增潜在糖基化位点126NWT和158NYT。2017年分离株与疫苗株A/Hong Kong/4801/2014对比,有4株分离株发生T135N/K变异,分别导致糖基化位点135NSS替代133NGT或133NGT消失,其中分离株A/Zhejiangkecheng/1841/2017同时出现S124R变异,导致糖基化位点122NES消失(表 2)。
| 毒株名称 | 糖基化位点 | |||||
| 122NES | 126NWT | 133NGT | 135NSS | 144NSS | 158NYT | |
| A/Zhejiangkecheng/1591/2015 | + | - | + | |||
| A/Zhejiangkecheng/1692/2015 | + | - | + | |||
| A/Zhejiangkecheng/1680/2017 | - | + | ||||
| A/Zhejiangkecheng/1771/2017 | - | + | ||||
| A/Zhejiangkecheng/1841/2017 | - | - | ||||
| A/Zhejiangkecheng/1854/2017 | - | |||||
| 注:糖基化位点参照同年度疫苗株;“+/-”表示增加(+)和缺失(-)潜在糖基化位点 | ||||||
H3N2亚型流感病毒由H2N2流感病毒和禽流感病毒H3重配变异而来,并于1968年在香港首先引起暴发流行[8];在过去的数十年内,H3N2亚型病毒经历着不断的变异进化,我国的流感疫情中,H3N2亚型流感病毒感染是主要型别之一[9-10];衢州市2015-2017年流感监测结果显示,在流感病例中,H3N2亚型流感病毒感染率居首位,其病毒分离株占总毒株数的51.33%,为流感流行的优势毒株,尤其在2017年夏季出现明显的流行高峰,与我国大部分南方省份流行情况一致[11],这提示我们需进一步加强H3N2亚型流感病毒的病原学监测,密切关注其变异进化情况。
H3N2亚型流感病毒HA基因分为多个基因型,目前全球流行的主要为3C系,而且3C系又可分为多个支系[12-13]。本研究通过对衢州市分离的18株H3N2亚型流感病毒HA基因分析发现,2015-2017年衢州市H3N2亚型流行株均为3C系,但支系从2015年的3C.3a逐渐转变为3C.2a支系,其中,2015年跨越2个支系,与同年度疫苗株A/Switzerland/9715293/2013的遗传距离逐渐加大,提示其疫苗免疫效果可能下降。同源性分析显示,2016年和2017年分离株与2016-2018年度疫苗株A/Hong Kong/4801/2014的核苷酸和氨基酸平均遗传距离呈现出扩大趋势,与系统进化树分析结果一致;说明WHO推荐的北半球疫苗株A/Hong Kong/4801/2014与当前流行株的匹配性可能降低,提示当前H3N2亚型流感病毒正逐渐变异进化。
甲型流感病毒的HA蛋白是其重要的表面抗原,也是变异频率最高的抗原之一,其HA1蛋白分子上最少含有5个抗原决定簇(A、B、C、D、E);抗原决定簇上氨基酸位点的变异,可能导致病毒抗原性和致病特征的改变[14-15]。本研究通过对分离株HA氨基酸序列与同年度疫苗株比较分析发现,涉及抗原决定簇上的位点变异共有22个,覆盖5个区域,其中A区变异率最高,有7个位点发生了变异,位点中变异频率最高的有T131K、R142G/S/K和S144N/K,均位于A区;上述位点的变异能够满足流感病毒抗原漂移的要求[16],提示病毒在抗原性上可能发生了较大变异,意味着其在一定范围内有可能出现更高强度的流行,这与我国部分南方省份的流感流行状况是一致的[11]。研究表明,流感病毒HA蛋白上受体结合位点的改变,能够影响病毒的增殖、免疫原性和宿主的特异性[17-18];本研究发现部分分离株在受体结合位点上发生了T131K和T135N/K变异,可能对病毒的机体免疫和进化有一定的影响,具体作用还有待进一步的研究。
HA蛋白上糖基化位点的改变,可能会导致病毒结构和抗原性发生变化,从而影响宿主的免疫系统反应,帮助病毒实现有效的免疫逃逸[19-20]。本研究中部分分离株有多个糖基化位点发生变异,包括144NSS、133NGT、122NES潜在糖基化位点的消失,以及126NWT、135NSS、158NYT糖基化位点的增加;其中135NSS和144NSS糖基化位点位于抗原决定簇A区,133NGT和135NSS糖基化位点位于受体结合部位,这些变异可能导致机体对感染病毒的清除障碍,促进病毒发生抗原性漂移,以及引发病毒与宿主细胞受体结合的改变。
综上所述,通过对衢州市2015-2017年H3N2亚型流感病毒HA的基因特征、抗原表位、受体结合位点和糖基化位点的分析研究,显示出流感病毒H3N2正处于不断变异进化之中,尤其在抗原位点上已呈现出较大变异,这意味着目前疫苗株A/Hong Kong/4801/2014对病毒的免疫效果可能下降,其防控策略有待进一步调整。
作者贡献:
黄世腾 ORCID:0000-0002-2789-9306
黄世腾:实验研究、数据分析及论文撰写
杨瑞军、吕磊:参与实验研究和数据分析
陈旭富、叶承华、万圣:参与数据分析和文章修改
| [1] |
Tong SX, Li Y, Rivailler P, et al. A distinct lineage of influenza A virus from bats[J]. Proc Natl Acad Sci USA, 2012, 109(11): 4269-4274. DOI:10.1073/pnas.1116200109 |
| [2] |
Centers for Disease Control and Prevention (CDC). Severe influenza among children and young adults with neurologic and neurodevelopmental conditions-Ohio, 2011[J]. Morb Mortal Wkly Rep, 2012, 60(51/52): 1729-1733. |
| [3] |
Maynard ND, Gutschow MV, Birch EW, et al. The virus as metabolic engineer[J]. Biotechnol J, 2010, 5(7): 686-694. DOI:10.1002/biot.201000080 |
| [4] |
Rewar S, Mirdha D, Rewar P. Treatment and prevention of pandemic H1N1 influenza[J]. Ann Glob Health, 2015, 81(5): 645-653. DOI:10.1016/j.aogh.2015.08.014 |
| [5] |
中华人民共和国卫生部. 卫生部办公厅关于印发《全国流感监测方案(2010年版)》的通知[EB/OL]. (2010-09-10)[2017-11-20]. http://www.nhfpc.gov.cn/jkj/s3577/201009/3fa356d0f4834d408fde6c12891a6482.shtml. National Health and Family Planning Commission of the People's Republic of China. The national influenza surveillance program (2010 Edition)[EB/OL]. (2010-09-10)[2017-11-20]. http://www.nhfpc.gov.cn/jkj/s3577/201009/3fa356d0f4834d408fde6c12891a6482.shtml. |
| [6] |
Lee HK, Tang JWT, Kong DHL, et al. Simplified large-scale Sanger genome sequencing for influenza A/H3N2 virus[J]. PLoS One, 2013, 8(5): e64785. DOI:10.1371/journal.pone.0064785 |
| [7] |
An YM, McCullers JA, Alymova I, et al. Glycosylation analysis of engineered H3N2 influenza A virus hemagglutinins with sequentially added historically relevant glycosylation sites[J]. J Proteome Res, 2015, 14(9): 3957-3969. DOI:10.1021/acs.jproteome.5b00416 |
| [8] |
WHO. Ten things you need to know about pandemic influenza (update of 14 October 2005)[J]. Wkly Epidemiol Rec, 2005, 80(49/50): 428-431. |
| [9] |
余靓, 张欣, 黄平, 等. 广东地区甲型H3N2流行性感冒病毒毒株血凝素基因变异与进化对抗原表位的影响[J]. 中华传染病杂志, 2016, 34(7): 404-409. Yu L, Zhang X, Huang P, et al. Effect of evolution and mutations of H3N2 virus hemagglutinin genes on the antigenic epitopes in Guangdong[J]. Chin J Infect Dis, 2016, 34(7): 404-409. DOI:10.3760/cma.j.issn.1000-6680.2016.07.004 |
| [10] |
黄维娟, 成艳辉, 李希姸, 等. 2014-2015监测年度中国H3N2亚型流感病毒病原学特征分析[J]. 中华微生物学和免疫学杂志, 2016, 36(1): 3-8. Huang WJ, Cheng YH, Li XY, et al. Virological characteristics of influenza A(H3N2)virus strains isolated in mainland China during 2014-2015 influenza surveillance year[J]. Chin J Microbiol Immunol, 2016, 36(1): 3-8. DOI:10.3760/cma.j.issn.0254-5101.2016.01.002 |
| [11] |
彭质斌, 陈涛, 康敏, 等. 2017年夏季全国流感疫情特征分析与风险评估[J]. 中国病毒病杂志, 2017, 7(4): 244-251. Peng ZB, Chen T, Kang M, et al. Epidemiology and risk assessment of seasonal influenza during the summer of 2017 in China[J]. Chin J Viral Dis, 2017, 7(4): 244-251. |
| [12] |
Sullivan SG, Chow MK, Barr IG, et al. Influenza viruses received and tested by the Melbourne WHO Collaborating Centre for Reference and Research on Influenza annual report, 2014[J]. Commun Dis Intell Q Rep, 2014, 39(4): E602-611. |
| [13] |
Stucker KM, Schobel SA, Olsen RJ, et al. Haemagglutinin mutations and glycosylation changes shaped the 2012/13 influenza A(H3N2) epidemic, Houston, Texas[J]. Euro Surveill, 2015, 20(18): 21122. |
| [14] |
余慧燕, 许可, 邓斐, 等. 2016-2017年江苏省人感染H7N9禽流感病毒HA和NA基因分子进化分析[J]. 病毒学报, 2017, 33(5): 685-690. Yu HY, Xu K, Deng F, et al. Molecular characteristics of the HA and NA genes of the human influenza A(H7N9)virus in Jiangsu Province, China, 2016-2017[J]. Chin J Virol, 2017, 33(5): 685-690. DOI:10.13242/j.cnki.bingduxuebao.003219 |
| [15] |
Li J, Zhou YY, Kou Y, et al. Interim estimates of divergence date and vaccine strain match of human influenza A(H3N2) virus from systematic influenza surveillance (2010-2015) in Hangzhou, Southeast of China[J]. Int J Infect Dis, 2015, 40: 17-24. DOI:10.1016/j.ijid.2015.09.013 |
| [16] |
Shih ACC, Hsiao TC, Ho MS, et al. Simultaneous amino acid substitutions at antigenic sites drive influenza A hemagglutinin evolution[J]. Proc Natl Acad Sci USA, 2007, 104(15): 6283-6288. DOI:10.1073/pnas.0701396104 |
| [17] |
Smith DJ, Lapedes AS, de Jong JC, et al. Mapping the antigenic and genetic evolution of influenza virus[J]. Science, 2004, 305(5682): 371-376. DOI:10.1126/science.1097211 |
| [18] |
Tang DJ, Lam YM, Siu YL, et al. A single residue substitution in the receptor-binding domain of H5N1 hemagglutinin is critical for packaging into pseudotyped lentiviral particles[J]. PLoS One, 2012, 7(11): e43596. DOI:10.1371/journal.pone.0043596 |
| [19] |
杨依丽, 吴军. 病毒抗原糖基化与免疫关系的研究进展[J]. 生物技术通讯, 2008, 19(5): 728-730. Yang YL, Wu J. Advances in the relationship between immune and glycosylation of viral antigen[J]. Lett Biotechnol, 2008, 19(5): 728-730. DOI:10.3969/j.issn.1009-0002.2008.05.026 |
| [20] |
Sun SS, Wang QZ, Zhao F, et al. Prediction of biological functions on glycosylation site migrations in human influenza H1N1 viruses[J]. PLoS One, 2012, 7(2): e32119. DOI:10.1371/journal.pone.0032119 |
 2018, Vol. 33
2018, Vol. 33


