扩展功能
文章信息
- 张军鸽, 赵飞, 刘立雍, 马闪珊, 单小云
- Zhang Junge, Zhao Fei, Liu Liyong, Ma Shanshan, Shan Xiaoyun
- 多种一步法快速提取DNA对肺炎支原体PCR检测的影响
- Influence of different rapid one-step DNA extraction on PCR detection of Mycoplasma pneumoniae
- 疾病监测, 2018, 33(6): 503-509
- Disease Surveillance, 2018, 33(6): 503-509
- 10.3784/j.issn.1003-9961.2018.06.014
-
文章历史
- 收稿日期:2018-01-03
2. 中国疾病预防控制中心传染病预防控制所, 北京 102206;
3. 浙江省金华市中心医院检验科, 浙江 金华 321000
2. National Institutes for Communicable Disease Prevention and Control, Chinese Center for Disease Control and Prevention, Beijing 102206, China;
3. Department of Clinical Laboratory, Jinhua County Central Hospital, Jinhua 321000, Zhejiang, China
DNA是遗传信息的载体,是最重要的生物信息分子。随着分子生物学技术的高速发展,以DNA杂交、扩增和序列分析为代表的分子诊断和检测技术在各领域中发挥重要作用。而现代分子生物学检测技术首先面临如何从复杂的样本中迅速有效地分离和提取DNA,且保证其质量可满足后续实验要求。1869年Friedrich Miescher首次成功提取DNA,较多研究者对其提取方法进行探索,对提取材料和试剂进行改进[1],有多种经典的提取类别和技术,以满足不同类型实验。目前,常用DNA提取方法包括碱裂解法[2]、酚氯仿抽提法[3]、离心过柱[4]、磁珠法[5]和煮沸法[6]等。各方法适用于不同类型样品,其提取DNA浓度、纯度和繁琐程度也各不相同。各种方法的第1步为破碎细胞,释放DNA;第2步为将释放的DNA与其他生化组分(如蛋白质、多糖、脂类等)分离;第3步为针对性的富集DNA。
随着DNA检测技术的推广,研究者常面对大批量样品的DNA提取,需更加快捷和简便的提取技术以满足需求,同时将污染风险降至最低。依赖于磁珠或柱膜提取DNA的试剂盒效果较好,但其步骤繁琐、批量提取时污染风险显著增加的特点难以满足大量标本的提取需求。因此,研究者在保证DNA提取质量的前提下尽量缩减核DNA取步骤,1992年de Lamballerie等[7]报道Chelex-100一步法,1995年Steiner等[8]报道ROSE法(Rapid one-step Extraction)提取特定样品中的DNA。上述两种一步法DNA提取方法均在传统的水煮法[9]基础上进行改进,通过添加裂解剂和吸附剂,简化了DNA与其他生化组分分离的步骤,确保提取DNA的质量,广泛应用于大量标本的DNA提取,减少了工作量。两种一步DNA提取法中使用了SDS和(或)EDTA等酶抑制剂,主要为确保部分难裂解标本,如植物和组织标本的裂解[10]和防止DNA降解[11]。提取的DNA往往需稀释后进行后续PCR扩增,否则高浓度的抑制物影响后续扩增。因此,在临床标本检测中很少使用传统一步法提取DNA,尤其是对于低拷贝病原体的检测。
肺炎支原体(Mycoplasma pneumoniae,MP)是临床常见呼吸道感染致病菌[12],因其分离培养困难,DNA检测技术日趋成熟,逐渐成为肺炎支原体检测的金标准[13],其检测需求日益增大。目前,市面上提取试剂盒主要以柱膜法和磁珠法居多,提取步骤繁琐。而肺炎支原体标本以咽拭子为主,相对于其他人体组织标本,更易于处理和提取,理论上非常适用于一步法DNA提取。但一步法提取的肺炎支原体DNA量、对后续PCR扩增的影响、改良后的效果及用于肺炎支原体低拷贝临床标本的应用等尚未有文献报道。本研究通过对比6种方法提取的肺炎支原体纯菌及其阳性临床咽拭子标本DNA进行PCR检测,评价不同方法提取的DNA对PCR扩增的影响,进一步明确一步DNA提取法在肺炎支原体扩增检测中的效果及应用范围。
1 材料与方法 1.1 菌株来源肺炎支原体菌株ATCC29342由中国疾病预防控制中心传染病预防控制所购买并保藏,30份临床咽拭子标本(经实验室培养验证为肺炎支原体阳性的标本)来源于北京市朝阳区三间房社区卫生服务中心。
1.2 试剂和仪器QIAamp DNA Mini Kit为QIAGEN公司产品;Platinum Taq DNA polymerase和Platinum quantitative PCR SuperMix-UDG购自美国Invitrogen公司;Nucleotide mix和Nuclease-free water购自美国Promega公司;Ex Taq Kit购自日本TaKaRa公司;EDTA、SDS、NP-40、Tween-20、Chelex- 100、PVPP购自索莱宝生物有限公司,荧光PCR仪C1000 Thermal Cycler和凝胶自动成像系统为美国BIORAD公司产品。
1.3 DNA提取将菌株液体培养至对数生长期,分别取200 μl培养液使用6种方法提取DNA(表 1),30份临床咽拭子标本各取200 μl使用6种方法提取,提取方法及步骤见表 1。
| 方法 | 提取液配方 | 提取步骤 |
| ROSE法 | 10mmol/L pH8.0 Tris-HCl 300 mmol/L pH8.0 EDTA 1%SDS 2% PVPP |
标本12 000×g 4 ℃离心5 min, 弃上清液; 沉淀加人200 μl提取液,95 ℃加热15 min; 12000×g,4 ℃离心5 min, 上清液备用 |
| 改良ROSE法 | 10 mmol/L pH8.0 Tris-HCl 300 mmol/L pH8.0 EDTA 2%PVPP |
同ROSE法 |
| Chelex-100法 | 1% Tween-20 1% NP-40 0.1% SDS 10% Chelex-100 |
同ROSE法 |
| 改良Chelex-100法 | 1% Tween-20 1% NP-40 10% Chelex-100 |
同ROSE法 |
| QIAGEN试剂盒法 | QIAamp DNA Mini Kit (51306) | 按操作说明提取,最终使用200 μl洗脱液洗脱 |
| 水煮法 | 纯水 | 同ROSE法 |
6种方法提取的菌株DNA分别使用无核酸水分别稀释10、100和1 000倍,作为浓度梯度模板使用。
1.5 荧光PCR检测使用文献报道的荧光PCR进行肺炎支原体检测[14]。反应体系:0.25 μl Platinum Taq DNA polymerase(C10966 -034),10 μl Platinum quantitative PCR SuperMix - UDG(C11730 - 025),0.8 μl MgCl2(50 mmol / L),0.4 μl正向引物Mp - F(25 μmol/L),0.4 μl反向引物Mp-R(25 μmol/L),0.2 μl探针Mp-P(25 μmol/L),1 μl PCR nucleotide mix(C1141),2 μl DNA模板,用Nuclease-free water(P1193)补至20 μl。体系混合后使用伯乐C1000 Thermal Cycler荧光PCR仪进行扩增检测。扩增条件:95 ℃ 2 min;95 ℃ 15 s,55 ℃ 15 s,共45个循环。
1.6 普通PCR检测使用文献报道的普通PCR肺炎支原体进行目的基因扩增[15]。反应体系:5 × PrimeSTAR Buffer,4 μl;dNTP Mixture,1.6 μl;引物各0.4 μl;PrimeSTAR HS DNA polymerase,0.2 μl;模板DNA,2 μl;用Nuclease-free water补至20 μl。反应条件:98 ℃,预变性2 min;98 ℃,15 s,58 ℃,15 s,72 ℃,1 min,30个循环,72 ℃延伸5 min。PCR产物经1.8%琼脂糖凝胶电泳。
2 结果 2.1 PCR检测结果ROSE法、改良ROSE法和Chelex-100法提取的核酸原液无扩增,其中ROSE法、改良ROSE法提取的DNA经≥100倍稀释、Chelex-100法提取DNA经≥10倍稀释后、改良Chelex-100法、QIAGEN试剂盒法和水煮法所提DNA均为阳性。改良Chelex-100法提取的DNA在6种方法中的荧光检测循环阈值(Ct值)在不同稀释浓度模板中均为最小值,见表 2。
| 方法 | 荧光PCR | 普通PCR | |||||||
| DNA原液 | 稀释10倍 | 稀释100倍 | 稀释1 000倍 | DNA原液 | 稀释10倍 | 稀释100倍 | 稀释1 000倍 | ||
| ROSE法 | N | N | 18.62 | 21.97 | N | P | P | P | |
| 改良ROSE法 | N | N | 21.45 | 24.74 | N | P | P | P | |
| Chelex-100法 | N | 13.52 | 17.07 | 20.51 | P | P | P | P | |
| 改良Chelex-100法 | 10.37 | 13.76 | 16.93 | 20.14 | P | P | P | P | |
| QIAGEN试剂盒法 | 12.62 | 16.01 | 19.31 | 22.97 | P | P | P | P | |
| 水煮法 | 11.11 | 14.46 | 17.91 | 21.37 | P | P | P | P | |
| 注:N代表阴性结果,P代表阳性结果,数值代表荧光PCR阳性Ct值 | |||||||||
改良Chelex-100法、QIAGEN试剂盒法和水煮法荧光提取的DNA经PCR检测,对结果进行对数曲线分析发现(图 1),3种方法的标准曲线均R2=1.000;斜率分别为-3.245、-3.437和-3.422;扩增效率分别为103.3%、95.4%和96.0%;原始浓度下扩增荧光信号值RFU分别为10 500、13 500和12 500。
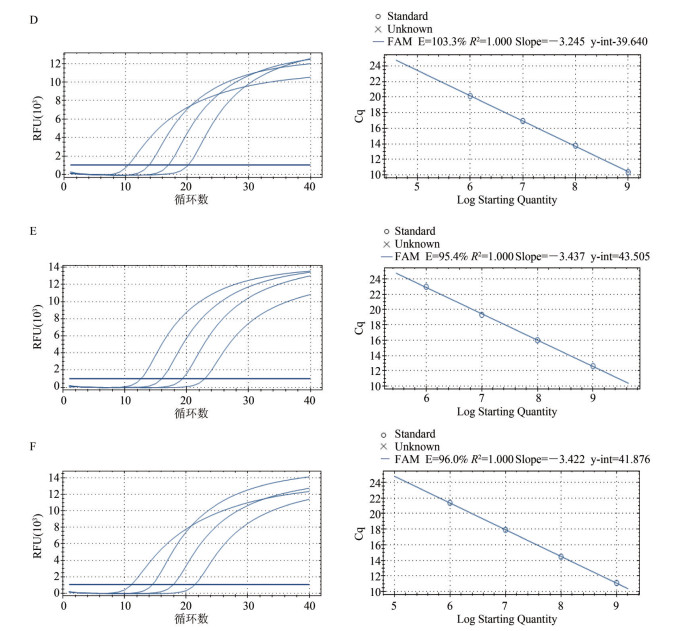
|
| 图 1 3种方法对4种浓度核酸荧光PCR检测线性关系 Figure 1 Three methods for four different concentrations of nucleic acid PCR assay to linear relation |
| |
普通PCR扩增只有ROSE法和改良ROSE法的原始浓度模板无扩增条带,其余均为阳性。与荧光PCR结果比较,Chelex-100法原液(图 2孔12)、ROSE法稀释10倍(图 2孔3)和改良ROSE法稀释10倍(图 2孔7)模板在普通PCR检测中为阳性,但荧光PCR检测均为阴性,见图 2。普通PCR产物的量与初始模板浓度呈正相关。
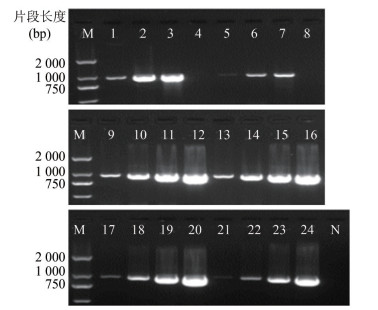
|
| 图 2 6种方法提取菌株稀释模板PCR结果 Figure 2 PCR results of dilution template by using 6 methods for extraction of strain 注:M.Marker;N.阴性对照;1~4.ROSE法;5~8.改良ROSE法;9~12. Chelex-100法;13~16.改良Chelex-100法;17~20. QIAGEN试剂盒法;21~24.水煮法;每组自左向右分别为原液稀释1 000、100、10倍和原液 |
| |
30份临床标本经荧光PCR检测,ROSE法、改良ROSE法和Chelex-100法所提DNA均为阴性,QIAGEN试剂盒法所提核酸均为阳性,改良Chelex- 100法、水煮法所提DNA分别有24和13份为阳性。经χ2检验,2种检测方法提取DNA阳性差异有统计学意义(χ2=9.090,P < 0.01);经普通PCR检测,4份经Chelex-100法、改良Chelex-100法和QIAGEN试剂盒法提取的DNA经PCR检测为阳性,经水煮法提取的DNA阳性2份,其余26份均为阴性(表 3)。改良Chelex-100法和水煮法提取DNA经荧光PCR检测,Ct值较QIAGEN试剂盒法提取样本分别后移1.95和2.38。
| 标本编号 | 阳性标本 | 荧光PCR(Ct值) | 普通PCR | |||||||||||
| ROSE法 | 改ROSE法 | Chelex-100法 | 改良Chelex-100法 | QIAGEN试剂盒法 | 水煮法 | ROSE法 | 改良ROSE法 | Chelex-100法 | 改良Chelex-100法 | QIAGEN试剂盒法 | 水煮法 | |||
| 1 | P | N | N | N | 37.17±0.12 | 35.66±0.21 | N | N | N | N | N | N | N | |
| 2 | P | N | N | N | 33.44±0.16 | 33.31±0.14 | 33.51±0.02 | N | N | P | P | P | P | |
| 3 | P | N | N | N | 34.88±0.13 | 34.81±0.23 | 35.24±0.12 | N | N | N | N | N | N | |
| 4 | P | N | N | N | 36.81±0.20 | 34.14±0.11 | 38.06±0.33 | N | N | N | N | N | N | |
| 5 | P | N | N | N | 37.12±0.25 | 36.00±0.12 | N | N | N | N | N | N | N | |
| 6 | P | N | N | N | 37.44±0.32 | 35.09±0.22 | 38.19±0.37 | N | N | N | N | N | N | |
| 7 | P | N | N | N | 36.39±0.23 | 31.42±0.16 | N | N | N | P | P | P | N | |
| 8 | P | N | N | N | 36.45±0.44 | 33.61±0.04 | 38.23±0.47 | N | N | N | N | N | N | |
| 9 | P | N | N | N | 36.12±0.05 | 34.78±0.25 | N | N | N | N | N | N | N | |
| 10 | P | N | N | N | 36.98±0.13 | 34.01±0.19 | N | N | N | N | N | N | N | |
| 11 | P | N | N | N | 34.87±0.29 | 33.82±0.14 | N | N | N | N | N | N | N | |
| 12 | P | N | N | N | 36.55±0.31 | 33.59±0.05 | N | N | N | P | P | P | N | |
| 13 | P | N | N | N | 36.47±0.11 | 34.55±0.07 | N | N | N | N | N | N | N | |
| 14 | P | N | N | N | 37.05±0.28 | 33.59±0.10 | N | N | N | N | N | N | N | |
| 15 | P | N | N | N | N | 37.79±0.08 | N | N | N | N | N | N | N | |
| 16 | P | N | N | N | N | 36.92±0.30 | N | N | N | N | N | N | N | |
| 17 | P | N | N | N | 40.11±0.41 | 37.12±0.23 | 40.02±0.29 | N | N | N | N | N | N | |
| 18 | P | N | N | N | N | 37.55±0.19 | N | N | N | N | N | N | N | |
| 19 | P | N | N | N | N | 39.94±0.44 | N | N | N | N | N | N | N | |
| 20 | P | N | N | N | N | 39.95±0.26 | N | N | N | N | N | N | N | |
| 21 | P | N | N | N | 36.83±0.09 | 36.44±0.19 | 39.46±0.51 | N | N | N | N | N | N | |
| 22 | P | N | N | N | 38.75±0.18 | 36.30±0.21 | N | N | N | N | N | N | N | |
| 23 | P | N | N | N | 36.99±0.14 | 37.91±0.19 | 37.22±0.23 | N | N | N | N | N | N | |
| 24 | P | N | N | N | 39.11±0.37 | 36.34±0.33 | 39.69±0.30 | N | N | N | N | N | N | |
| 25 | P | N | N | N | 37.61±0.25 | 36.69±0.24 | N | N | N | N | N | N | N | |
| 26 | P | N | N | N | 38.88±0.11 | 35.29±0.10 | 39.08±0.32 | N | N | N | N | N | N | |
| 27 | P | N | N | N | 38.44±0.27 | 37.31±0.20 | 39.17±0.21 | N | N | N | N | N | N | |
| 28 | P | N | N | N | 34.28±0.22 | 34.12±0.02 | 34.59±0.38 | N | N | P | P | P | P | |
| 29 | P | N | N | N | 39.07±0.39 | 35.01±0.19 | 38.99±0.42 | N | N | N | N | N | N | |
| 30 | P | N | N | N | N | 38.04±0.22 | N | N | N | N | N | N | N | |
ROSE法和Chelex-100法最初用于提取较大数量级DNA的组织等标本,而组织难以处理,必须加入SDS作为裂解剂和(或)EDTA作为核酸保护剂。而该2种物质均为后续PCR扩增Taq酶的强力抑制剂,因此,2种方法提取的DNA均应进行一定稀释后可作为PCR模板进行扩增。咽拭子标本主要为由人体上皮细胞、定植的微生物和少量黏液组成,成分较为简单且易于处理,且肺炎支原体缺乏细胞壁[16-17]。热处理易于核酸释放,故本研究去除2种经典一步法中的SDS,优化咽拭子的提取方法,完全依赖于加热使肺炎支原体菌体破坏释放核酸。6种方法对肺炎支原体纯培养物提取DNA及其梯度稀释物进行普通PCR和荧光PCR,通过比较经典的ROSE法和Chelex-100法可知,体系中SDS浓度为0.2%时,2种PCR均不能扩增,体系中SDS浓度为0.02%时,仅PCR可以进行扩增,SDS浓度为0.002%时,荧光PCR方可扩增,说明SDS对于PCR的Taq酶有显著抑制作用,该抑制作用对于荧光PCR更为显著。分析ROSE法发现,体系中EDTA浓度为60 mmol/L时,2种PCR均不能扩增,体系中EDTA浓度为6 mmol/L时,仅PCR可以进行扩增,EDTA浓度为0.6 mmol/L时,荧光PCR方可扩增,说明EDTA对于PCR的Taq酶有显著抑制作用,且抑制作用对于荧光PCR更为显著。因此,经典的一步法提取的DNA并不适合直接作为模板进行PCR扩增,必须将提取物中的抑制剂稀释到不足以抑制Taq酶活性的程度,否则即使模板中含有高浓度的待扩增模板也无法进行有效扩增。改良Chelex-100法、QIAGEN试剂盒提取法和水煮法提取的肺炎支原体纯菌核酸均可有效扩增,在4种浓度梯度下3种方法扩增均未受到抑制物显著影响,仅对改良Chelex-100法和水煮法初始浓度的荧光PCR扩增信号值略有影响,稀释后其荧光信号值反而升高,说明改良Chelex-100法和水煮法提取的肺炎支原体纯菌DNA中的抑制物对于扩增的影响非常小,仅对于荧光PCR信号强度有微弱影响。分析3种方法的Ct值不难发现,改良Chelex-100法和水煮法提取的DNA量较QIAGEN试剂盒多,可能因QIAGEN试剂盒为基于柱膜法提取,在提取大量DNA时,过载导致的DNA吸附不全及单次洗脱不充分均会造成部分目的DNA丢失,影响总获得率。在该种条件下,改良Chelex-100法和水煮法提取的DNA损失量非常小,且提取的肺炎支原体纯菌DNA中抑制物来源于菌体自身,故抑制作用有限,适用于大批量肺炎支原体纯菌DNA的快速提取。
肺炎支原体临床咽拭子标本较纯菌含有人体上皮细胞、黏液成分和未知的外源性物质,存在抑制物的可能性大大增加,且标本中肺炎支原体的含量非常少。因此,肺炎支原体临床标本一般采用试剂盒提取,一方面可对标本中的肺炎支原体进行浓缩,另一方面可有效去除复杂样本中的多种干扰物质。30份肺炎支原体阳性咽拭子标本经6种方法提取DNA后进行PCR扩增,因外源性抑制物SDS和EDTA的存在,ROSE法、改良ROSE法和Chelex-100法提取的DNA均无法对肺炎支原体进行有效扩增。改良Chelex-100法、QIAGEN试剂盒法和水煮法提取,只有2、3、23和28号标本的Ct值接近,其余26份肺炎支原体阳性标本 Ct值差异较大,表明2、3、23和28号标本的DNA扩增抑制物较少,提取的DNA不经分离纯化也可有效扩增,与肺炎支原体纯菌PCR扩增结果类似。但其他26份阳性标本中均存在不同程度的DNA扩增抑制物,造成水煮法的Ct值显著后移甚至出现假阴性结果。由此可见,大多数咽拭子标本中的抑制物足以影响PCR体系中的Taq酶活性,未经去除干扰物处理的DNA提取方式不适合临床标本中肺炎支原体的PCR检测。Chelex-100法因Chelex-100吸附部分标本中携带的DNA扩增抑制物,使其检测结果与QIAGEN试剂盒法接近,但残余抑制物使该方法的 Ct值发生不同程度后移,无法正确体现标本中肺炎支原体的DNA量,其中6份Ct值偏大的极低拷贝标本(Ct 值>36)出现假阴性结果。因此,该方法提取的DNA不适合标本中肺炎支原体的定量检测,仅作为定性检测的DNA提取方式。普通PCR仅有4份阳性标本可以检测,Ct值约31~ 34。Ct值对应的肺炎支原体拷贝数为102~103,接近普通PCR技术的检测限[18]。因此,普通PCR不能有效检测其余拷贝数更少的标本,为方法自身灵敏度所限。2、7、12和28号肺炎支原体阳性标本经Chelex-100法提取的DNA可以进行普通PCR扩增但荧光PCR不能扩增,进一步验证SDS对荧光PCR的影响更大。
综上所述,通过多种DNA提取方法对肺炎支原体纯菌和临床标本中处理后的2种PCR扩增检测结果比较,传统一步法提取的DNA不能直接作为PCR扩增的模板使用,需经过稀释后方可作为模板扩增;普通PCR对于SDS和EDTA的容忍度高于荧光PCR;改良Chelex-100法在提取肺炎支原体纯菌DNA用于PCR检测时具有显著优势,DNA产量最高且操作简便。但对于临床标本中肺炎支原体DNA提取仍以QIAGEN试剂盒法优势最明显。改良Chelex-100法因无法完全去除干扰物质,不能作为定量检测提取方法,仅限于标本中肺炎支原体的定性检测,存在部分肺炎支原体低拷贝标本检测假阴性结果。
作者贡献:
张军鸽 ORCID:0000-0001-8911-9742
张军鸽:主要完成实验设计、实施及文章写作
刘立雍:菌株培养及核酸提取,并协助完成部分分子生物学实验
马闪珊:协助数据分析与论文修改赵飞、单小云:实验设计及技术指导
| [1] |
Dahm R. Friedrich miescher and the discovery of DNA[J]. Dev Biol, 2005, 278(2): 274-288. DOI:10.1016/j.ydbio.2004.11.028 |
| [2] |
Feliciello I, Chinali G. A modified alkaline lysis method for the preparation of highly purified plasmid DNA from Escherichia coli[J]. Anal Biochem, 1993, 212(2): 394-401. DOI:10.1006/abio.1993.1346 |
| [3] |
Sambrook J, Fritsch EF, Maniatis T. Molecular cloning:a laboratory manual[M]. 2th. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press, 1989.
|
| [4] |
Lee HY, Park MJ, Kim NY, et al. Simple and highly effective DNA extraction methods from old skeletal remains using silica columns[J]. Forensic Sci Int Genet, 2010, 4(5): 275-280. DOI:10.1016/j.fsigen.2009.10.014 |
| [5] |
Flagstad Ø, Røed K, Stacy JE, et al. Reliable noninvasive genotyping based on excremental PCR of nuclear DNA purified with a magnetic bead protocol[J]. Mol Ecol, 1999, 8(5): 879-883. DOI:10.1046/j.1365-294X.1999.00623.x |
| [6] |
Sepp R, Szabó I, Uda H, et al. Rapid techniques for DNA extraction from routinely processed archival tissue for use in PCR[J]. J Clin Pathol, 1994, 47(4): 318-323. DOI:10.1136/jcp.47.4.318 |
| [7] |
de Lamballerie X, Zandotti C, Vignoli C, et al. A one-step microbial DNA extraction method using"Chelex 100"suitable for gene amplification[J]. Res Microbiol, 1992, 143(8): 785-790. DOI:10.1016/0923-2508(92)90107-Y |
| [8] |
Steiner JJ, Poklemba CJ, Fjellstrom RG, et al. A rapid one-tube genomic DNA extraction process for PCR and RAPD analyses[J]. Nucleic Acids Res, 1995, 23(13): 2569-2570. |
| [9] |
Lench N, Stanier P, Williamson R. Simple non-invasive method to obtain DNA for gene analysis[J]. Lancet, 1988, 331(8599): 1356-1358. DOI:10.1016/S0140-6736(88)92178-2 |
| [10] |
Goldenberger D, Perschil I, Ritzler M, et al. A simple "universal" DNA extraction procedure using SDS and proteinase K is compatible with direct PCR amplification[J]. Genome Res, 1995, 4(6): 368-370. DOI:10.1101/gr.4.6.368 |
| [11] |
Lahiri DK, Schnabel B. DNA isolation by a rapid method from human blood samples:effects of MgCl2, EDTA, storage time, and temperature on DNA yield and quality[J]. Biochem Genet, 1993, 31(7/8): 321-328. DOI:10.1007/BF00553174 |
| [12] |
Waites KB, Talkington DF. Mycoplasma pneumoniae and its role as a human pathogen[J]. Clin Microbiol Rev, 2004, 17(4): 697-728. DOI:10.1128/CMR.17.4.697-728.2004 |
| [13] |
Dorigo-Zetsma JW, Zaat SA, Wertheim-Van Dillen PM, et al. Comparison of PCR, culture, and serological tests for diagnosis of Mycoplasma pneumoniae respiratory tract infection in children[J]. J Clin Microbiol, 1999, 37(1): 14-17. |
| [14] |
Zhao F, Cao B, He LH, et al. Evaluation of a new real-time PCR assay for detection of Mycoplasma pneumoniae in clinical specimens[J]. Biomed Environ Sci, 2012, 25(1): 77-81. DOI:10.3967/0895-3988.2012.01.011 |
| [15] |
Matsuoka M, Narita M, Okazaki N, et al. Characterization and molecular analysis of macrolide-resistant Mycoplasma pneumoniae clinical isolates obtained in Japan[J]. Antimicrob Agents Chemother, 2004, 48(12): 4624-4630. DOI:10.1128/AAC.48.12.4624-4630.2004 |
| [16] |
Kishaba T. Community-acquired pneumonia caused by Mycoplasma pneumoniae: how physical and radiological examination contribute to successful diagnosis[J]. Front Med (Lausanne), 2016, 3: 28. DOI:10.3389/fmed.2016.00028 |
| [17] |
Waites KB, Xiao L, Liu Y, et al. Mycoplasma pneumoniae from the respiratory tract and beyond[J]. Clin Microbiol Rev, 2017, 30(3): 747-809. DOI:10.1128/CMR.00114-16 |
| [18] |
Zhao F, Liu Z, Gu YX, et al. Detection of Mycoplasma pneumoniae by colorimetric loop-mediated isothermal amplification[J]. Acta Microbiol Immunol Hung, 2013, 60(1): 1-9. DOI:10.1556/AMicr.60.2013.1.1 |
 2018, Vol. 33
2018, Vol. 33


