扩展功能
文章信息
- 杨元斌, 高红, 章丹阳, 闫鹏, 沈玄艺, 宋启发
- Yang Yuanbin, Gao Hong, Zhang Danyang, Yan Peng, Shen Xuanyi, Song Qifa
- 浙江省宁波市多重耐药肯塔基沙门菌的检出及病原学分析
- Etiology of multi-drug resistant Salmonella Kentucky isolates from retail chicken and a patient in Ningbo
- 疾病监测, 2018, 33(6): 510-514
- Disease Surveillance, 2018, 33(6): 510-514
- 10.3784/j.issn.1003-9961.2018.06.015
-
文章历史
- 收稿日期:2018-01-02
非伤寒沙门菌(nontyphoidal Salmonella,NTS) 能够引起肠胃炎(食物中毒)、菌血症以及继发的病灶感染等,是威胁全球的公共卫生问题[1]。据报道,我国细菌性肠胃炎的常见致病菌中,NTS仅次于副溶血弧菌,位居致病谱的第2位[2-3]。
大多数NTS的感染归因于食用了受污染的动物性食品,引发带有腹部绞痛的肠胃炎、高热及腹泻等胃肠炎症状,虽然大多预后良好,但是对于年老体弱者或者免疫力低下者,仍会有致命的危险。禽类食品是该类感染的重要载体[4-5];氟喹诺酮类和三代头孢类抗生素是目前治疗沙门菌感染的首选用药。由于上述两类药物在禽类养殖业中也被广泛应用,因此养鸡场和市售鸡肉已成为含超广谱β-内酰胺酶(extended spectrum β-lactamases,ESBLs)和喹诺酮耐药沙门菌的重要储存库[6]。
肯塔基血清型沙门菌,最早鉴定于1937年,分离自养殖鸡体,考夫曼抗原式为8,20 i:z6,该菌被公认为与禽类相关[7]。肯塔基沙门菌流行于北非、欧洲、北美等地区,对环丙沙星及三代头孢菌素等抗生素呈现多重、高水平耐药,因此亦被欧美地区称为“超级耐药”肯塔基沙门菌[8-10]。有流行病学证据证实“超级耐药”肯塔基沙门菌系通过旅游者从北非传播到欧洲和北美地区[11],因而欧洲和北美地区的食品及农业管理部门对其检出及传播高度关注,是上述地区非伤寒沙门菌重点监测的血清型之一。
我国肯塔基沙门菌属于罕见血清型,分离率很低,该血清型的多重耐药株无论在食品还是患者中均未见国内文献报道。本研究在宁波市2016年市售鸡肉样品以及2017年住院腹泻患者粪便中均检出“超级耐药”肯塔基沙门菌,现将该菌的病原学分析如下。
1 材料与方法 1.1 材料 1.1.1 菌株来源肯塔基沙门菌共2株,分别来自2016年食源性疾病监测项目中的市售鸡肉样品以及2017年住院腹泻患者的粪便标本。
1.1.2 主要仪器与试剂全自动细菌鉴定仪Vitek 2 Compact(生物梅里埃,法国);脉冲场凝胶电泳仪CHEF Mapper XA和凝胶成像仪Gel Doc XR+(BioRad,美国);蛋白酶K、限制性内切酶Xba Ⅰ、Bln Ⅰ(Promega,美国);低熔点琼脂糖Seakem Gold Agar(LONZA,美国);沙门菌显色平板(CHROMagar®,法国);沙门菌诊断血清(SSI,丹麦);Thermo Fisher Vizion药物敏感性分析仪(Thermo Fisher,美国);梯度PCR仪(Eppendorf,德国);Ex taq DNA聚合酶(TaKaRa,大连)。
1.2 方法 1.2.1 细菌的鉴定及血清学分型细菌分离及生化鉴定按照GB 4789.4-2016《食品微生物学检验沙门氏菌检验》及WS/T 13-1996《沙门氏菌食物中毒诊断标准及处理原则》操作;血清学分型依据GB 4789.4-2010《食品微生物学检验沙门氏菌检验》和国际通用的White Kaufmann Le MinorScheme WKLM表解[12]。生化鉴定为沙门菌者,且血清凝集结果为8,20,i:z6判断为肯塔基沙门菌。
1.2.2 抗生素敏感试验参照2017年临床实验室标准研究所(CLSI)的操作规程,采用微量肉汤稀释法对12种抗生素进行药物敏感试验,Thermo Fisher Vizion药物敏感性分析仪确定最低抑菌浓度(minimum inhibitory concentration,MIC)值。参考M100-S27抗生素敏感性试验的判断标准进行折点判断[13],试验结果依此判断敏感(S)、中介(I)和耐药(R)。根据欧洲抗生素敏感性测定委员会(The Uropean Committee on Antimicrobial Susceptibility Testing,EUCAST)以及CLSI的指导标准,将环丙沙星对细菌的MIC值≥4 μg/ml判断为环丙沙星高水平耐药菌株[14]。质控菌株为大肠埃希菌ATCC25922。
1.2.3 ESBLs表型确证试验参照CLSI推荐的纸片确证扩散法标准进行,采用头孢噻肟、头孢噻肟/克拉维酸检测,对头孢噻肟加克拉维酸的抑菌圈直径与不加克拉维酸的抑菌圈直径进行比较,若差值≥5 mm,确证该菌株产ESBLs。
1.2.4 分子分型参照PulseNet China沙门菌脉冲场凝胶电泳(pulsed field gel electrophoresis,PFGE)实验步骤[15],选择XbaⅠ和Bln Ⅰ内切酶对2株肯塔基沙门菌进行酶切分型。标准分子质量菌株为布伦登卢普沙门菌(H9812)[16]。电泳图谱使用Bionumerics 6.6软件进行聚类分析,以H9812作为相对分子质量标准。每条带型间的相似度用Dice系数来衡量,条带位置差异容许度选择1.5%,优化值选择1.5%。用非加权组平均法(unweighted pairgroup method with arithmetic means,UPGMA)进行分析,最后导出聚类图。参考Tenover原则:酶切后图谱完全一致定为同一带型;酶切后图谱有1~3条带不同者,可认为菌株间关系较密切;酶切后图谱如有3条以上条带不同者则认为菌株间关系不密切[17]。
1.2.5 耐药基因检测 1.2.5.1 耐药基因的扩增煮沸法制备细菌的DNA模板。分别参考GenBank中的DNA旋转酶A亚单位基因(gyrA)、ESBLs耐药基因blaCTX-M14-like、blaCTX-M15-like、blaOXA、blaTEM耐药基因的序列,设计相应引物见表 1。PCR反应条件:94 ℃ 5 min,94 ℃ 45 s,50 ℃ 45 s,72 ℃ 90 s,30个循环;72 ℃ 5 min,4 ℃保存。PCR扩增产物经1%的琼脂糖凝胶电泳,用凝胶成像系统观察结果。gyrA基因扩增PCR产物用核酸回收试剂盒回收后进行测序分析。
| 基因名称 | 引物序列 | 片段大小(bp) |
| gyrA | 正向:CGT TGG TGA CGT AAT CGG TA 反向:CCG TAC CGT CAT AGT TAT CC |
251 |
| blaCTX-M14-like | 正向:AAA ACT TGC CGA ATT AGA GC 反向:TTA GGT TGA GGC TGG GTG AA |
711 |
| blaCTX-M15-like | 正向:ATG AAC GCTTTC CAATGT GC 反向:GGT CGT ATT GCC TTT GAG CC |
461 |
| blaTEM | 正向:TGT CGC CCTTATTCC CTTTT 反向:ATA GTT GCC TGA CTC CCC GT |
783 |
| blaOXA | 正向:AAT GGC ACC AGA TTC AAC TT 反向TGG CTT TTA TGC TTG ATG TT |
593 |
PCR扩增产物采用一代测序技术,由上海生工生物工程股份有限公司进行正反向测序,用DNAman软件对测序结果进行各序列间的比较,提交序列资料至美国国立生物技术信息中心(www.ncbi.nlm.nih.gov/nucleotide),利用Blast功能进行基因序列比对。
2 结果 2.1 肯塔基沙门菌分子分型和聚类2株肯塔基沙门菌基因组经Xba Ⅰ酶切,均获得12个片段,见图 1a;2株肯塔基沙门菌基因组经Bln Ⅰ酶切,均获得9个片段,见图 1b;< 20.5 kb片段不参与分析。经BioNumerics软件分析,根据菌株的条带位置及数量差异,当菌株间相似度系数为100%,判定为同一PFGE型别。结果2株肯塔基沙门菌经Xba Ⅰ与Bln Ⅰ酶切后的单酶切PFGE指纹图聚类分析、双酶切指纹图组合聚类分析(composite cluster analysis)[18],相似度均为100%,见图 1c,证明为2株肯塔基沙门菌为完全相同的PFGE指纹型。
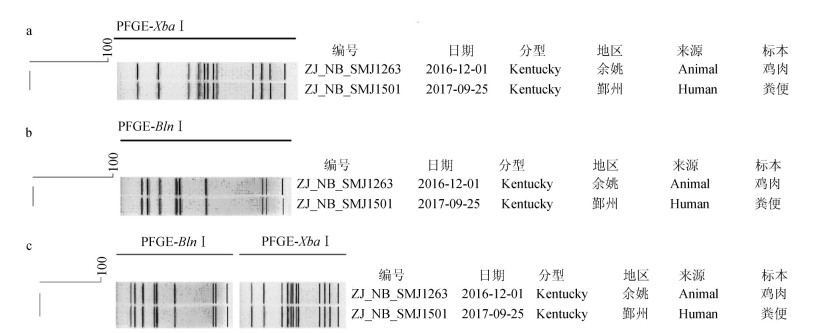
|
| 图 3 2株肯塔基沙门菌的脉冲场凝胶电泳分型聚类结果 Figure 3 Cluster analysis of 2 S. Kentucky isolates |
| |
2株肯塔基沙门菌对12种抗生素的耐药谱完全相同,见表 1。2株肯塔基沙门菌均为多重耐药菌株。其中对环丙沙星的MIC均为16 μg/ml。2株肯塔基沙门菌ESBLs表型均为阳性。
| 抗生素名称 | 药物敏感性 |
| 氨苄西林 | R |
| 头孢唑啉 | R |
| 头孢呋辛 | R |
| 头孢噻肟 | R |
| 头孢吡肟 | R |
| 氨曲南 | R |
| 复方新诺明 | S |
| 氯霉素 | S |
| 庆大霉素 | R |
| 亚胺培南 | S |
| 环丙沙星 | R |
| 左氧氟沙星 | R |
| 注:S.敏感;R:耐药 | |
本研究中肯塔基沙门菌的ESBLs耐药基因blaCTX-M14-like、blaCTX-M15-like、blaTEM检测结果均阳性,blaOXA基因检测阴性。gyrA基因的序列分析结果显示,其第83位密码子TCC(Ser)突变为TTC(Phe),第87位密码子GAC(Asp)点突变为GGC(Gly)。
3 讨论随着现代禽类养殖业的发展,抗生素的滥用情况愈发严重。由于喹诺酮类以及超广谱头孢菌素类抗生素是治疗沙门菌感染的首选抗生素,因此不但用在人类自身,而且在禽类养殖业内也被广泛使用甚至滥用。养殖鸡类身上携带的细菌由于选择压力,更加容易变异成耐药菌或者多重耐药菌。人类食入未充分煮熟或者被污染的鸡肉,易感染耐药沙门菌。
目前禽类相关的NTS的耐药十分严重。市售鸡肉中常携带大量的多重耐药沙门菌[19],使得临床治疗药物的选择越来越困难。文献报道我国市售鸡肉中检出最多的沙门菌为印第安纳血清型,该血清型也是我国NTS中多重耐药最严重者[20],对印第安纳沙门菌的监测已经引起了全国各地区的高度重视。
“超级耐药”肯塔基沙门菌是欧美地区重点监测的禽源沙门菌,但在我国未见文献报道、未受到充分重视。本研究中宁波市分离到的2株“超级耐药”肯塔基沙门菌,PFGE分型显示,来自于鸡肉标本的肯塔基沙门菌分离株指纹图与分离自患者的分离株指纹图完全一致。2株肯塔基沙门菌的抗生素敏感性测试结果也完全一致,均为环丙沙星高水平耐药,MIC值=16 μg/ml,属于国际上关注的“超级耐药”肯塔基沙门菌。2株不同来源的肯塔基沙门菌,PFGE指纹图与耐药谱完全一致,说明该型“超级耐药”肯塔基沙门菌不仅存在于养殖鸡身上,而且可引起住院患者的严重感染。由于我国各级细菌实验室在沙门菌血清型的鉴定能力受到血清质量、检验人员技术能力等相关因素影响,加上沙门菌监测网点不足,食品或腹泻患者中是否存在更多的“超级耐药”肯塔基沙门菌的污染或感染,需要有更多的监测数据支持。宁波地区检出高耐药肯塔基沙门菌,也说明鸡肉中多重耐药沙门菌血清型在一定程度上呈多样化趋势[21],不应局限于我国已知的多重耐药沙门菌血清型的监测,应不断更新我国监测网络发现的多重耐药沙门菌血清谱。
本研究发现宁波市2株肯塔基沙门菌不但对奎诺酮类中的环丙沙星、左氧氟沙星呈现高水平耐药,而且对Ⅲ代头孢、Ⅳ代头孢(头孢吡肟)也出现了耐药现象,耐药情况比该菌在欧洲国家报道的情形更为严重。这种超级耐药菌对于临床治疗抗生素的选择提出了更大的挑战,使治疗更为困难。因此应加强对这种高水平、多重耐药菌的监控。针对目前多重耐药菌不断产生的严峻形势,应切实加强对养殖业抗生素的使用管理,呼吁动物养殖业自主退出头孢菌素类的使用,以免造成临床治疗无药可用的状态。
耐药基因的检测结果显示,2株肯塔基沙门菌blaCTX-M14-like、blaCTX-M15-like、blaTEM等ESBLs决定基因阳性。1株细菌同时产生上述2型(CTX-M and TEM)β-内酰胺酶,报道比较少见[22]。其中blaCTX-M14-like、bla CTX-M15-like基因阳性,与文献报道关于CTX-M14和CTX-M15酶是最流行的2型ESBLs相符[23]。有研究认为欧美国家以CTX-M15型酶最为流行,而在中国等亚洲国家则以CTX-M14型酶最为流行[24]。上述2种ESBLs酶同时在本研究中的2株肯塔基沙门菌中检出,需引起重视。对于喹诺酮类抗生素耐药的分子机制,本研究测序结果显示细菌的DNA旋转酶A亚基(GyrA)的83位氨基酸由丝氨酸突变为苯丙氨酸,与相关报道一致[25];不同之处在于GyrA的87位氨基酸由天冬酰胺突变为甘氨酸,产生的新氨基酸导致GyrA构象变化,从而使喹诺酮类药物失去作用靶点。上述耐药基因的检测结果,从基因水平反映了本研究中肯塔基沙门菌的耐药机制。
从2017年开始,“国家致病菌识别网”已经在全国全面开展工作,对于耐药菌的识别与监控也已纳入常规的网络监测任务。今后应在工作中不断加强多重耐药沙门菌的监测工作,实现信息共享,实时比对各地监测结果,对相似度高的菌株进行流行病学溯源,以防范多重耐药沙门菌在禽类养殖业以及人群中的传播。
志谢: 本研究中来自住院腹泻患者的肯塔基沙门菌的分离及鉴定工作,由宁波市中医院细菌室夏晴晴主任技师完成,在此表示衷心的感谢!作者贡献:
杨元斌 ORCID:0000-0002-9666-1123
杨元斌:论文撰写,细菌血清学分型,耐药分析,耐药基因检测,脉冲场凝胶电泳的全部操作、聚类分析、结果分析
高红:细菌的生化、血清学鉴定;脉冲场凝胶电泳的部分操作
章丹阳:细菌的生化、血清学鉴定,脉冲场凝胶电泳的部分操作,菌株收集,结果分析
闫鹏:细菌的耐药分析,菌株收集,结果分析
沈玄艺:脉冲场凝胶电泳的部分操作,菌株收集,结果分析
宋启发:耐药基因的检测,gyrA基因的测序分析
| [1] |
Majowicz SE, Musto J, Scallan E, et al. The global burden of nontyphoidal Salmonella Gastroenteritis[J]. Clin Infect Dis, 2010, 50(6): 882-889. DOI:10.1086/650733 |
| [2] |
毛雪丹, 胡俊峰, 刘秀梅. 2003-2007年中国1060起细菌性食源性疾病流行病学特征分析[J]. 中国食品卫生杂志, 2010, 22(3): 224-228. Mao XD, Hu JF, Liu XM. Epidemiological characteristics of bacterial foodborne disease during the year 2003-2007 in China[J]. Chin J Food Hyg, 2010, 22(3): 224-228. DOI:10.13590/j.cjfh.2010.03.017 |
| [3] |
李剑森, 梁骏华, 柯碧霞, 等. 2012年广东省食源性疾病监测结果分析[J]. 华南预防医学, 2013, 39(6): 10-16. Li JS, Liang JH, Ke BX, et al. Surveillance of foodborne disease in Guangdong, 2012[J]. South Chin J Prev Med, 2013, 39(6): 10-16. DOI:10.13217/j.scjpm.2013.06.010 |
| [4] |
王军, 郑增忍, 王晶钰. 动物源性食品中沙门菌的风险评估[J]. 中国动物检疫, 2007, 24(4): 23-25. Wang J, Zheng ZR, Wang JY. Risk assessment of Salmonella in animal derived food[J]. Chin J Anim Quar, 2007, 24(4): 23-25. DOI:10.3969/j.issn.1005-944X.2007.04.012 |
| [5] |
Wang Y, Chen Q, Cui SH, et al. Enumeration and characterization of Salmonella isolates from retail chicken carcasses in Beijing, China[J]. Foodborne Pathog Dis, 2014, 11(2): 126-132. DOI:10.1089/fpd.2013.1586 |
| [6] |
Wong MHY, Zeng L, Liu JH, et al. Characterization of Salmonella food isolates with concurrent resistance to ceftriaxone and ciprofloxacin[J]. Foodborne Pathog Dis, 2013, 10(1): 42-46. DOI:10.1089/fpd.2012.1266 |
| [7] |
Edwards PR. A new Salmonella type:Salmonella Kentucky[J]. J Hyg (Lond), 1938, 38(3): 306-308. DOI:10.1017/S0022172400011177 |
| [8] |
Le Hello S, Hendriksen RS, Doublet B, et al. International spread of an epidemic population of Salmonella enterica serotype Kentucky ST198 resistant to ciprofloxacin[J]. J Infect Dis, 2011, 204(5): 675-684. DOI:10.1093/infdis/jir409 |
| [9] |
Mulvey MR, Boyd DA, Finley R, et al. Ciprofloxacin-resistant Salmonella enterica Serovar Kentucky in Canada[J]. Emerg Infect Dis, 2013, 19(6): 999-1001. DOI:10.3201/eid1906.121351 |
| [10] |
Rickert-Hartman R, Folster JP. Ciprofloxacin-resistant Salmonella enterica serotype Kentucky sequence type 198[J]. Emerg Infect Dis, 2014, 20(5): 910-911. DOI:10.3201/eid2005.131575 |
| [11] |
Weill FX, Bertrand S, Guesnier F, et al. Ciprofloxacin-resistant Salmonella Kentucky in travelers[J]. Emerg Infect Dis, 2006, 12(10): 1611-1612. DOI:10.3201/eid1210.060589 |
| [12] |
WHOCC-Salm. White-Kauffmann-le-minor scheme[M]//Grimont PAD, Weill FX. Antigenic Formulae of the Salmonella Serovars. 9th ed. Paris: WHO Collaborating Centre for Reference and Research on Salmonella, Institut Pasteur, 2007: 15-107.
|
| [13] |
Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing: seventh informational supplement[S]. Wayne, PA, USA: CLSI, 2017.
|
| [14] |
European Committee on Antimicrobial Susceptibility Testing. Breakpoint tables for interpretation of MICs and zone diameters. V7. 1[EB/OL]. (2017-03-10)[2017-12-10]. http://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_7.1_Breakpoint_Tables.pdf.
|
| [15] |
Ribot EM, Fair MA, Gautom R, et al. Standardization of pulsed field gel electrophoresis protocols for the subtyping of Escherichia coli O157:H7, Salmonella, and Shigella for PulseNet[J]. Foodborne Pathog Dis, 2006, 3(1): 59-67. DOI:10.1089/fpd.2006.3.59 |
| [16] |
Hunter SB, Vauterin P, Lambert-Fair MA, et al. Establishment of a universal size standard strain for use with the PulseNet standardized pulsed field gel electrophoresis protocols:converting the national databases to the new size standard[J]. J Clin Microbiol, 2005, 43(3): 1045-1050. DOI:10.1128/JCM.43.3.1045-1050.2005 |
| [17] |
Tenover FC, Arbeit RD, Goering RV, et al. Interpreting chromosomal DNA restriction patterns produced by pulsed field gel electrophoresis:criteria for bacterial strain typing[J]. J Clin Microbiol, 1995, 33(9): 2233-2239. |
| [18] |
Zheng J, Keys CE, Zhao SH, et al. Simultaneous analysis of multiple enzymes increases accuracy of pulsed field gel electrophoresis in assigning genetic relationships among homogeneous Salmonella strains[J]. J Clin Microbiol, 2011, 49(1): 85-94. DOI:10.1128/JCM.00120-10 |
| [19] |
Wang Y, Chen Q, Cui SH, et al. Enumeration and characterization of Salmonella isolates from retail chicken carcasses in Beijing, China[J]. Foodborne Pathog Dis, 2014, 11(2): 126-132. DOI:10.1089/fpd.2013.1586 |
| [20] |
胡豫杰, 赫英英, 王晔茹, 等. 中国六省份零售整鸡中环丙沙星与头孢噻肟双耐药印第安纳沙门菌流行状况及分子分型研究[J]. 中国预防医学, 2015, 49(8): 716-721. Hu YJ, He YY, Wang YR, et al. Epidemic condition and molecular subtyping of ciprofloxacin and cefotaxime co-resistant Salmonella Indiana isolated from retail chicken carcasses in six provinces, China[J]. Chin J Prev Med, 2015, 49(8): 716-721. DOI:10.3760/cma.j.issn.0253-9624.2015.08.010 |
| [21] |
Kasimoglu DA, Ayaz ND, Gencay YE. Serotype identification and antimicrobial resistance profiles of Salmonella spp. isolated from chicken carcasses[J]. Trop Anim Health Prod, 2010, 42(5): 893-897. DOI:10.1007/s11250-009-9504-7 |
| [22] |
Ahmed D, Hoque A, Mazumder R, et al. Salmonella enterica serovar typhi strain producing extended-spectrum β-lactamases in Dhaka, Bangladesh[J]. J Med Microbiol, 2012, 61(7): 1032-1033. DOI:10.1099/jmm.0.044065-0 |
| [23] |
Naseer U, Sundsfjord A. The CTX-M conundrum:dissemination of plasmids and Escherichia coli clones[J]. Micmb Drug Resist, 2011, 17(1): 83-97. DOI:10.1089/mdr.2010.0132 |
| [24] |
Shin J, Kim DH, Ko KS. Comparison of CTX-M-14-and CTX-M-15-producing Escherichia coli and Klebsiella pneumoniae isolates from patients with bacteremia[J]. J Infect, 2011, 63(1): 39-47. DOI:10.1016/j.jinf.2011.05.003 |
| [25] |
Fábio C, Antonio SR, Vicente MV, et al. Prevalence of gyrA mutations in nalidixic acid-resistant strains of Salmonella Enteritidis isolated from humans, food, chickens, and the farm environment in Brazil[J]. Microb Drug Resist, 2017, 23(4): 421-428. DOI:10.1089/mdr.2016.0024 |
 2018, Vol. 33
2018, Vol. 33


