扩展功能
文章信息
- 牛桓彩, 吴杨, 舒高林, 刘毅, 马文军, 李东迅
- Niu Huancai, Wu Yang, Shu Gaolin, Liu Yi, Ma Wenjun, Li Dongxun
- 北京市昌平区食源性单增李斯特菌特征分析
- Characteristics of Listeria monocytogenes isolated from foods in Changping district of Beijing
- 疾病监测, 2018, 33(6): 463-468
- Disease Surveillance, 2018, 33(6): 463-468
- 10.3784/j.issn.1003-9961.2018.06.006
-
文章历史
- 收稿日期:2018-04-25
单增李斯特菌(Listeria monocytogenes,Lm)为革兰阳性无芽孢短杆菌,对营养要求不高,可在宽泛的温度(2~42 ℃)和pH范围(pH值4~9)生长,是人畜共患的食源性病原菌[1]。Lm感染可导致孕妇流产或新生儿死亡,感染新生儿、儿童、老年人及免疫力低下人群后病死率较高,世界卫生组织(WHO)将其列为四大重要的食源性病原菌之一[2]。为了解北京市昌平区食源性Lm毒力基因、抗生素敏感性及分子特征,为Lm病预防及治疗提供参考,本研究对昌平区12类食品中分离到的22株Lm进行了血清分型、毒力基因、药物敏感性检测、多位点序列分型(MLST)和脉冲场凝胶电泳(PFGE)分型。
1 材料与方法 1.1 材料 1.1.1 样品来源按照分层整群随机抽样方法,在昌平区城镇、城乡结合部和农村地区采集的12类(生禽肉、生畜肉、鲜冻水产品、生食水产品、熟肉制品、豆制品、焙烤食品、沙拉、冷冻饮品、生食蔬菜、中式凉拌菜、速冻米面制品)478份食品样品。经过增菌、分离、生化鉴定,在生禽生畜肉(19株)、鲜冻水产品(2株)和速冻米面制品(1株)中共分离到Lm 22株,分别来自农贸市场9株(9/172),超市/便利店8株(8/194),餐饮单位5株(5/112)。
1.1.2 仪器与试剂LB增菌液、PALCAM和脑心琼脂培养基(北京陆桥公司);Lm显色培养基(郑州博赛公司产品)、血平板和革兰阳性细菌鉴定卡(法国生物梅里埃公司)、Seakem Gold琼脂糖(美国Cambrex Bio Science Rockland)、蛋白酶K(德国Merck)、限制性内切酶Xba Ⅰ和Asc Ⅰ(美国New England Biolabs)、2×T5 Super PCR Mix和PCR引物合成(北京擎科新业生物技术有限公司)、革兰阳性菌(单增)药敏检测板(上海星佰生物技术有限公司)、李斯特菌分型血清抗体(日本DenkaSeiken公司)、药敏质控菌株肺炎链球菌ATCC49619由中国疾病预防控制中心(CDC)慧赠;生物安全柜(美国Baker)、恒温培养箱(日本三洋)、McFarland标准比浊仪(法国生物梅里埃公司),SW22水浴摇床(德国Julabo)、VITEK2细菌鉴定仪(法国生物梅里埃公司);PCR仪、电泳仪、PFGE仪及配套设备和凝胶成像系统(美国Bio⁃Rad公司)。
1.2 方法 1.2.1 菌株分离依据GB 4789.30-2010 [3]进行菌株分离及鉴定。
1.2.2 血清分型检测采用日本DenkaSeiken公司生产的Lm分型血清,严格按照血清抗体产品说明书对菌株进行血清分型。
1.2.3 基因组DNA提取采用水煮法,取脑心琼脂上培养的Lm单菌落于1.5 ml离心管中,加入500 μl 1 × TE缓冲液旋涡振荡混匀,放入100 ℃水浴煮10 min,8 000 ×g离心5 min,取200 μl上清液(DNA)于-20 ℃保存备用。
1.2.4 毒力基因检测采用PCR法对12个毒力基因(plcA、mpl、actA、plcB、inlA、inlB、inlC、inlJ、hlyA、prfA、iap、flaA)进行检测,引物[4-6]及退火温度见表 1,采用2×T5 Super PCR Mix对上述毒力基因进行PCR扩增,按说明书配制PCR扩增体系并进行扩增反应。
| 基因 | 引物 | 引物序列(5′~3′) | 产物大小(bp) | 退火温度(℃) |
| actA | actA-F | AAA CAG AAG AGC AGC CAA GC | 571 | 53 |
| actA-R | TTC ACT TCG GGA TTT TCG TC | |||
| hlyA | hlyA-F | GCA GTT GCA AGC GCT TGG AGT GAA | 456 | 61 |
| hlyA-R | GCA ACG TAT CCT CCA GAG TGA TCG | |||
| prfA | prfA-F | CTG TTG GAG CTC TTC TTG GTG AAG CAA TCG | 1 060 | 59 |
| prfA-R | AGC AAC CTC GGT ACC ATA TAC TAA CTC | |||
| flaA | flaA-F | AGC TCT TAG CTC CAT GAG TT | 450 | 53 |
| flaA-R | ACA TTG TAG CTA AGG CGA CT | |||
| plcA | plcA-F | TCC CAT TAG GTG GAA AAG CA | 840 | 53 |
| plcA-R | CGG GGA AGT CCA TGA TTA GA | |||
| plcB | plcB-F | CAG CTC CGC ATG ATA TTG AC | 723 | 53 |
| plcB-R | CTG CCA AAG TTT GCT GTG AA | |||
| inlA | inl A-F | GGC TGG GCA TAA CCA AAT TA | 629 | 53 |
| inl A-R | CTT TTG TTG GTG CCG TAG GT | |||
| inlB | inl B-F | CCT AAA CCT CCG ACC AAA CA | 293 | 55 |
| inl B-R | CCA TTT CGC GCT TCT CTA TC | |||
| inlC | inlC-F | AAT TCC CAC AGG ACA CAA CC | 517 | 51 |
| inlC-R | CGG GAA TGC AAT TTT TCA CTA | |||
| inlJ | inlJ-F | TGT AAC CCC GCT TAC ACA GTT | 238 | 55 |
| inlJ-R | AGC GGC TTG GCA GTC TAA TA | |||
| iap | iap-F | ACA AGC TGC ACC TGT TGC AG | 131 | 55 |
| iap-R | TGA CAG CGT GTG TAG TAG CA | |||
| mpl | mpl-F | AAA GGT GGA GAA ATT GAT TCG | 450 | 51 |
| mpl-R | AGT GAT CGT ATT GTA GGC TGC TT |
参照美国临床和实验室标准化协会(CLSI)推荐的微量肉汤稀释法进行药物敏感性试验。将菌株复苏,于脑心琼脂培养基上传代2次后,挑取数个单克隆菌落悬浮于5 ml 0.85%盐水中,调制菌液浓度为1.5×108 cfu/ml,取60 μl菌液加入到12 ml(预加入600 μl无菌马血清)营养肉汤中完全混匀,吸取100 μl稀释菌液加入96孔微量药敏板中,35 ℃培养20~24 h,进行结果判读(混浊为阳性,清晰为阴性)。抗生素种类及浓度见表 2。
| 抗生素 | 抗生素浓度梯度(μg/ml) | ||||||||||
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | |
| 氨苄西林 | 32 | 16 | 8 | 4 | 2 | 1 | 0.5 | 0.25 | 0.125 | 0.06 | 0.03 |
| 青霉素 | 32 | 16 | 8 | 4 | 2 | 1 | 0.5 | 0.25 | 0.125 | 0.06 | 0.03 |
| 四环素 | 32 | 16 | 8 | 4 | 2 | 1 | 0.5 | 0.25 | 0.125 | 0.0625 | 0.03 |
| 美罗培南 | 8 | 4 | 2 | 1 | 0.5 | 0.25 | 0.125 | 0.06 | 0.03 | 0.015 | 0.0075 |
| 复方新诺明 | 8/152 | 4/74 | 2/38 | 1/9 | 0.5/9.5 | 0.25/4.8 | 0.12/2.4 | 0.06/1.2 | 0.03/06 | 0.015/0.3 | 0.007/0.15 |
| 红霉素 | 32 | 16 | 8 | 4 | 2 | 1 | 0.5 | 0.25 | 0.125 | 0.06 | 0.03 |
| 万古霉素 | 32 | 16 | 8 | 4 | 2 | 1 | 0.5 | 0.25 | 0.125 | 0.0625 | 0.03125 |
| 环丙沙星 | 32 | 16 | 8 | 4 | 2 | 1 | 0.5 | 0.25 | 0.125 | 0.0625 | 0.03125 |
参照http://bigsdb.pasteur.fr/listeria/primers_used.html提供的方法,对Lm的7个管家基因(abcZ、bglA、cat、dapE、dat、ldh、lhkA)进行PCR扩增。PCR扩增体系配制和扩增条件详见2× T5 Super PCR Mix说明书,扩增产物送北京擎科新业生物技术有限公司进行测序,用SeqmanⅡ软件对等位基因序列进行拼接和校正,校正后的序列提交到L. monocytogenes MLST数据库(http://bigsdb.pasteur.fr/perl/bigsdb/bigsdb.pl?db=pubmlst_listeria_seqdef_public)获取等位基因型,7个等位基因的组合即为该菌的序列型(ST)。将7个管家基因序列拼接后,用MEGA 6.0软件进行非加权类平均法(unweighted pair group method using arithmetic averages,UPGMA)分析,构建系统发育树,分析菌株之间的亲缘关系。
1.2.7 PFGE分型按照美国CDC PulseNet(https://www.cdc.gov/pulsenet/pathogens/pfge.html)制定的Lm的PFGE标准,Lm用限制性内切酶AscⅠ进行酶切,沙门菌标准株H9812作为相对分子质量标记,用限制性内切酶XbaⅠ进行酶切。PFGE图像用BioNumerics(Version 5.0)软件根据UPGMA方法进行聚类分析。
2 结果 2.1 血清分型22株Lm分为5种血清型:1/2c(10/ 22,45.45%)、3a(7/22,31.82%)、1/2a(2/22,9.10%)、3b(2/22,9.10%)、1/2b(1/22,4.55%),其中1/2c和3a为优势血清型。
2.2 毒力基因检测12个毒力基因在22株Lm分离株中均有不同程度的携带。inlJ、hlyA、iap和flaA在22株菌中均为阳性,其他毒力基因的携带率分别为inlC(20/22,90.91%)、inlA(19/22,86.36%)、inlB(16/22,72.73%)、prfA(16/22,72.73%)、plcB(15/22,68.18%)、plcA(11/22,50.00%)、mpl(9/22,40.91%)、actA(9/22,40.91%)。Lm分离菌株分别携带6~12个毒力基因,分为19种毒力基因携带谱(T1~T19),包括3株T9型(13.64%),2株T2型(9.09%),其他型别各1株(占4.55%)(图 1、2)。
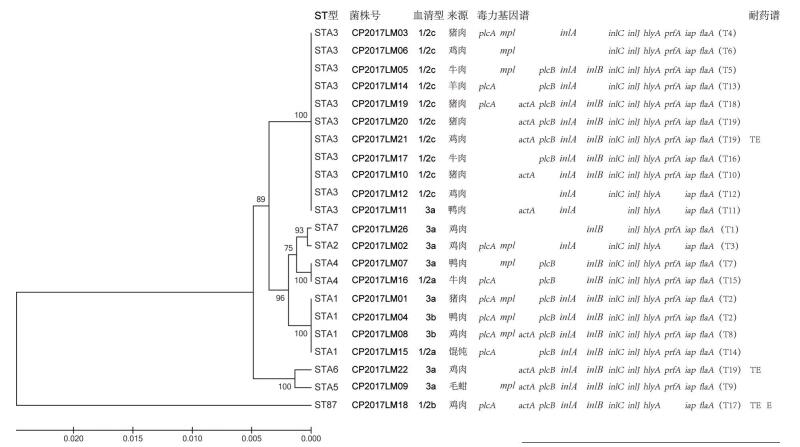
|
| 图 1 22株单增李斯特菌7个管家基因的系统发育树 Figure 1 Phylogenetic tree based on concatenated sequences of 7 housekeeping genes of all 22 L. monocytogenes isolates 注:TE为四环素;E为红霉素 |
| |
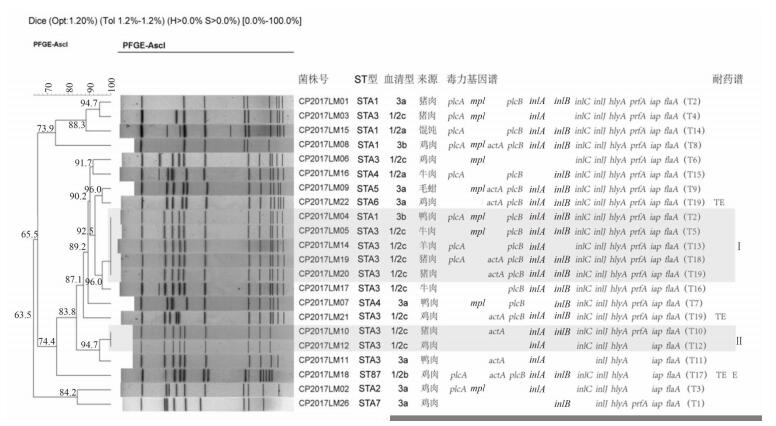
|
| 图 2 22株单增李斯特菌的脉冲场凝胶电泳聚类图、序列型别、毒力基因谱及耐药谱 Figure 2 Dendrogram of PFGE patterns, ST types, virulence genes profiles and antibiotics spectrum of all 22 L. monocytogenes strains 注:TE为四环素;E为红霉素 |
| |
对22株Lm菌进行了8种抗生素药敏试验,参照CLSI 2017年颁布的标准进行结果判定。结果显示全部Lm分离株对6种抗生素(氨苄西林、青霉素、美罗培南、复方新诺明、万古霉素和环丙沙星)均敏感,3株生鸡肉分离株对部分抗生素耐药(3/22,13.64%),其中2株对四环素耐药,1株对四环素和红霉素双重耐药(图 1、2)。
2.4 MLST分型22株Lm分离株分为8个ST:ST87(1株)和7个新ST(STA1~STA7,21株),见表 3。用UPGMA方法构建的系统发育树结果显示(图 1),在优势血清型(1/2c,3a)菌株中,1/2c血清型菌株均为STA3型,3a则散在分布。相同ST型菌株的血清型有所不同,如STA3包括1/2c(10)和3a(1),STA1包括3b(2)、3a(1)和1/2a(1),STA4包括3a(1)、1/2a(1)。相同毒力基因谱的菌株ST型别相同或不同,如2株毒力基因谱型T2菌株均为STA1型,而2株STA3和1株STA6菌株均为T19。耐药性方面,STA3、STA6各有1株菌对四环素耐药,1株ST87对四环素和红霉素同时耐药。
| 序列型别 | 管家基因 | 菌株数 | ||||||
| abcZ | bglA | cat | dapE | dat | ldh | lhkA | ||
| STA1 | 7 | 6 | 8 | 8 | 6 | 37 | 62 | 4 |
| STA2 | 7 | 10 | 8 | 7 | 5 | 2 | 62 | 1 |
| STA3 | 6 | 5 | 6 | 4 | 1 | 4 | 62 | 11 |
| STA4 | 5 | 6 | 2 | 9 | 5 | 3 | 62 | 2 |
| STA5 | 7 | 49 | 19 | 6 | 1 | 24 | 62 | 1 |
| STA6 | 6 | 49 | 19 | 6 | 1 | 24 | 62 | 1 |
| STA7 | 7 | 10 | 16 | 7 | 5 | 2 | 62 | 1 |
22株Lm共分为17种PFGE带型(PT),菌株间相似系数为63.5%~100.0%,其中带型Ⅰ(PTⅠ)含5株菌,为主要PT型;结合PT型和ST型分析发现,相同PT型的大部分菌株具有相同ST型,但其毒力基因谱型不同,如图 2中Ⅰ和Ⅱ;相同ST型菌株PT型相同或散在分布;相同毒力基因谱型(T2和T19)和耐药谱相同菌株的PT型有所不同(图 2)。
3 讨论Lm是一种重要的食源性病原体,可引起Lm病,主要通过污染的食品感染人类[1-2]。目前Lm被分为13种血清型,1/2c血清型菌株常分离自食品标本,而1/2a、1/2b和4b血清型菌株常分离自患者标本[1, 7-8]。本研究分离的22株Lm中19株(86.36%)分离自生肉类(鸡肉7株、猪肉5株、鸭肉和牛肉各2株,羊肉1株),以1/2c血清型为主,与马爱静等[2]报道北京地区生肉样本中1/2c为优势血清型(占47.60%)一致。
Lm致病过程中,内化素起重要作用,它作为Lm的表面黏附因子与组织细胞的表面受体结合,使Lm黏附在细胞表面,从而完成入侵[9]。inlA、inlB、inlC和inlJ是主要的内化素基因,鱼或鱼市环境样品中分离的Lm均携带inlA、inlB、inlC基因,且inlJ阳性率高达97.70%[10]。本研究中生肉样品分离的Lm均携带inlJ基因,inlA、inlB、inlC阳性率分别为86.36%(19/22)、72.73%(16/22)、90.91%(20/22)。伊朗、印度的早期研究显示[11-12],海产品中分离的Lm均携带actA、hlyA、iap、plcA和prfA基因,本研究中Lm均携带hlyA、iap基因,actA、plcA和prfA阳性率分别为40.91%(9/22)、50.00%(11/22)、72.73%(16/22),提示不同来源的菌株毒力基因携带情况可能不同。
Lm通常对大部分抗生素敏感,但也有多重耐药菌株出现[5]。多重耐药菌株已在临床病例样本及食品、水和环境样品中出现[13-14]。药敏结果显示,本研究中的Lm对大部分抗生素敏感,出现对四环素、红霉素耐药的菌株,其中对四环素耐药率为13.64%,高于王天姝等[15]的报道,3株耐药株均分离自生鸡肉,可能与我国畜牧业饲料中添加抗生素有关。根据《抗菌药物临床应用指导原则》,红霉素是治疗Lm病的备选药物,其耐药株的出现应予以重视。
研究发现,我国临床分离Lm菌株和食源性Lm菌株具有相同的ST,但其优势型别有所不同,如ST3、ST7和ST87型菌株在临床Lm病例样本中多见,ST9和ST8型为食源性菌株优势菌型[7]。本研究中Lm菌株出现7个新ST(STA1~STA7),占总菌数的95.45%(21/22),STA3为主要ST型别(11/22),且优势血清型1/2c菌株均属于STA3。22株Lm经PFGE分型分析,分为17种PT型,提示昌平区食源性Lm呈现高度多态性;相同PT的大部分菌株ST相同,但毒力基因谱不同;相同ST菌株PT相同或散在分布。
本次研究发现,昌平区分离的食源性Lm具有遗传多样性,优势血清型为1/2c、3a,STA3为主要ST型别,PTⅠ为主要PFGE带型,毒力基因的携带具有差异,出现对四环素、红霉素耐药的菌株,为本地区的食源性Lm感染的预防控制提供了参考。
作者贡献:
牛桓彩 ORCID:0000-0002-9640-9489
牛桓彩:菌株分离,分型、毒力基因及药敏检测,文章撰写
吴杨:分型、毒力基因及药敏检测
舒高林:菌株分离
刘毅、马文军:菌株分离
李东迅:分型、毒力基因及药敏检测,文章修改
| [1] |
李东迅, 王艳, 马爱静, 等. 单增李斯特菌1/2c血清型菌株生物膜形成能力的研究[J]. 疾病监测, 2015, 30(6): 474-478. Li DX, Wang Y, Ma AJ, et al. Study on biofilm formation of foodborne Listeria monocytogenes serotype 1/2c strains[J]. Dis Surveill, 2015, 30(6): 474-478. DOI:10.3784/j.issn.1003-9961.2015.06.011.issn.1003-9961.2015.06.011 |
| [2] |
马爱静, 王艳, 王毅, 等. 北京市一些地区生肉标本中单增李斯特菌的分离及其分子流行病学特征分析[J]. 中国人兽共患病学报, 2015, 31(5): 403-407. Ma AJ, Wang Y, Wang Y, et al. Molecular epidemiological characteristics of Listeria monocytogenes isolated from raw meat samples in some regions of Beijing, China[J]. Chin J Zoon, 2015, 31(5): 403-407. DOI:10.3969/cjz.j.issn.1002-2694.2015.05.003 |
| [3] |
中华人民共和国卫生部. GB 4789. 30-2010食品安全国家标准食品微生物学检验单核细胞增生李斯特氏菌检验[S]. 北京: 中国标准出版社, 2010. Ministry of Health, PRC. GB 4789. 30-2010 National food safety standard Food microbiological examination: Listeria monocytogenes[S]. Beijing: Standards Press of China, 2010. |
| [4] |
Montero D, Bodero M, Riveros G, et al. Molecular epidemiology and genetic diversity of Listeria monocytogenes isolates from a wide variety of ready-to-eat foods and their relationship to clinical strains from listeriosis outbreaks in Chile[J]. Front Microbiol, 2015, 6: 384. DOI:10.3389/fmicb.2015.00384 |
| [5] |
Soni DK, Singh M, Singh DV, et al. Virulence and genotypic characterization of Listeria monocytogenes isolated from vegetable and soil samples[J]. BMC Microbiol, 2014, 14: 241. DOI:10.1186/s12866-014-0241-3 |
| [6] |
Indrawattana N, Nibaddhasobon T, Sookrung N, et al. Prevalence of Listeria monocytogenes in raw meats marketed in Bangkok and characterization of the isolates by phenotypic and molecular methods[J]. J Health Popul Nutr, 2011, 29(1): 26-38. |
| [7] |
Wang Y, Jiao Y, Lan RT, et al. Characterization of Listeria monocytogenes isolated from human Listeriosis cases in China[J]. Emerg Microbes Infect, 2015, 4(8): e50. DOI:10.1038/emi.2015.50 |
| [8] |
王毅, 王艳, 叶正兴, 等. 鉴定单增李斯特菌四个进化家系及两个亚系的多PCR方法的建立[J]. 中国人兽共患病学报, 2013, 29(12): 1151-1156. Wang Y, Wang Y, Ye ZX, et al. Differentiation of 4 Listeria monocytogene phylogenetic lineages and 2 sublineages by a novel multiplex PCR[J]. Chin J Zoonoses, 2013, 29(12): 1151-1156. DOI:10.3969/cjz.j.issn.1002-2694.2013.12.005 |
| [9] |
McGann P, Raengpradub S, Ivanek R, et al. Differential regulation of Listeria monocytogenes internalin and internalin-like genes by σB and PrfA as revealed by subgenomic microarray analyses[J]. Foodborne Pathog Dis, 2008, 5(4): 417-435. DOI:10.1089/fpd.2008.0085 |
| [10] |
Jamali H, Paydar M, Ismail S, et al. Prevalence, antimicrobial susceptibility and virulotyping of Listeria species and Listeria monocytogenes isolated from open-air fish markets[J]. BMC Microbiol, 2015, 15: 144. DOI:10.1186/s12866-015-0476-7 |
| [11] |
Das S, Lalitha KV, Thampuran N, et al. Isolation and characterization of Listeria monocytogenes from tropical seafood of Kerala, India[J]. Ann Microbiol, 2013, 63(3): 1093-1098. DOI:10.1007/s13213-012-0566-9 |
| [12] |
Momtaz H, Yadollahi S. Molecular characterization of Listeria monocytogenes isolated from fresh seafood samples in Iran[J]. Diagn Pathol, 2013, 8: 149. DOI:10.1186/1746-1596-8-149 |
| [13] |
Sharma D, Sharma PK, Saharan BS, et al. Isolation, identification and antibiotic susceptibility profiling of antimicrobial resistant Listeria monocytogenes from dairy milk[J]. Int J Microb Resour Technol, 2012, 1(1): 1-4. |
| [14] |
Oni DK, Singh RK, Singh DV, et al. Characterization of Listeria monocytogenes isolated from Ganges water, human clinical and milk samples at Varanasi, India[J]. Infect Genet Evol, 2013, 14: 83-91. DOI:10.1016/j.meegid.2012.09.019 |
| [15] |
王天姝, 王艳, 贺春月, 等. 中国部分食品分离单增李斯特菌的抗菌药物敏感性及耐药基因检测[J]. 疾病监测, 2013, 28(3): 224-229. Wang TS, Wang Y, He CY, et al. Detection of drug susceptibility and resistant genes in selected food borne Listeria monocytogenes in China[J]. Disease Surveill, 2013, 28(3): 224-229. DOI:10.3784/j.issn.1003-9961.2013.3.016 |
 2018, Vol. 33
2018, Vol. 33


